| |
| Med Sci (Paris). 2003 January; 19(1): 55–62. Published online 2003 January 15. doi: 10.1051/medsci/200319155.Réparation des erreurs de réplication, microsatellites et cancer Alex Duval* and Richard Hamelin Inserm U.434, CEPH, 27, rue Juliette Dodu, 75010 Paris, France |
Il est maintenant admis, aussi bien chez les procaryotes que chez les eucaryotes, que la préservation de l’intégrité du génome n’est possible que grâce à l’existence de systèmes multiples intervenant dans des processus complexes de réplication, de réparation et de recombinaison de la molécule d’ADN. Le système de réparation des erreurs de réplication de l’ADN (système MMR, mismatch repair), capital pour la cellule, en est une illustration. Les mésappariements de l’ADN susceptibles d’être réparés par le système MMR sont, dans la grande majorité des cas, la conséquence d’erreurs commises par l’ADN polymérase au cours du processus de réplication [1]. En effet, il a été constaté in vitro que cette enzyme faisait parfois des erreurs en introduisant sur sa copie des nucléotides erronés, particulièrement lors de la réplication de séquences répétées [2]. L’existence d’un processus MMR post-réplicatif permet d’augmenter la fidélité de la réplication de 2 à 3 unités logarithmiques [3]. D’autres systèmes tout aussi fondamentaux, mais dont nous ne parlerons pas ici, concourent eux aussi à la préservation de l’intégrité du génome; ils permettent la réparation de nucléotides modifiés chimiquement, que ce soit à la suite d’agressions physico-chimiques de la molécule d’ADN par des rayonnements cosmiques, ultraviolets, par l’exposition à une source radioactive ou à des molécules réactives ou des radicaux libres. |
Les différents complexes MMR : de la bactérie à l’homme Le système MMR le mieux connu aujourd’hui est celui d’Escherichia coli, appelé système MutHLS, comprenant les protéines MutH, MutL, MutS. Ce système fonctionne selon un processus post-réplicatif d’excision-resynthèse d’une longue portion d’ADN comprenant un mésappariement [4]. Cette réparation se fait sur le brin néo-synthétisé, reconnu parce qu’hypométhylé. Un système MutHLS fonctionnel a pu être totalement reconstitué in vitro [5]. La protéine MutS reconnaît d’abord le mésappariement et se lie à l’ADN. En présence d’homodimères de MutS et de MutL, MutH est activée et, grâce à son activité endonucléasique, coupe le brin d’ADN néosynthétisé non méthylé en regard d’un site GATC situé sur l’autre brin méthylé [6]. L’excision requiert la présence de la protéine UvrD ou MutU (hélicase II), et d’une exonucléase simple brin (RecJ, Exo I, Exo VII ou encore Exo X). La resynthèse est alors possible grâce à l’holoenzyme ADN polymérase III, protéine de liaison à l’ADN simple brin, et à une ADN ligase. Ce modèle a servi de base pour analyser les mécanismes MMR intervenant dans d’autres espèces. Le Tableau I résume les principales homologies existantes entre les systèmes MMR de la bactérie, de la levure et de l’homme. Chez l’homme, les principaux complexes MMR sont essentiellement composés de protéines homologues des protéines MutL et MutS bactériennes (Figure 1) mais, contrairement à ce qui a pu être décrit chez les procaryotes (voir ci-dessus), le choix du brin d’ADN à réparer ne repose pas sur la reconnaissance de sites méthylés ou non. D’autres mécanismes ont été évoqués et le PCNA (proliferating cell nuclear antigen), co-facteur de la polymérase δ, pourrait jouer un rôle important dans ce contexte.
 | Tableau I.
Homologie des gènes MMR chez E. coli, S. cerevisiae et H. sapiens.
|
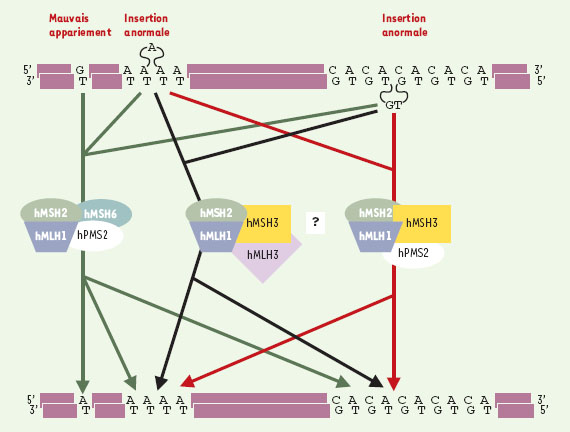
 | Figure 1. Les différents complexes du système MMR chez l’homme. Trois types de mésappariements de la molecule d’ADN sont donnés en exemple. Ils sont tous la cible des complexes MMR MSH2/MSH6 - MLH1/PMS2. En l’absence de MSH6, les complexes MSH2/MSH3 sont également capables d’intervenir dans certains cas, en s’associant soit aux complexes présomptifs MLH1/MLH3, soit aux complexes MLH1/PMS2. Les flèches précisent le (ou les) complexe(s) MMR impliqué(s) dans la reconnaissance et la réparation des mésappariements de la molécule d’ADN en fonction de leur nature (mauvais appariement, insertion anormale d’un ou de plusieurs nucléotides…). |
|
Anomalie du système MMR et cancers La fréquence des altérations génétiques des séquences répétées microsatellites de l’ADN dans certains cancers du côlon chez l’homme [7–9] a conduit à postuler l’existence d’anomalies de fonctionnement du système MMR. Cette hypothèse a été confirmée dans le syndrome dit de Lynch [10] ou syndrome HNPCC (hereditary non polyposis colorectal cancer). En effet, les gènes prédisposant à la survenue d’un tel syndrome de cancers héréditaires chez l’homme, mutés de manière constitutionnelle chez les patients atteints, se sont avérés être les homologues humains des gènes MMR de levure MSH2, MLH1, PMS1 et PMS2 [11–14]. L’inactivation somatique de certains de ces gènes dans des tumeurs de survenue sporadique arborant également un phénotype MSI-H a été par la suite démontrée. Dans ces cancers où le système MMR est déficient, les séquences répétées mono-, di-, tri- ou tétra-nucléotidiques sont particulièrement instables du fait de l’accumulation d’erreurs de réplication par glissement (slippage) de la polymérase, erreurs qui ne sont pas (ou mal) réparées. Cette instabilité nucléotidique est utilisée pour identifier ces tumeurs sur le plan moléculaire. Elles sont de ce fait nommées tumeurs MSI-H, ou microsatellite instabilityhigh, terme qui reflète l’instabilité des séquences répétées de type microsatellite de l’ADN [15]. Le phénotype MSI-H caractérise toute une série de tumeurs de localisation variable. Il s’agit d’un phénotype tumoral fréquent puisqu’il est présent chez l’homme dans 15 % environ des cancers du côlon, de l’estomac et de l’endomètre, et plus rarement dans d’autres localisations tumorales [15]. Cliniquement, les cancers MSI-H colorectaux sont de loin les mieux caractérisés. Ils surviennent plus fréquemment chez la femme que chez l’homme (3 femmes pour 2 hommes atteints). Leur pronostic est meilleur que celui des tumeurs non MSI, et il seraient plus sensibles à la chimiothérapie [16, 17]. Les cancers MSI-H ont, dans la grande majorité des cas, une localisation colique droite, depuis le cæcum jusqu’à l’angle splénique. Histologiquement, il s’agit d’adénocarcinomes mucineux de haut grade, qu’accompagne fréquemment un infiltrat lymphocytaire péritumoral. |
L’instabilité des microsatellites et son rôle dans la carcinogenèse MSI-H Les microsatellites sont constitués de la répétition en tandem d’un motif nucléotidique de une à quelques bases. Ils sont très nombreux et dispersés dans tout le génome. L’immense majorité d’entre eux est localisée dans des régions non codantes de l’ADN et, chez les individus normaux, ils sont souvent très polymorphes du fait de la variation importante d’un individu à l’autre du nombre des répétitions qui les composent. L’instabilité observée à leur niveau dans les cancers MSI-H est importante, mais ne devrait pas entraîner de conséquences phénotypiques secondaires lorsqu’elle affecte ces séquences répétées anonymes a priori non fonctionnelles. Certaines séquences répétées de l’ADN, principalement des microsatellites mono-nucléotidiques répétant une seule base en tandem, peuvent être parfois contenues, au contraire, dans la séquence codante de certains gènes humains. Ces gènes sont appelés gènes cibles de l’instabilité des cancers avec altération du système MMR. Leur responsabilité dans la carcinogenèse MSI-H, en raison de l’instabilité de ces répétitions, a été récemment démontrée. Parmi ces gènes, on trouve le gène codant pour le récepteur de type II du TGFβ (TGFβRII) [18], le gène du récepteur de l’IGFII (IGFIIR) [19], les deux gènes MMR humains hMSH3 et hMSH6 homologues de MutS [20], et le gène de la protéine pro-apoptotique BAX [21]. Plus de vingt autres gènes cibles de l’instabilité génétique de ces tumeurs se sont depuis ajoutés à cette liste (Figure 2). Une revue exhaustive récente rapporte les fréquences d’altérations de ces gènes cibles telles qu’elles sont publiées dans la littérature [22]. Certains d’entre eux sont impliqués dans une même voie de signalisation, comme par exemple les gènes de l’axine [23] et de la protéine TCF-4 [24], tous deux associés à la transduction du signal Wingless/Wnt. D’autres participent au contraire à des voies de signalisation différentes.
 | Figure 2. Les nombreux gènes cibles de l’instabilité génétique des cancers colorectaux MSI-H chez l’homme. Les microsatellites codants altérés à des fréquences variables dans les cancers MSI-H sont représentés par des rectangles blancs. L’effet attendu des mutations de ces gènes cibles au niveau de telles répétitions est variable. Il dépend de la fonction de ces gènes cibles qui est ici précisée dans chaque cas lorsqu’elle est connue, et de la localisation de ces microsatellites sur les ADN complémentaires qui les contiennent. L’échelle graduée de 0 à 100 %, représentée par les rectangles rouges, permet de préciser la position de la répétition sur la séquence codante du gène |
Les altérations de ces gènes sont observées à des fréquences variables dans ces cancers. Il s’agit de délétions ou d’insertions d’une ou deux bases dans une répétition codante qui décalent le cadre de lecture de la séquence codante dans laquelle elles surviennent, et sont donc susceptibles d’entraîner le plus souvent une inactivation par troncature de la protéine correspondante. Ces altérations ont été recherchées en premier lieu dans des gènes dont la perte de fonction pourrait être un événement transformant ou pourrait favoriser la progression tumorale. Selon le désormais classique modèle de Knudson, il s’agit alors de mutations dont l’effet attendu sur le phénotype tumoral ne sera obtenu que lorsque les deux allèles d’un même gène auront été inactivés (effet récessif). De telles altérations ont déjà été observées dans des gènes suppresseurs de tumeurs tels que TGFβRII [18], PTEN [25] ou RIZ [26], dans des gènes codant pour des facteurs pro-apoptotiques tels que BAX [21] ou Caspase-5 [27], ou encore dans des gènes de réparation de la molécule d’ADN tels que hMSH3 [20], hMSH6 [20], hMLH3 [28] ou RAD-50 [29]. Dans certains cas, ces mutations semblent pouvoir favoriser l’activité de la protéine qu’elles affectent plutôt que de l’inactiver. Dans ce cas, l’effet attendu (effet dominant) pourrait être déjà obtenu même si une mutation n’affecte qu’un seul allèle. C’est ce que nous avons proposé dans le cas de l’altération fréquente (40 % des cas) du gène TCF-4 au cours des cancers colorectaux MSI-H [30]. En effet, ces mutations, en décalant le cadre de lecture des transcrits mutés, suppriment les domaines codant normalement pour deux sites de liaison de TCF-4 à un partenaire protéique chargé de réprimer son activité (protéine CtBP, C-terminal binding protein). |
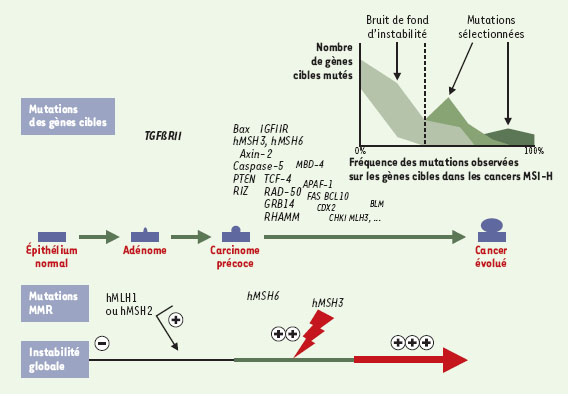
Le rôle des gènes cibles dans la carcinogenèse MSI-H Dans les cancers MSI-H, le nombre de gènes cibles connus pour être mutés par instabilité de répétitions codantes est aujourd’hui élevé. En conséquence, la difficulté pour les équipes de recherche travaillant sur la carcinogenèse MSI-H n’est plus tant de mettre en évidence de nouvelles altérations génétiques dans ces cancers, mais plutôt d’interpréter la signification du grand nombre des altérations décrites de ces gènes cibles. Compte tenu du bruit de fond d’instabilité élevé dans ces cancers, la présence de ce type de mutations ne signe pas forcément leur implication dans le processus de tumorigenèse. En 1998, au cours d’une réunion internationale de consensus, certains critères ont été proposés pour définir la notion de gène cible jouant un rôle dans la carcinogenèse MSI-H [15]. Ils ont été d’emblée sévèrement critiqués et ne seront donc pas détaillés ici [31]. Schématiquement, il s’agit aujourd’hui d’essayer de mettre en évidence, parmi ces nombreuses altérations, celles dont la survenue semble vraiment conférer un avantage sélectif au clone dans lequel elles surviennent, par rapport à de nombreuses autres dont la présence ne pourrait être que la conséquence de l’instabilité qui caractérise ces cancers, sans autres raisons fonctionnelles. Les preuves fonctionnelles qui permettraient d’affirmer ou d’infirmer la responsabilité de ces mutations au cas par cas sont malheureusement aujourd’hui rarement établies. Aussi, seules les plus fréquentes de ces mutations sont supposées jouer un rôle au cours de la carcinogenèse MSI-H en raison de leur sélection fréquente au cours de la progression de ces cancers. Par une approche statistique analysant la fréquence avec laquelle les gènes sont altérés dans ces cancers MSI-H du côlon, nous avons d’ailleurs pu montrer récemment que les gènes cibles affectés par ces altérations pouvaient être effectivement séparés en deux grands groupes bien distincts: (1) les gènes dont les mutations sont fréquentes et probablement sélectionnées au cours de la progression tumorale (mutations « fonctionnelles »); (2) les gènes dont les altérations sont plus rares et qui ne devraient donc pas jouer un rôle majeur dans la carcinogenèse MSI-H (mutations « bruit de fond ») (Figure 3) [29].
 | Figure 3. Les mutations affectant les gènes cibles de l’instabilité génétique des cancers du côlon MSI-H au cours de la progression tumorale. L’accumulation du nombre des altérations affectant les répétitions du génome tumoral (instabilité globale) est symbolisée par un trait d’épaisseur croissante. Parmi les nombreuses altérations des gènes cibles de l’instabilité génétique des cancers du côlon MSI-H, seules certaines d’entre elles semblent véritablement sélectionnées au cours de la progression tumorale. Elles peuvent jouer leur rôle au cours de la progression tumorale, soit de manière isolée, soit en synergie l’une avec l’autre. D’autres semblent associées au bruit de fond d’instabilité qui caractérise ces tumeurs, et ne devraient pas jouer un rôle majeur dans ces cancers. Une étude statistique de la distribution de la fréquence de ces nombreuses mutations utilisant la loi du maximum de vraisemblance permet, en l’absence de critères fonctionnels, de séparer ces deux types de mutations comme cela est indiqué dans l’encart. Ces mutations se divisent en mutations « bruit de fond », de fréquence basse, ne jouant probablement pas un rôle majeur au cours de la progression tumorale et en rapport avec l’instabilité génétique élevée de ces tumeurs, et les mutations « sélectionnées » observées à des fréquences plus élevées et qui, du fait de cette sélection, devraient jouer un rôle plus important dans ces cancers. Le gène TGFβRII est le plus précocement muté dans ces cancers MSI. Les autres gènes sont altérés dans un ordre qui peut varier. La taille des caractères des gènes est à peu près proportionnelle à la fréquence de leurs mutations rapportée dans la littérature. |
Lorsque l’on prend en compte l’ensemble des mutations des gènes cibles de l’instabilité génétique des cancers MSI-H du côlon publiées dans la littérature, on obtient pour ces altérations un profil analogue à celui qui est représenté sur la Figure 3. Les mutations peuvent être sélectionnées en étant hétérozygotes (effet dominant lié à un gain de fonction de la protéine mutée, ou effet lié à un mécanisme d’haplo-insuffisance classique) ou homozygotes (effet récessif lié à une perte de fonction de la protéine mutée). Dans certains cas, les processus de sélection de ces mutations sont interdépendants (effet additif de deux mutations affectant deux gènes cibles impliqués dans une même voie de carcinogenèse par exemple). Enfin, d’autres critères peuvent intervenir, taille et nature des répétitions, composition des séquences nucléotidiques voisines, critères de spécificité tissulaire modulant le rôle de tel ou tel gène en fonction de la localisation tumorale… |
L’instabilité génétique dans les cancers MSI-H : un processus évolutif lié au sexe Si des centaines de gènes cibles sont concernés par le phénomène d’instabilité de microsatellites codants de l’ADN dans les cancers MSI-H, des centaines de milliers de séquences répétées non codantes sont instables dans ces tumeurs. Dans certains cas, ces microsatellites sont localisés dans les régions introniques, ou 5’ ou 3’ non traduites de certains gènes humains, et il a été décrit qu’ils pouvaient jouer un rôle régulateur de l’expression de ces gènes (stabilisation des transcrits, séquences répétées promotrices, microsatellite intronique contrôlant l’épissage…). L’instabilité constatée de telles séquences pourrait intervenir au cours de la carcinogenèse MSI-H, mais cela reste à démontrer [32]. Cependant, dans l’immense majorité des cas, les séquences répétées non codantes du génome semblent être des séquences anonymes, et l’instabilité nucléotidique observée à leur niveau dans les tumeurs MSI-H ne devrait pas avoir de conséquences fonctionnelles. De telles séquences ont surtout été utilisées jusqu’alors dans un but diagnostique [15], et certaines d’entre elles ont pu être individualisées aujourd’hui comme des marqueurs sensibles et spécifiques de l’instabilité génétique des tumeurs dont le système MMR est déficient. Par exemple, les deux répétitions mononucléotidiques introniques, nommées Bat-25 et Bat-26 sont communément utilisées pour caractériser le phénotype MSI-H. Il s’agit de répétitions de respectivement 25 et 26 adénosines, dont l’instabilité est presque systématiquement observée dans les cancers MSI-H (délétions variant selon la tumeur de quelques bases à plus d’une dizaine de nucléotides) alors que ces microsatellites ont la particularité de rester très monomorphes dans les ADN de cellules normales ou ceux des tumeurs non MSI [33]. L’utilisation de Bat-25 et de Bat-26 a permis de constater que, de manière surprenante, l’intensité de l’instabilité de ces marqueurs dans les cancers du côlon MSI-H sporadiques, qui, rappelons-le, sont fréquents chez la femme, était liée au sexe [34]. En effet, en étudiant une grande série de patients, nous avons pu constater que les raccourcissements alléliques de ces microsatellites étaient plus importants en taille chez les femmes que chez les hommes. Ces résultats suggèrent que des facteurs dépendant du sexe mais encore inconnus, génétiques et/ou environnementaux, pourraient influencer le fonctionnement du système MMR chez l’homme. Dans les cancers du côlon MSI-H, Bat-25 et Bat-26 sont de plus en plus raccourcis au cours de la progression tumorale, ce qu’explique un processus d’instabilité probablement cumulatif: les délétions les plus petites en taille ont été observées dans les tumeurs MSI-H encore très peu évoluées, ou même dans des lésions pré-cancéreuses (polypes adénomateux) [35]. De plus, la taille de ces délétions est positivement corrélée au nombre d’altérations accumulées progressivement sur les nombreux gènes cibles de l’instabilité génétique dans le génome de ces tumeurs [29]. Ces observations suggèrent que ces microsatellites non codants de l’ADN seraient des marqueurs simples de progression tumorale. Nous les avons d’ailleurs utilisés afin de proposer un ordre le plus probable de survenue des mutations des gènes cibles de ces cancers [29]. Ainsi, l’altération du gène TGFβRII est apparue comme étant la plus précoce, ce qui est en accord avec l’observation selon laquelle il est souvent le seul gène cible connu muté dans des adénomes coliques MSI-H pré-cancéreux [36]. En revanche, l’altération du gène cible hMSH3 codant pour une protéine MMR, qui est systématiquement retrouvée dans des tumeurs arborant des délétions de grande taille de Bat-25 et de Bat-26, serait un événement capable de provoquer une augmentation significative des phénomènes d’instabilité génétique affectant les microsatellites au cours de la progression tumorale (Figure 3) [29]. |
Il existe de nombreux arguments pour penser que la plupart des cancers humains sont caractérisés par une instabilité génétique : instabilité chromosomique comme dans la grande majorité des tumeurs solides (conduisant à des phénomènes de gains ou de pertes d’allèles, des translocations réciproques conduisant à des gènes de fusion, des amplifications géniques) ou instabilité nucléotidique comme au niveau des séquences répétées microsatellites dans les tumeurs MSI-H. Ce phénomène est même considéré par de nombreux auteurs comme une condition absolument indispensable à l’établissement du processus de transformation cellulaire [37, 38]. Dans la plupart des cancers MSIH sporadiques, d’autres mécanismes de mutation que l’instabilité des microsatellites ont pu cependant être décrits, et notamment l’inactivation de certains gènes par méthylation de novo et bi-allélique de leurs séquences promotrices (en premier lieu hMLH1, responsable du phénotype MSI-H de ces cancers, mais aussi au niveau de gènes suppresseurs de tumeurs comme p16, ou encore APC). Malgré tout, la progression de ces tumeurs semble être un processus essentiellement permis et entretenu par l’accumulation de centaines de mutations au niveau de séquences répétées codantes ou même parfois non codantes (séquences dont l’instabilité pourrait modifier le niveau d’expression de certains gènes). Une meilleure compréhension de la carcinogenèse de ces tumeurs va désormais dépendre en grande partie du développement de nouvelles technologies récemment apparues, puces d’expression à ADN mais aussi puces tissulaires, qui devraient permettre de préciser, parmi ces nombreuses altérations, celles susceptibles de participer à la progression tumorale et pour lesquelles des conséquences fonctionnelles devraient être objectivées par ce type d’approche. |
1. Modrich P. Mechanisms and biological effects of mismatch repair. Annu Rev Genet 1991; 25: 229–53. 2. Schaaper RM, Radman M. The extreme mutator effect of Escherichia coli mutD5 results from saturation of mismatch repair by excessive DNA replication errors. EMBO J 1989; 8: 3511–6. 3. Cheng KC, Loeb LA. Genomic instability and tumor progression: mechanistic considerations. Adv Cancer Res 1993; 60: 121–56. 4. Wagner R, Meselson M. Repair tracts in mismatched DNA heteroduplexes. Proc Natl Acad Sci USA 1976; 73: 4135–9. 5. Lahue RS, Au KG, Modrich P. DNA mismatch correction in a defined system. Science 1989; 245: 160–4. 6. Radman M, Wagner RE, Glickman RW, Meselson M. DNA methylation, mismatch correction and genetic stability. Prog Environment Carcinogen 1984; 121–30. 7. Aaltonen LA, Peltomäki P, Leach FS, et al. Clues to the pathogenesis of familial colorectal cancer. Science 1993; 260: 812–6. 8. Thibodeau SN, Bren G, Schaid D. Microsatellite instability in cancer of the proximal colon. Science 1993; 260: 816–9. 9. Ionov Y, Peinado M, Malkhosyan S, Shibata D, Perucho M. Ubiquitous somatic mutations in simple repeated sequences reveal a new mechanism for colonic carcinogenesis. Nature 1993; 363: 558–61. 10. Lynch HT, Smyrk TC, Watson P, et al. Genetics, natural history, tumor spectrum, and pathology of hereditary nonpolyposis colorectal cancer: an updated review. Gastroenterology 1993; 104: 1535–49. 11. Bronner CE, Baker SM, Morrison PT, et al. Mutation in the DNA mismatch repair gene homologue hMLH1 is associated with hereditary non-polyposis colon cancer. Nature 1994; 368: 258–61. 12. Papadopoulos N, Nicolaïdes NC, Wei YF, et al. Mutation of a MutL homolog in hereditary colon cancer. Science 1994; 263: 1625–9. 13. Leach FS, Nicolaïdes NC, Papadopoulos N, et al. Mutation of a MutS homolog in hereditary non polyposis colorectal cancer. Cell 1993; 75: 1215–25. 14. Nicolaides NC, Papadopoulos N, Liu B, et al. Mutations of two PMS homologues in hereditary non polyposis colon cancer. Nature 1994; 371: 75–80. 15. Boland CR, Thibodeau SN, Hamilton SR, et al. A National Cancer Institute workshop on microsatellite instability for cancer detection and familial predisposition: development of international criteria for the determination of microsatellite instability in colorectal cancer. Cancer Res 1998; 58: 5248–57. 16. Elsaleh H, Joseph D, Grieu F, et al. Association of tumour site and sex with survival benefit from adjuvant chemotherapy in colorectal cancer. Lancet 2000; 355: 1745–50. 17. Hemminki A, Mecklin JP, Jarvinen H, Aaltonen LA, Joensuu H. Microsatellite instability is a favorable prognostic indicator in patients with colorectal cancer receiving chemotherapy. Gastroenterology 2000; 119: 921–8. 18. Markowitz S, Wang J, Myeroff L, et al. Inactivation of the type II TGF-β receptor in colon cancer cells with microsatellite instability. Science 1995; 268: 1336–8. 19. Souza RF, Appel R, Yin J, et al. Microsatellite instability in the insulin-like growth factor II receptor gene in gastrointestinal tumours. Nat Genet 1996; 14: 255–7. 20. Malkhosyan S, Rampino N, Yamamoto H, Perucho M. Frameshift mutator mutations. Nature 1996; 382: 499–500. 21. Rampino N, Yamamoto H, Ionov Y, et al. Somatic frameshift mutations in the BAX gene in colon cancers of the microsatellite mutator phenotype. Science 1997; 275: 967–9. 22. Duval A, Hamelin R. Mutations at coding repeat sequences in mismatch repair-deficient human cancer: toward a new concept of target genes for instability. Cancer Res 2002; 62: 2447–54. 23. Liu W, Dong X, Mai M, et al. Mutations in AXIN2 cause colorectal cancer with defective mismatch repair by activating betacatenin/ TCF signalling. Nat Genet 2000; 26: 146–7. 24. Duval A, Gayet J, Zhou XP, et al. Frequent frameshift mutations of the TCF-4 gene in colorectal cancers with microsatellite instability. Cancer Res 1999; 59: 4213–5. 25. Guanti G, Resta N, Simone C, et al. Involvement of PTEN mutations in the genetic pathways of colorectal cancerogenesis. Hum Mol Genet 2000; 9: 283–7. 26. Chadwick RB, Jiang GL, Bennington GA, et al. Candidate tumor suppressor RIZ is frequently involved in colorectal carcinogenesis. Proc Natl Acad Sci USA 2000; 97: 2662–7. 27. Schwartz S, Yamamoto H, Navarro M, et al. Frameshift mutations at mononucleotide repeats in caspase-5 and other target genes in endometrial and gastrointestinal cancer of the microsatellite mutator phenotype. Cancer Res 2000; 59: 2995–3002. 28. Loukola A, Vilkki S, Singh J, Launonen V, Aaltonen LA. Germline and somatic mutation analysis of MLH3 in MSI-positive colorectal cancer. Am J Pathol 2000; 157: 347–52. 29. Duval A, Rolland S, Compoint A, et al. Evolution of instability at coding and non-coding repeat sequences in human MSI-H colorectal cancers. Hum Mol Genet 2001; 10: 513–8. 30. Duval A, Rolland S, Tubacher E, et al. The human T-cell transcription factor-4 gene: structure, extensive characterization of alternative splicings, and mutational analysis in colorectal cancer cell lines. Cancer Res 2000; 60: 3872–9. 31. Perucho M. Correspondence re: C.R. Boland et al. A National Cancer Institute workshop on microsatellite instability for cancer detection and familial predisposition: development of international criteria for the determination of microsatellite instability in colorectal cancer (Cancer Res 1998; 58: 5248–57). Cancer Res 1999; 59: 249- 56. 32. Suraweera N, Iacopetta B, Duval A, et al. Conservation of mononucleotide repeats within 3’ and 5’ untranslated regions and their instability in MSI-H colorectal cancer. Oncogene 2001; 20: 7472–7. 33. Zhou XP, Hoang JM, Li YJ, et al. Determination of the replication error phenotype in human tumors without the requirement for matching normal DNA by analysis of mononucleotide repeat microsatellites. Genes Chromosom Cancer 1998; 21: 101–7. 34. Duval A, Iacopetta B, Thorntensen L, et al. Gender difference for mismatch repair deficiency in human colorectal cancer. Gastroenterology 2001; 121: 1026–7. 35. Pedroni M, Sala E, Scarselli A, et al. Microsatellite instability and mismatchrepair protein expression in hereditary and sporadic colorectal carcinogenesis. Cancer Res 2001; 61: 896–9. 36. Grady WM, Rajput A, Myeroff L, et al. Mutation of the type II transforming growth factor-β receptor is coincident with the transformation of human colon adenomas to malignant carcinomas. Cancer Res 1998; 58: 3101–4. 37. Loeb LA. Mutator phenotype may be required for multistage carcinogenesis. Cancer Res 1991; 51: 3075–9. 38. Hartwell L. Defects in a cell cycle checkpoint may be responsible for the genomic instability of cancer cells. Cell 1992; 71: 543–6. |