| |
| Med Sci (Paris). 2003 January; 19(1): 92–99. Published online 2003 January 15. doi: 10.1051/medsci/200319192.Microscopie à force atomique : de l’imagerie cellulaire à la manipulation moléculaire Marie-Cécile Giocondi,1 Pierre Emmanuel Milhiet,2 Eric Lesniewska,3 and Christian Le Grimellec1* 1Centre de Biochimie Structurale, Cnrs UMR 5048, Université Montpellier 1, Inserm U.554, 29, rue de Navacelles, 34090 Montpellier Cedex, France 2Laboratoire CRRET, Université Paris 12, 94000 Créteil Cedex, France 3Laboratoire de Physique, Cnrs URA 5027, UFR Sciences et Techniques, 9, rue Alain Savary, BP 47870, 21078 Dijon Cedex, France |
L’invention en 1982 du microscope à effet tunnel, qui permet de visualiser et de manipuler les atomes [
1], est à l’origine d’une nouvelle famille de microscopes, les microscopes à champ proche ou à sonde locale, dédiés essentiellement à l’étude des propriétés de surface des échantillons. Ils sont caractérisés par l’absence de tout système de lentille optique ou électromagnétique. Leur principe consiste à positionner une sonde (généralement une pointe effilée) à une distance de la surface à étudier de l’ordre du nanomètre et à déterminer localement, point par point, la valeur d’une grandeur physique choisie (transfert d’électrons, flux de photons ou d’ions, topographie ou température). Ce principe apparente ces microscopes aux systèmes de micromanipulation cellulaire, couplés à des prélèvements ou à des mesures locales via des micropipettes ou des microélectrodes, qui sont couramment utilisés en physiologie ou en biologie. Le microscope à force atomique (AFM, atomic force microscope) [
2] permet d’observer les atomes à la surface d’échantillons cristallins, sous vide ou dans l’air. Il peut aussi fonctionner en milieu liquide, propriété qui a très rapidement permis le transfert de cette technologie à la biologie [
3]. En dix ans, des progrès considérables ont été réalisés tant dans la compréhension des phénomènes régissant l’interaction entre la pointe d’un microscope à force atomique et la surface d’échantillons biologiques (matière molle), que dans l’instrumentation elle-même. Aux applications topographiques qui vont de l’imagerie de molécules dans une solution saline, avec une résolution latérale actuelle de 5 Å, à l’imagerie cellulaire 3-D en passant par le domaine mésoscopique (meso: au milieu) (~10-300 nm), se sont ajoutées les mesures de forces d’interaction intra- et intermoléculaires, regroupées sous le terme « spectroscopie de force », ainsi que les techniques de manipulation et de chirurgie moléculaires [
4]. Par ses capacités, le microscope à force atomique s’est imposé - au carrefour de la biologie, de la chimie et de la physique - comme un outil indispensable au développement des nanosciences. |
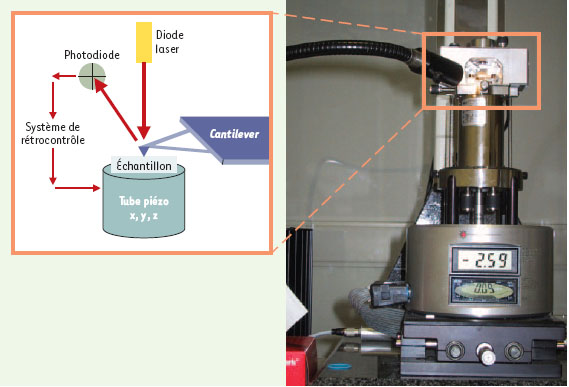
Principe et modes de fonctionnement du microscope à force atomique Le principe consiste à amener, soit au contact de la surface à étudier, soit à une distance de quelques Å, une pointe dont l’extrémité a un rayon de quelques nm, et à lui faire balayer ligne par ligne la topographie de cette surface en évitant de la déformer. Cette pointe, généralement en nitrure de silicium (Si3N4), se situe à l’extrémité d’un levier flexible, le cantilever. La déflexion du cantilever, provoquée par la présence d’une aspérité, entraîne l’intervention d’une boucle de rétrocontrôle qui va maintenir constante la distance, et donc la force d’interaction, entre la pointe et l’échantillon (Figure 1). En milieu liquide, selon un modèle simplifié, cette force d’interaction peut être considérée comme la résultante des forces électrostatiques attractives ou répulsives à longue portée, des forces attractives de van der Walls qui décroissent très rapidement à quelques nanomètres de la surface, et de la force de répulsion qui s’exerce lorsque les atomes viennent au contact. Le « mode contact », mode de fonctionnement dans lequel la pointe et la surface restent en contact ou quasi-contact, permet d’obtenir des images à haute résolution sur des objets peu rugueux [4]. Durant un balayage, trois types d’images peuvent être collectés simultanément: les images topographiques (encore appelées images « iso-force » ou images hauteur), les images de déflexion correspondant à la dérivée des mouvements de réajustement de la position relative entre la pointe et l’échantillon qui font ressortir les détails de la surface, et les images de friction résultant de la torsion du cantilever. Ces phénomènes de friction, susceptibles d’endommager l’échantillon biologique, sont à l’origine du développement du « mode oscillant avec contact » (tapping mode) où la pointe oscille à la fréquence de résonance du cantilever et ne touche que de façon transitoire la surface de l’échantillon. Au-delà des informations topographiques, ce mode permet d’apprécier les propriétés viscoélastiques locales via un signal de phase corrélé à la dissipation d’énergie lors de chaque contact transitoire. Il est particulièrement utile pour l’étude d’échantillons faiblement adsorbés sur leur support ou de topographie très irrégulière, comme des cellules. En effet, la microscopie à force atomique impose que l’échantillon soit adsorbé sur un support plan et y adhère suffisamment pour n’être pas déplacé sous l’effet du balayage par la pointe. Les supports les plus utilisés pour analyser en haute résolution des molécules isolées ou des complexes moléculaires sont le mica (échantillons hydrophiles) et le graphite orienté HOPG (échantillons hydrophobes) qui présentent de grandes plages planes à l’échelle atomique. Pour l’imagerie de cellules intactes ou d’extraits subcellulaires, les substrats généralement utilisés sont des lamelles de verre traitées ou non par des agents facilitant l’ancrage comme la polylysine.
 | Figure 1.
Schéma de principe et équipement courant de microscopie à force atomique. L’équipement présenté est un microscope à force atomique Nanoscope (Digital Instruments, USA). Un faisceau laser dirigé sur l’extrémité du cantilever est réfléchi vers une photodiode à deux ou quatre cadrans. Les déplacements du faisceau sur la photodiode permettront de détecter les mouvements du cantilever. Le positionnement dans les trois plans de l’espace (x, y, z), avec une précision voisine de la taille de l’atome, de la pointe par rapport à l’échantillon durant le balayage est assuré par des céramiques piézo-électriques. Le système est piloté par un ordinateur qui reconstitue les informations recueillies par la pointe sous forme d’images 3-D. |
Un élément essentiel à prendre en considération en microscopie à force atomique est la taille (et la forme) des objets à analyser. Les éléments piézoélectriques qui assurent les déplacements de la pointe ou de l’échantillon limitent actuellement à 100 μm environ la taille du balayage dans le plan avec une amplitude verticale maximum de 7 à 10 μm. Il est donc impossible d’effectuer une prévisualisation à faible grossissement pour sélectionner la zone d’intérêt comme cela se fait en microscopie électronique. Enfin, le temps d’acquisition d’une image varie de 1 à 10 min selon l’ampleur du balayage et la nature de l’échantillon. L’utilisation d’un microscope à force atomique en biologie diffère de son utilisation en physique et en chimie par la nécessité d’utiliser des forces de balayage aussi faibles que possible pour éviter la déformation des objets mous. Dans les circonstances favorables, la pointe glisse à une distance de 5 à 10 Å de la surface sans la toucher, ce qui permet d’obtenir une excellente résolution sur des objets non déformés. |
Imagerie cellulaire L’obtention d’images 3-D de la surface de cellules dans un environnement physiologique (Figure 2) fut l’un des premiers objectifs biologiques de la microscopie à force atomique. Les résultats obtenus à partir de cellules eucaryotes [
5,
6], procaryotes [
7] et végétales [
8], qui ont fait l’objet d’une revue récente [
9], démontrent que ce microscope peut fournir des informations topographiques de la surface cellulaire, immergée dans du tampon, avec une résolution nettement supérieure à celle d’un microscope optique classique ou confocal et souvent supérieure à celle d’un microscope électronique à balayage. Le recueil et la validité des informations dépendent avant tout du soin apporté à limiter la force appliquée durant le balayage, et de la résistance mécanique des cellules étudiées. Pour les cellules eucaryotes, l’observation de l’apparition, en fonction du temps, de structures en forme de puits (diamètre compris entre 100 et 180 nm), responsables de l’exocytose à la surface de cellules vivantes d’acinus du pancréas dont les protéines Go et Gi ont été stimulées par le mastoparan, illustre bien les possibilités du microscope à force atomique [
10].
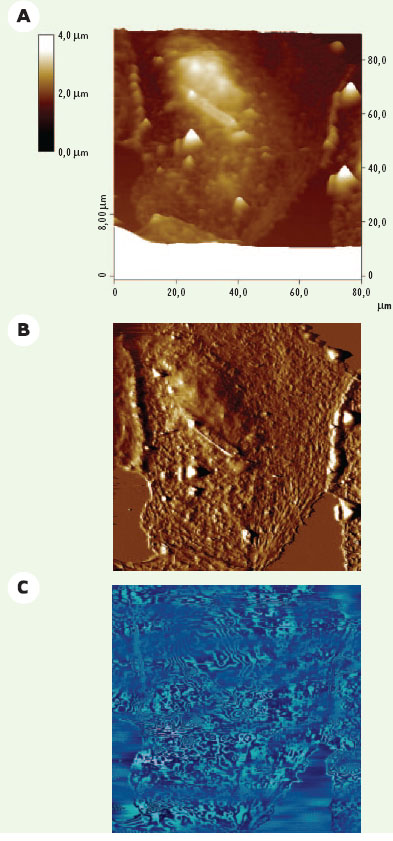
 | Figure 2.
Imagerie d’une cellule CV-1 vivante par microscopie à force atomique en mode contact. Les cellules CV-1, cultivées sur lamelle de verre, sont examinées dans un mélange (1/1, V/V) de solution saline tamponnée (PBS, pH 7,4) et de milieu de culture. Les trois images sont obtenues simultanément au cours du même balayage, effectué avec une force < 150 pN. A. Image hauteur (topographie): cette image tridimensionnelle montre le relief de la surface cellulaire en respectant les distances verticales données par une échelle de couleur présentée à gauche. B. Image de déflexion: les détails de la surface apparaissent plus nettement mais l’information dans la troisième dimension n’est plus quantitative. De telles images ressemblent à celles que l’on peut obtenir par microscopie électronique à balayage. C. Image de friction: l’interprétation de ces images sur des systèmes complexes reste à développer. |
L’utilisation du mode oscillant présente l’avantage d’offrir simultanément des images de la topographie de surface et de l’organisation du cytosquelette sousmembranaire [9]. La comparaison des images recueillies à partir de cellules intactes ou traitées par un détergent (Triton X-100) permet, par exemple, de mettre en évidence les relations entre les domaines membranaires résistants aux détergents (DRM) et le cytosquelette [
11]. Récemment, des images 3-D ont pu décrire, avec une résolution latérale de l’ordre de 5 nm et une résolution verticale voisine de 1 nm, la structure de la paroi latérale de cellules ciliées de l’oreille interne, fixées et examinées dans du tampon [9]. La découverte de la complexité de l’organisation topographique membranaire à cette échelle implique le développement de nouvelles techniques d’identification des structures observées. Parmi ces dernières, l’utilisation de pointes dites « fonctionnalisées » sur lesquelles ont été fixés un anticorps spécifique [
12] ou des groupements réactifs [
13] ouvre de nombreuses perspectives. En ce qui concerne les cellules vivantes, l’obtention d’une résolution à l’échelon moléculaire de la membrane se heurte cependant à des difficultés liées, d’une part, à la présence de glycocalyx ou de matrice extracellulaire plus ou moins homogènes et abondants, et, d’autre part, à la dynamique de la surface cellulaire, comme la diffusion latérale de ses constituants à une vitesse supérieure à celle du balayage. Récemment, la technique a pu être appliquée avec succès à des tranches de tissus [
14]. Parallèlement à ces études structurales, le microscope à force atomique a été utilisé pour estimer les propriétés mécaniques locales des cellules: en appliquant une force assez élevée [~500 picoNewtons (pN)] et en supprimant le balayage, la pointe déforme la surface cellulaire. L’ampleur de cette déformation verticale permet d’estimer l’élasticité (module de Young) de la zone sous la pointe. Cette opération peut s’effectuer en différents endroits d’une cellule, dans différentes conditions physiologiques [
15], par exemple dans des cellules endothéliales soumises aux forces de cisaillement créées par le flux sanguin. À ce jour, l’imagerie de cellules intactes reste un exercice délicat, difficilement envisageable en routine. Membranes isolées L’analyse de la topographie des fragments membranaires, purifiés par les méthodes de fractionnement sub-cellulaires et adsorbés sur du mica, révèle en général une surface d’où émergent de très nombreuses structures, le plus souvent globulaires, qui très probablement correspondent aux protéines membranaires [
16]. D’autres structures fréquemment observées, en forme de couronnes de 10 à 20 nm de diamètre entourant une dépression centrale, suggèrent une organisation de type pore transmembranaire (Figure 3). Comme pour les cellules intactes, l’exploitation de ces informations va dépendre du développement des méthodes d’identification locale. Certaines protéines membranaires, comme celles des jonctions communicantes chez les eucaryotes ou la bactériorhodopsine chez Halobacterium halobium, s’organisent spontanément en un réseau bidimensionnel pour exercer leur fonction. Les jonctions communicantes sont purifiées sous forme de plaques composées de deux membranes superposées et déposées sur le mica. Les faces membranaires cytoplasmiques examinées dans du tampon apparaissent peu structurées. La pointe, utilisée comme instrument de nanodissection, permet d’ôter la membrane supérieure et d’exposer la face extracellulaire de la deuxième membrane. L’arrangement en réseau hexagonal des canaux individuels (connexons) formant la jonction, qui émergent de la surface de 1,4 nm, peut alors s’observer avec une résolution latérale de 2,5 nm [
17]. L’organisation trimérique de la bactériorhodopsine, pompe à protons à sept hélices transmembranaires, est révélée avec une résolution latérale pouvant atteindre 5 Å et une résolution verticale entre 1 et 2 Å [ 4].
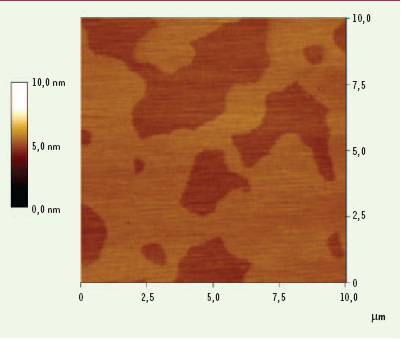
 | Figure 3.
Image d’une membrane de plante: plasmalemme d’épinard. Des structures en forme de pores d’un diamètre voisin de 20 nm émergent de la surface membranaire d’une hauteur de 1 à 2 nm. Elles sont formées de quatre sous-unités entourant une dépression centrale et apparaissent dans le voisinage de plus larges structures (la membrane est dans du tampon). Image en 3-D. X et Y = 500 nm. (Travail réalisé en collaboration avec M. Crèvecœur, Université de Genève, Suisse). |
À l’échelle mésoscopique, l’étude de la structure de l’enveloppe nucléaire excisée d’ovocytes de xénope suggère l’existence de pores de petite taille à la périphérie des complexes qui forment les pores nucléaires de grande taille (NPC). Ces pores de petite taille s’ouvrent en fonction de la concentration de Ca2+ et d’ATP et pourraient constituer une voie de passage préférentielle pour les ions, les NPC étant réservés au transport des macromolécules [
18]. Systèmes membranaires reconstitués L’étude des propriétés de systèmes modèles, monocouches et bicouches lipidiques, dans lesquelles sont insérés ou non un peptide ou une protéine, constitue une étape importante pour la compréhension des relations structure-fonction dans les membranes biologiques. Le microscope à force atomique a donné de nombreuses informations, non accessibles par d’autres techniques, sur l’existence, la forme et la taille des microdomaines membranaires [
19,
20]. L’étude de leur cinétique de formation, dans une membrane artificielle constituée d’un mélange de deux phospholipides, suggère fortement que les phénomènes à la base de l’élaboration des microdomaines (Figure 4) des membranes biologiques obéissent, avec des coefficients différents, aux lois qui commandent la formation des alliages métalliques. Pour différents peptides, l’examen des relations lipidepeptide en mono- et bicouche révèle l’existence d’assemblages supramoléculaires, qui n’avaient pas été détectés précédemment, ce qui impose une ré-évaluation de leur mode d’action [
21].
 | Figure 4.
Visualisation de microdomaines membranaires. Les microdomaines sont dans une bicouche lipidique formée d’un mélange de phosphatidylcholine et de sphingomyéline (1:3) et 20 % de cholestérol. L’examen de bicouches constituées de mélanges de lipides permet de caractériser certains des paramètres intervenant dans la formation des microdomaines (rafts) présents dans les membranes biologiques. Dans cet exemple, les domaines enrichis en sphingomyéline et en cholestérol, qui apparaissent plus clairs, sont à la fois larges et connectés. Si la concentration en cholestérol augmente, leur taille diminuera et ils se déconnecteront. Ils disparaîtront pour une concentration de cholestérol de 33 %. |
Visualiser la topographie des protéines membranaires reconstituées en cristal bidimensionnel, avec une résolution latérale qui peut atteindre 5 Å, représente actuellement une application importante de la microscopie à force atomique en biologie [4,
22,
23]: les images en 3-D très contrastées, obtenues dans des tampons physiologiques, permettent de suivre d’éventuels changements de conformation spontanés ou induits. C’est le cas des hexamères qui constituent la paroi intermédiaire de Deinococcus radiodurans dont le pore peut se présenter sous une conformation ouverte ou fermée [22]. De même, l’observation en temps réel, sous le microscope, des changements de conformation de la porine OmpF d’Escherichia coli induits par une différence de potentiel ou une variation de pH [
24], illustrent les possibilités qu’offre la technique dans l’étude des relations structure-fonction (Figure 5). Pouvoir observer des détails topographiques à une résolution latérale de 1 nm sur des protéines non cristallisées [
25] constitue un atout supplémentaire de la microscopie à force atomique pour la biologie structurale.
 | Figure 5.
Modification de la surface de la porine OmpF sous l’effet d’une différence de potentiel transmembranaire. A. L’examen, dans du tampon, de la surface d’une membrane de porine OmpF reconstituée sous forme de cristal 2-D, permet d’identifier l’organisation en trimères (marqués par les cercles) qui apparaissent sous forme de structures hautes de 13 Å. B. L’application d’une différence de potentiel transmembranaire provoque un changement de la conformation des protéines qui se rétractent; leur hauteur n’est plus que de 6 Å. C. Avec un potentiel transmembranaire peu élevé, les deux formes, étendue (de couleur claire) et rétractée (plus sombres), peuvent co-exister (rangées de cercles). (Image obtenue par D.J. Müller et A. Engel, Université de Bâle, Suisse [ 24]). |
|
Le microscope électronique était, jusqu’à l’arrivée du microscope à force atomique, le seul capable de caractériser la structure des brins d’ADN isolés et celle des complexes ADN-protéine. Les premières études réalisées à l’air montrèrent qu’il était possible d’obtenir une image de l’ADN avec une très bonne résolution. Depuis, le microscope à force atomique a permis d’étudier les changements de conformation de l’ADN induits par la liaison de ligands ou de protéines, la condensation de l’ADN sous l’influence des polycations, la détermination de la stœchiométrie de l’interaction protéine-ADN au site de liaison et la structure des nucléosomes et de la chromatine [
26–
29]. La structure tertiaire de l’ADN super-enroulé en présence de concentrations ioniques variables a également été caractérisée. L’imagerie en milieu liquide a permis, là aussi, de se rapprocher des conditions physiologiques. Elle a rendu possible l’étude des mouvements et de la cinétique de déplacement d’une seule molécule d’ARN polymérase le long d’un brin d’ADN [
30], ou encore le clivage, en fonction du temps, de l’ADN par l’endonucléase EcoKI [
27]. |
Autres exemples d’imagerie Le microscope à force atomique semble particulièrement adapté à l’étude des peptides et des protéines amyloïdes comme les peptides Aβ-40 et Aβ-42 impliqués dans la maladie d’Alzheimer, l’amyline pancréatique ou encore les protéines prion. La formation des fibrilles amyloïdes, ainsi que leur structure, peuvent s’observer en temps réel [
31]. Ces expériences devraient aider à comprendre la formation des plaques amyloïdes et à en sélectionner les inhibiteurs. La structure des éléments du cytosquelette (filaments d’actine, microtubules) peut aussi s’observer en solution, avec une résolution de quelques nanomètres (Figure 6). La topographie du complexe oligomérique de la protéine chaperon GroES a été obtenue en milieu liquide avec une résolution latérale de 1 nm [
32]. Une telle résolution est exceptionnelle pour des protéines solubles dont l’étude par microscopie à force atomique reste très délicate et n’a toujours pas donné de résultats vraiment convaincants. Pour les cristallographes, le microscope à force atomique est devenu un outil précieux d’étude in situ des cristaux biologiques de protéines, d’acides nucléiques et de virus, et permet de définir le mécanisme et la cinétique de leur croissance [
33,
34].
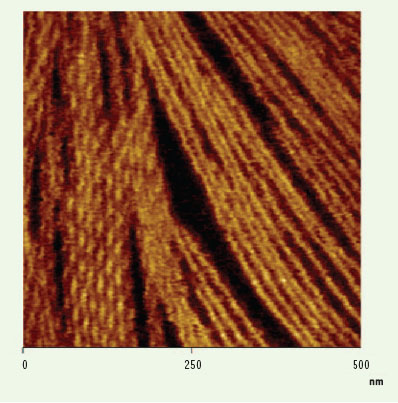
 | Figure 6.
Faisceaux de filaments d’actine. Imagerie dans du tampon. Travail en collaboration avec C. Roy, Université Montpellier II. |
|
Mesure des forces d’interaction moléculaire par microscopie à force atomique La mesure de la force d’interaction entre la pointe et l’échantillon, sur laquelle repose l’imagerie par microscopie à force atomique, a servi également à déterminer les forces de liaison entre deux molécules. Les premières expériences furent réalisées sur le couple avidine-biotine [
35]. Elles furent suivies de très nombreux travaux sur la mesure des forces d’interaction inter- et intramoléculaires, incluant les interactions entre les paires de bases dans l’ADN (~9 pN pour A-T, ~20 pN pour G-C) [
36], entre un anticorps et son antigène, un ligand et son récepteur ou entre polysaccharides [
37]. Ce mode expérimental (« spectroscopie de force ») [4], peut également s’appliquer à la détermination des forces d’adhérence entre cellules vivantes [
38]. Une utilisation importante concerne l’étude des forces associées au dépliement mécanique et au repliement spontané des protéines [
39]. Dans ces expériences, la pointe du microscope sert à étirer la protéine fixée à son support. L’extension de la protéine puis le dépliement de ses différents domaines entraînent des modifications de la position du cantilever qui permettent de déterminer la valeur des forces assurant leur cohésion. Associés à la biologie moléculaire et structurale (résonance magnétique nucléaire, cristallographie), ces travaux devraient aider à comprendre les facteurs déterminant la stabilité des domaines protéiques. Spectroscopie de force et imagerie à haute résolution des protéines membranaires peuvent se combiner [
40]. Après l’obtention d’une image des trimères de la bactériorhodopsine, la pointe est appuyée sur le COOH terminal de la protéine puis progressivement soulevée. Les hélices transmembranaires sont alors extraites deux par deux de la surface, ce qui permet de mesurer leurs forces d’ancrage dans la membrane, forces comprises entre 100 et 200 pN. Le balayage subséquent de la même zone confirmera l’extraction de la protéine. |
Conclusions et perspectives Le microscope à force atomique permet d’observer, dans du tampon, la surface d’échantillons biologiques avec des résolutions pouvant atteindre 5 Å en latéral et 1 à 2 Å en vertical. Ces performances en font un instrument de choix pour contribuer - avec la microscopie électronique et optique, la diffraction X et la résonance magnétique nucléaire - à la compréhension des relations structure-fonction dans les systèmes biologiques. Les multiples applications développées en dix ans attestent du large éventail de sujets que cet outil permet d’aborder, de l’étude des changements de conformation d’une protéine ayant une fonction de canal à l’effet de l’ablation laser sur la structure de la cornée, en passant par la formation des fibrilles amyloïdes et la caractérisation des interactions ADN-protéine. La spectroscopie de force, combinée ou non à l’imagerie à haute résolution, apparaît également comme un outil extrêmement utile pour la détermination des forces s’exerçant entre deux molécules uniques. Son application aux cellules devrait jouer un rôle important dans la compréhension des mécanismes d’adhérence cellulaire et de ses dysfonctionnements. La pointe du microscope, déjà utilisée comme instrument de nanodissection, sera certainement amenée à jouer un rôle dans la nanoconstruction d’objets soit purement biologiques, soit en association à d’autres composants, par exemple électroniques. De nombreuses améliorations techniques comme l’utilisation de pointes terminées par des nanotubes de carbone, la compensation par la pression de radiation des mouvements spontanés du cantilever à très faible force, l’obtention d’images à une vitesse vidéo, sont actuellement développées par divers laboratoires et devraient être prochainement offertes aux utilisateurs. Les progrès récents dans la compréhension de la formation des images en microscopie de champ proche optique, technique qui permet aussi la détection et la localisation de la fluorescence émise par une molécule unique, entretiennent l’espoir de pouvoir, dans un avenir proche, combiner topographie et identification moléculaire. |
1. Binig G, Rohrer H, Gerber C, Weibel E. Surface studies by scanning tunneling microscopy. Phys Rev Lett 1982; 49: 57–61. 2. Binnig G, Quate CF, Gerber C. Atomic force microscope. Phys Rev Lett 1986; 56: 930–3. 3. Kasas S. La microscopie à force atomique dans la recherche en biologie. Med Sci 1992; 8: 140–8. 4. Engel A, Gaub HE, Müller DJ. Atomic force microscopy: a forceful way with single molecules. Curr Biol 1999; 9: R133–6. 5. Radmacher M, Tillmann RW, Fritz M, Gaub HE. From molecules to cells: imaging soft samples with the atomic force microscope. Science 1992; 257: 1900–5. 6. Le Grimellec C, Lesniewska E, Cachia C, Schreiber JP, de Fornel F, Goudonnet JP. Imaging the membrane surface of MDCK cells by atomic force microscopy. Biophys J 1994; 67: 36–41. 7. Dufrêne YF. Atomic force microscopy of microbial cells. Microsc Analysis 2001; 3: 27–9. 8. Crèvecoeur M, Lesniewska E, Vié, V, Goudonnet JP, Greppin H, Le Grimellec C. Atomic-force microscopy imaging of plasma membranes purified from spinach leaves. Protoplasma 2000; 212: 46–55. 9. Le Grimellec C, Lesniewska E, Milhiet PE, Giocondi MC. AFM imaging of cells and membranes. In: Jena BP, ed. Atomic force microscopy in cell biology. Methods in cell biology. New York: Academic Press, 2002; 68: 51–65. 10. Schneider SW, Sritharan KC, Geibel JP, Oberleithner H, Jena BP. Surface dynamics in living acinar cells imaged by atomic force microscopy: identification of plasma membrane structures involved in exocytosis. Proc Natl Acad Sci USA 1997; 94: 316–21. 11. Giocondi MC, Vié V, Leniewska E, Goudonnet JP, Le Grimellec C. In situ imaging of detergentresistant membranes by atomic force microscopy. J Struct Biol 2000; 131: 38–43. 12. Hinterdorfer P, Baumgartner W, Gruber HJ, Schilcher K, Schindler H. Detection and localization of individual antibodyantigen recognition events by atomic force microscopy. Proc Natl Acad Sci USA 1996; 93: 3477–81. 13. Wong SW, Joselevich E, Woolley AT, Cheung CL, Lieber CM. Covalently functionalized nanotubes as nanometer-sized probes in chemistry and biology. Nature 1998; 394: 52–5. 14. Tsilimbaris MK, Lesniewska E, Lydataki S, Le Grimellec C, Goudonnet JP, Palikaris IG. The use of atomic force microscopy for the observation of corneal epithelium surface. Invest Ophthalmol Vis Sci 2000; 41: 680–6. 15. Matzke R, Jacobson K, Radmacher M. Direct, highresolution measurement of furrow stiffening during division of adherent cells. Nat Cell Biol 2001; 3: 607- 10. 16. Le Grimellec C, Lesniewska E, Giocondi MC, Cachia C, Shreiber JP, Goudonnet JP. Imaging of the cytoplasmic leaflet of the plasma membrane by atomic force microscopy. Scanning Microsc 1995; 9: 401–11. 17. Hoh JH, Sosinsky GE, Revel JP, Hansma PK. Structure of the extracellular surface of the gap junction by atomic force microscopy. Biophys J 1993; 65: 149–63. 18. Shahin V, Danker T, Enss K, Ossig R, Oberleithner H. Evidence for Ca2+ and ATPsensitive peripheral channels in nuclear pore complexes. FASEB J 2001; 15: 1895–901. 19. Giocondi MC, Vié V, Lesniewska E, Milhiet PE, Zinke-Allmang M, Le Grimellec C. Imaging the phase topology and growth of domains in supported lipid bilayers. Langmuir 2001; 17: 1653–9. 20. Milhiet PE, Giocondi MC, Le Grimellec C. Cholesterol is not crucial for the existence of microdomains in kidney brush-border membrane models. J Biol Chem 2002; 277: 875–8. 21. Vié V, Van Mau N, Chaloin L, Lesniewska E, Le Grimellec C, Heitz, F. Detection of peptide-lipid interactions in mixed monolayers using isotherms, atomic force microscopy and Fourier transform infrared analyses. Biophys J 2000; 78: 846–56. 22. Engel A, Müller DJ. Observing single biomolecules at work with the atomic force microscope. Nat Struct Biol 2000; 7: 715–8. 23. Reviakine I, Bergsma- Schutter W, Brisson A. Growth of protein 2-D crystals on supported planar lipid bilayers imaged in situ by AFM. J Struct Biol 1998; 121: 356–61. 24. Müller DJ, Engel A. Voltage and pH-induced channel closure of porin OmpF visualized by atomic force microscopy. J Mol Biol 1999; 285: 1347–51. 25. Milhiet PE, Vié V, Giocondi MC, Le Grimellec C. AFM characterization of model rafts in supported bilayers. Single Mol 2001; 2: 109–12. 26. Le Cam E, Frechon D, Barray M, Fourcade A, Delain E. Observation of binding and polymerisation of Fur repressor onto operatorcontaining DNA with electron and atomic force microscopes. Proc Natl Acad Sci USA 1994; 91: 11816–20. 27. Hansma HG. Surface biology of DNA by atomic force microscopy. Annu Rev Phys Chem 2001; 52: 71–92. 28. Ladoux B, Quivy JP, Doyle P, du Roure O, Almouzni G, Viovy JL. Fast kinetics of chromatin assembly revealed by singlemolecule videomicroscopy and scanning force microscopy. Proc Natl Acad Sci USA 2000; 97: 14251–6. 29. Sanchez A, Thimonier J, Mariley M, Rocca-Serra J, Barbet J. Accuracy of AFM measurements on the contour length of DNA fragments adsorbed on mica in air and aqueous buffer. Ultramicroscopy 2002; 92: 151–8. 30. Bustamante C, Guthold M, Zhu X, Yang G. Facilitated target location on DNA by individual Escherichia coli RNA polymerases molecules observed with the scanning force microscope operating in liquid. J Biol Chem 1999; 274: 16665–8. 31. Ding TT, Harper JD. Analysis of amyloid-β asemblies using tapping mode atomic force microscopy under ambient conditions. In: Abelson JN, Simon MI, eds. Amyloids, prions and other protein aggregates. Methods in enzymology. New York : Academic Press, 1999; 309: 510–25. 32. Mou JX, Czajkowski DM, Sheng SJ, Ho RY, Shao ZF. High resolution surface structure of E. coli GroES oligomer by atomic force microscopy. FEBS Lett 1996; 381: 161–4. 33. McPherson A, Malkin AJ, Kuznetsov YG. Atomic force microscopy in the study of macromolecular crystal growth. Annu Rev Biophys Biomol Struct 2000; 29: 361–410. 34. Zhu DW, Lorber B, Sauter C et al. Correlation between growth kinetics and properties of a novel crystal form of Thermus thermophilus aspartyltRNA synthetase-1. Acta Cryst D 2001; 57: 552–8. 35. Florin EL, Moy VT, Gaub HE. Adhesion forces between individual ligand-receptor pairs. Science 1994; 264: 415–7. 36. Rief M, Clausen- Schaumann H, Gaub HE. Sequence dependent mechanics of single DNA molecules. Nat Struct Biol 1999; 6: 346–9. 37. Willemsen OH, Snel MM, Cambi A, Greve J, De Grooth BG, Figdor CG. Biomolecular interactions measured by atomic force microscopy. Biophys J 2000; 79: 3267–81. 38. Benoit M, Gabriel D, Gerisch G, Gaub HE. Discrete interactions in cell adhesion measured by single-molecule force spectroscopy. Nat Cell Biol 2000; 6: 313–7 39. Fisher TE, Oberhauser AF, Carrion-Vazquez M, Marszalek PE, Fernandez JM. The study of protein mechanics with the atomic force microscope. Trends Biochem Sci 1999; 24: 379–84. 40. Oesterhelt F, Oesterhelt D, Pfeiffer M, Engel A, Gaub HE, Müller DJ. Unfolding pathways of individual bacteriorhodopsins. Science 2000; 288: 143–6. |