| |
| Med Sci (Paris). 2003 December; 19(12): 1175–1179. Published online 2003 December 15. doi: 10.1051/medsci/200319121175.Liaisons « accrocheuses » et mécanique moléculaire Anne Pierres, Anne-Marie Benoliel, and Pierre Bongrand* 1Laboratoire d’Immunologie, Inserm U. 387, Cnrs FRE 2059, Université de la Méditerranée, Hôpital de Sainte-Marguerite, BP 29, 13274 Marseille Cedex 09, France MeSH keywords: Sites de fixation, Communication cellulaire, Humains, Ligands, Liaison aux protéines, Sélectines |
> Dans un récent article [
1], Cheng Zhu et al. décrivent pour la première fois la propriété des molécules de P-sélectine de former avec leur ligand des « liaisons accrocheuses » (catch bonds), c’est-à-dire des liaisons dont la durée de vie augmente lorsqu’elles sont soumises à des contraintes mécaniques. Ce travail démontre la réalité d’un concept imaginé par un théoricien, Micah Dembo, quinze ans auparavant [
2]. Il marque aussi une étape supplémentaire dans un domaine de recherche qui s’est montré très actif au cours de la dernière décennie : l’observation directe des liaisons moléculaires individuelles [
3]. Enfin, cette découverte constitue un progrès dans notre compréhension de l’inflammation, un phénomène de grande importance en médecine et en biologie et dont l’étude est à la base du travail entrepris par Cheng Zhu. Au cours de la réaction inflammatoire, les cellules endothéliales « agressées » peuvent « accrocher » les leucocytes qui circulent à une vitesse élevée (quelques mm/s). Cela réduit alors considérablement la vitesse de ces cellules (d’un facteur cinquante à cent) qui présentent un déplacement saccadé très caractéristique au contact de l’endothélium : le « roulement » (rolling). Cette interaction est suffisamment étroite et lente pour permettre à d’autres récepteurs leucocytaires de reconnaître leur ligand (par exemple une chimiokine) lorsque celui-ci est associé aux parois vasculaires. Il en résulte une activation des intégrines leucocytaires, qui contribue à l’immobilisation de ces cellules, qui pourront ensuite migrer vers les tissus périphériques. |
Le phénomène de roulement a été visualisé bien avant que l’on ne caractérise la nature des molécules d’adhésion capables de s’attacher en une fraction de milliseconde, et de résister aux forces hydrodynamiques de plusieurs centaines de piconewtons entraînant les leucocytes dans le courant sanguin. L’énigme a été résolue en 1991 grâce à des expériences réalisées in vivo par U.H. Von Adrian et al. [
4] et in vitro par M.B. Lawrence et T.A. Springer [
5] : les cellules endothéliales activées expriment des molécules de E- et P-sélectine qui interagissent avec un ligand leucocytaire tel que PSGL-1 (P selectin glycosylated ligand). Au cours du roulement, des liaisons se forment (à l’avant du leucocyte) et se rompent (à l’arrière) continuellement. Le processus de roulement est entièrement déterminé par les propriétés moléculaires des sélectines et de leurs ligands, puisqu’il a pu être reproduit avec des leucocytes tués avec des fixateurs [
6] et même avec des particules entièrement artificielles [
7]. Mais il subsistait un problème : ces molécules d’adhésion ne formaient pas de liaison persistant plus de quelques secondes, et pourtant, elles étaient capables de capter des cellules se déplaçant à grande vitesse et soumises à des forces hydrodynamiques élevées. Il était encore plus étonnant de constater que le roulement induit par les sélectines était d’autant plus « stable » que les forces hydrodynamiques étaient plus élevées. Comment expliquer ce comportement de molécules supposées produire des liaisons particulièrement so-lides ? Une observation ultérieure [
8] conduisait en fait à la conclusion paradoxale que le roulement induit par les sélectines était d’autant plus « stable » que les forces hydrodynamiques étaient plus élevées. M.B. Lawrence et T.A. Springer avaient immédiatement souligné l’importance des considérations cinétiques : les liaisons formées par les sélectines devaient se former et se rompre beaucoup plus rapidement que ne le font les associations de molécules d’adhésion « habituelles », en conditions statiques. La signification révolutionnaire de cette évolution conceptuelle a d’ailleurs aussitôt été soulignée par un immunologiste, Alan Williams, qui avait noté que le concept d’affinité paraissait jusqu’ici suffisant pour expliquer les fonctions des anticorps ou des molécules d’adhésion, sans nécessité de considérations cinétiques [
9]. Il ne restait plus qu’à vérifier cette hypothèse. |
Étude des molécules d’adhésion liées à des surfaces La compréhension du fonctionnement des récepteurs d’adhésion nécessitait donc de connaître leurs propriétés cinétiques et mécaniques. Il s’agissait là d’un domaine totalement nouveau : alors que les biochimistes savaient depuis longtemps mesurer les propriétés de liaison des molécules solubles (constante d’affinité, constantes d’association et de dissociation), les concepts traditionnels ne s’appliquaient absolument pas à la description des propriétés des molécules liées à des surfaces. Quelle est la durée de vie d’une liaison P-sélectine/PSGL-1 soumise à une force de traction de 50 piconewtons ? Les outils théoriques et expérimentaux aujourd’hui disponibles permettent d’aborder ces problèmes à l’échelle de la molécule individuelle. Les progrès rapides réalisés au cours de la dernière décennie résultent de la rencontre des modèles et des problèmes biologiques que nous venons d’évoquer et d’un courant plus fondamental que nous allons maintenant décrire. |
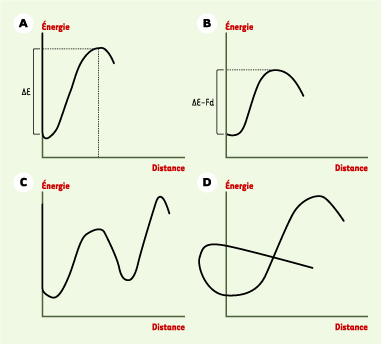
Des théoriciens avaient (presque) tout deviné … En 1978, un physicien, George Bell, proposait un cadre théorique pour déduire les propriétés d’association des molécules liées à des surfaces du comportement des formes solubles de ces mêmes molécules [
10]. Ce travail conduisait à une représentation naïve et intuitive de la rupture des liaisons moléculaires : un complexe ligand-récepteur présente des oscillations régulières du fait de l’agitation thermique. À chaque « oscillation », il existe une certaine probabilité de rupture P qui est calculée par la loi de Boltzmann (P = exp[-E/kT]), où k est la constante de Boltzmann, T la température absolue et E l’« énergie d’activation », nécessaire pour rompre la liaison). Si le complexe est soumis à une force étrangère F, l’énergie nécessaire pour rompre la liaison sera diminuée puisque la force contribue à la rupture en fournissant une énergie Fd, où d est la portée de la liaison (Figure 1). Ainsi, pour diminuer substantiellement la durée de vie d’une liaison, il faut exercer une force de l’ordre de kT/d. G. Bell avait estimé à 0,5 nm la portée d’une liaison typique : pensant à une liaison antigène-anticorps (en raison de son intérêt pour l’immunologie !), il avait remarqué que le site anticorps est assimilable à un « sillon » d’une profondeur de 0,5 nm. La force de rupture kT/d attendue est alors de l’ordre de 20 piconewtons. Cette conception peut être dans une certaine mesure justifiée sur la base de travaux effectués durant la première moitié du xxe siècle par H. Eyring [
11] et H.A. Kramers [
12] et remises en lumière à la suite des travaux que nous avons évoqués [
13]. Actuellement, la loi de variation de la constante de dissociation : koff = k0 exp (Fd/kT) est souvent appelée « loi de Bell » par les biologistes. Rappelons que koff est la fréquence de rupture de la liaison (‹). Cette rupture constitue un événement aléatoire que l’on peut comparer, en quelque sorte, à la désintégration d’une substance radioactive. (‹) m/s 2000, n° 8–9, p. 974 Quelques années plus tard, M. Dembo et al. [2] soulignaient que les lois de la thermodynamique impliquaient que l’affinité d’une liaison ligand récepteur soit diminuée par la présence d’une force séparatrice. Cependant, étant donné que la constante d’affinité (Ka) est égale au rapport de la constante cinétique d’association (kon) à la constante de dissociation (koff), la constante de dissociation n’est pas nécessairement augmentée par la force, contrairement à ce qui était considéré comme « intuitif ». Ces auteurs ont donc proposé de distinguer les « liaisons glissantes » (slip bonds) dont la constante de dissociation était – conformément à l’intuition – augmentée par une force de séparation, d’hypothétiques « liaisons accrocheuses » (catch bonds) qui présenteraient un comportement opposé. Bien sûr, le comportement du rapport kon/koff est le même pour les deux types de liaisons, puisque ce rapport est déterminé par les lois de la thermodynamique. Au cours des années suivantes, de nombreux développements méthodologiques (Figure 2) ont permis d’observer la rupture de liaisons moléculaires individuelles et de soumettre la loi de Bell à l’épreuve de l’expérience.
 | Figure 1. Fréquence de rupture d’une liaison et loi de Bell. A. Exemple simple de variation de l’énergie d’un complexe ligand-récepteur en fonction de la distance des molécules. On voit une barrière de hauteur DE à la distance d du minimum correspondant à la position d’équilibre. Le modèle de Bell revient à estimer la fréquence de dissociation de la liaison comme le produit d’une fréquence de «tentatives de sortie » par la probabilité de réussite d’un essai, calculée comme la probabilité de disposer de l’énergie DE. B. Conséquences de l’application d’une force F : l’énergie à chaque point d’abscisse x est diminuée du produit Fx, travail fourni par la force lorsque les molécules sont éloignées de la distance d’équilibre d’une valeur x. C. Courbe énergie-distance plus réaliste, présentant deux maximums correspondant à des « barrières » s’opposant à la dissociation. D. Exemple de courbe énergie-distance particulière, représentative d’une « liaison accrocheuse » : le détachement implique d’abord un rapprochement des constituants du complexe, parce que la barrière s’opposant à la séparation est plus basse que celle qui comporte un éloignement. Il faut rappeler que ces courbes représentent des coupes d’une hypersurface « énergie-position » localisée dans un espace à quatre dimensions (énergie, x, y et z). |
|
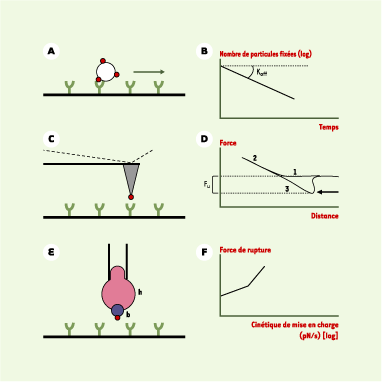
La « loi de Bell » semble d’abord vérifiée Avant même les expériences de M.B. Lawrence et T.A. Springer, quelques pionniers avaient montré que Bell avait correctement deviné l’ordre de grandeur de la résistance mécanique des liaisons ligand/récepteur. Ces expériences s’appuyaient sur des méthodologies originales telles que le «microtube mobile » construit par Harry Goldsmith [
14] ou la micromanipulation de cellules ou de vésicules adhérentes développée par Evan Evans [
15]. Cependant, la chambre à flux laminaire est sans doute l’appareil le plus accessible et le plus utilisé pour étudier la cinétique de rupture des liaisons moléculaires individuelles. Une chambre à flux laminaire (Figure 2A, B) permet d’observer le mouvement de particules ou de cellules couvertes de récepteurs au voisinage d’une surface couverte de ligands en présence d’une force hydrodynamique constante. Si cette force est suffisamment faible pour permettre à une liaison unique de maintenir la particule à l’arrêt pendant une période de temps assez longue pour être détectée, l’analyse des trajectoires permettra de visualiser directement la formation et la dissociation des liaisons. La mesure de la durée d’un nombre suffisant d’arrêts permettra alors de construire la « courbe de dissociation » des complexes et d’en déduire la constante de dissociation koff. Les expériences peuvent être répétées en variant la valeur du débit du liquide, et par conséquent de la force d’entraînement, ce qui permet de déterminer l’influence d’une force sur la constante de dissociation. Cette méthode permet de visualiser le caractère aléatoire de la dissociation des liaisons : en d’autres termes, la durée de vie des liaisons formées avec un ligand et un récepteur donnés présente des variations statistiques. La chambre à flux laminaire a été utilisée pour la première fois dans notre laboratoire (Marseille, France) pour déterminer la durée de vie de liaisons formées par l’intermédiaire de sélectines entre des cellules endothéliales et des granulocytes circulants [
16]. Un écoulement très lent avait été utilisé de manière à ce que la force hydrodynamique exercée sur les cellules soit très faible (de l’ordre de 1 pN). La durée de vie des liaisons formées était alors de l’ordre de la seconde. Ces estimations ont été confirmées par l’équipe de T. Springer [
17] : la mesure de la constante de dissociation (koff) pour plusieurs valeurs de la force hydrodynamique a conduit à une vérification de la loi de Bell, avec une constante de dissociation proche de 1 s-1 en l’absence de force et une distance d égale à 0,049 nm. Cette loi semblait également s’appliquer à de nombreux systèmes moléculaires [
18].
 | Figure 2. Étude expérimentale de la dissociation de liaisons moléculaires soumises à une force. La chambre à flux laminaire (A) permet de suivre le mouvement de particules couvertes de récepteurs au voisinage d’une surface couverte de ligands en présence d’une force hydrodynamique F. Si cette force est suffisamment faible, on peut observer des arrêts dus à la formation de liaisons isolées. La mesure des temps d’arrêts permet de construire des courbes de détachement (B) dont la pente initiale (en coordonnées semi-logarithmiques) est égale à la constante de dissociation koff. Cette expérience peut être répétée pour différentes valeurs du débit et de la force F. Des calculs approximatifs permettent d’estimer la force F exercée sur une liaison maintenant la particule à l’arrêt, en fonction de F. Un microscope de force atomique (C) permet d’approcher une pointe très fine (le rayon de courbure est inférieur à quelques dizaines de nm) couverte de récepteurs d’une surface couverte de ligands. Après une certaine période de contact, la pointe est éloignée de la surface et peut subir une traction si une liaison s’est formée. Finalement, la liaison se rompt, provoquant une remontée brutale de la pointe. Étant donné que la pointe est montée sur un ressort plat très souple (cantilever), l’apparition d’une force d’interaction se traduit par une torsion du cantilever qui est mesurée en temps réel grâce au faisceau lumineux émis par un laser (ligne pointillée) qui est réfléchi par un miroir lié à la pointe. La figure (D) représente les étapes successives de l’approche (1), suivie du contact pointe/surface engendrant une force de compression (2). Lorsque l’échantillon est retiré, la force de compression diminue, et une traction apparaît au moment de la séparation pointe/surface si une liaison s’est formée (3). La pointe remonte brutalement lors de la rupture de la liaison (flèche épaisse), ce qui permet de déterminer la force de détachement Fu. La méthode BFP (biomembrane force probe) (E) consiste à remplacer le cantilever par une vésicule (en pratique, il peut s’agir d’une hématie h) et la pointe est remplacée par une microbille b collée à la vésicule. Cette technique permet de faire varier la raideur du transducteur en contrôlant la pression d’aspiration dans la micropipette maintenant la vésicule. Par ailleurs, la position de la bille sphérique peut être déterminée avec une grande précision en analysant l’image de l’interface bille/substrat obtenue en microscopie de contraste de réflexion. On peut ainsi faire varier la cinétique de mise en charge (loading rate) sur plusieurs ordres de grandeur. La force de rupture moyenne est déterminée pour chaque condition. La courbe obtenue (F) est en principe formée de plusieurs segments dont chacun représente une « barrière » (Figure 1C) dont la position peut être déduite de la pente du segment de droite considéré. |
|
Un degré supplémentaire dans la complexité : le « paysage énergétique » Cependant qu’un ensemble d’expériences pouvaient laisser croire qu’une représentation simple et précise du comportement mécanique des liaisons avait été obtenue, des études réalisées sur d’autres modèles révélaient déjà l’existence de difficultés supplémentaires. Dès 1994, deux équipes de chercheurs [
19,
20] observaient la rupture de liaisons moléculaires individuelles au moyen d’une méthode entièrement différente : la microscopie de force atomique. Cette approche puissante repose sur un concept extrêmement simple (Figure 2C, D) : une surface couverte de ligands est approchée d’une pointe extrêmement fine (un rayon de courbure de quelques dizaines de nanomètres) recouverte de récepteurs. Après un contact susceptible d’entraîner la formation d’une liaison, une force de traction croissante est exercée sur la pointe par l’intermédiaire d’une lame flexible, ce qui permet de mesurer une force de détachement (unbinding force) au moment de la rupture de la liaison. Le modèle de l’interaction avidine/biotine avait été choisi parce qu’il s’agit de la plus forte interaction non covalente caractérisée en biologie. La force de rupture mesurée était comprise entre 160 pN [19] et 300–400 pN [20]. Ces variations pouvaient-elles s’expliquer par les incertitudes expérimentales ? Quelques années ont suffi pour qu’il apparaisse que certaines difficultés étaient explicables par la complexité des interactions moléculaires : il n’existe pas seulement deux états, « libre » et « lié », un complexe ligand-récepteur peut parcourir un continuum d’états d’énergie différente. En fait, dès 1995, la chambre à flux laminaire permettait de détecter des « complexes de transition » de durée de vie faible au cours de la formation d’une liaison antigène-anticorps [
21]. Grâce à une amélioration considérable de sa méthode [
22], qui constituait une extension de la microscopie de force atomique, Evan Evans a pu faire varier la « rapidité de mise en charge » (loading rate) des liaisons dans un intervalle extrêmement large, de 10 à 105 pN/s : l’analyse des variations de la force de rupture (Figure 2E, F) a permis d’obtenir des détails précis concernant la forme de la courbe «énergie/distance » dans les complexes avidine/biotine [
23] et PSGL-1/L-sélectine [
24] : il a ainsi été possible mettre en évidence des barrières qui, dans le dernier modèle, se situaient à des distances respectives de 0,06 nm et 0,4 nm de la position d’équilibre. En particulier, des forces supérieures à 75 pN pouvaient entraîner une dissociation des liaisons en moins de 10 millisecondes. Ce résultat était en accord avec des travaux de l’équipe de M. Lawrence qui, en combinant l’utilisation d’une vidéocaméra ultra rapide et de la chambre de flux, avait décrit des interactions très brèves entre les molécules de L-sélectine et leur ligand [
25]. La rupture d’une liaison nécessitait donc le franchissement d’une série de barrières. Chaque étape était approximativement décrite par la « loi de Bell ». |
Les « liaisons accrocheuses » Alors même que la « loi de Bell » semblait décrire correctement le comportement mécanique de la plupart des liaisons moléculaires [17], Cheng Zhu avait entrepris de mettre en évidence les mythiques « liaisons accrocheuses » en utilisant la microscopie de force atomique [
26]. Il faut souligner les difficultés de cette entreprise dans la mesure où il est très difficile d’étudier le comportement de liaisons labiles soumises à des forces faibles. En effet, la chambre à flux laminaire est probablement mieux adaptée à l’étude des interactions faibles et transitoires [
27] que la microscopie de force atomique, dont les premiers résultats ont été obtenus sur le modèle de l’interaction avidine/biotine qui est particulièrement forte. La preuve définitive de l’existence des liaisons accrocheuses a pu être obtenue par une utilisation habile de la microscopie de force atomique et des chambres à flux laminaire en conditions de faible flux hydrodynamique [1]. Ce travail montre une fois de plus combien la réalisation de progrès nécessite l’association d’une idée originale, d’une bonne maîtrise technologique et d’un acharnement suffisant. D’un point de vue théorique, ces résultats peuvent s’interpréter en attribuant aux courbes «énergie distance » un aspect extrêmement particulier (Figure 1 D). Les expériences que nous venons de décrire présentent un double intérêt : d’une part, elles montrent qu’une analyse plus approfondie des mécanismes des interactions moléculaires permet de comprendre des phénomènes expérimentaux jusque-là inexplicables [8]. D’autre part, nous abordons maintenant une étape nouvelle de la compréhension des relations entre la structure et la fonction des macromolécules : les structures cristallographiques des couples ligand-récepteur étudiés étant connues, il est possible de simuler la rupture des complexes au moyen de techniques de « dynamique moléculaire » [
28]. Il sera ainsi possible de tester expérimentalement tous les outils développés par les chimistes théoriciens pour prédire le « paysage énergétique » décrivant l’interaction d’un complexe ligand/récepteur. |
1. Marshall BT, Long M, Piper JW, Yago T, McEver RP, Zhu C. Direct observation of catch bonds involving cell adhesion molecules. Nature 2003; 423 : 190–3. 2. Dembo M, Torney DC, Saxman K, Hammer D. The reaction-limited kinetics of membrane-to-surface adhesion and detachment. Proc Roy Soc Lond 1988; 234 : 55–83. 3. Bongrand P. Ligand-receptor interactions. Rep Prog Phys 1999; 62 : 921–68. 4. Von Andrian UH, Chambers JD, McEvoy LM, Bargatze RF, Arfors KE, Butcher EC. Two-step model of leukocyte-endothelial cell interaction in inflammation : distinct roles for LECAM-1 and the leukocyte beta 2 integrins in vivo. Proc Natl Acad Sci USA 1991; 88 : 7538–42. 5. Lawrence MB, Springer TA. Leukocytes roll on a selectin at physiologic flow rates : distinction from and prerequisite for adhesion through integrins. Cell 1991; 65 : 859–73. 6. Lawrence MB, Springer TA. Neutrophils roll on E-selectin. J Immunol 1993; 151 : 6338–46. 7. Brunk DK, Goetz DJ, Hammer DA. Sialyl Lewisx/E-selectin-mediated rolling in a cell free system. Biophys J 1996; 71 : 2902–7. 8. Finger EB, Puri KD, Alon R, Lawrence MB, VonAndrian UH, Springer TA. Adhesion through L-selectin requires a threshold hydrodynamic shear. Nature 1996; 379 : 266–9. 9. Williams AF. Cellular interactions. Out of equilibrium. Nature 1991; 352 : 473–4. 10. Bell GI. Models for the specific adhesion of cells to cells. Science 1978; 200 : 618–27. 11. Eyring H. The activated complex in chemical reactions. J Chem Phys 1935; 3 : 107–15. 12. Kramers HA. Brownian motion in a field of force and the diffusion model of chemical reactions. Physica 1940; VII : 284–304. 13. Bruinsma R. Les liaisons dangereuses. Adhesion molecules do it statistically. Proc Natl Acad Sci USA 1997; 94 : 375–6. 14. Tha SP, Shuster J, Goldsmith HL. Interaction forces between red cells agglutinated by antibody. IV Time and force dependence of break-up. Biophys J 1986; 50 : 1117–26. 15. Evans E, Berk D, Leung A. Detachment of agglutinin-bonded red blood cells. I. Forces to rupture molecular-point attachments. Biophys J 1991; 59 : 838–48. 16. Kaplanski G, Farnarier C, Tissot O, et al. Granulocyte-endothelium initial adhesion. Analysis of transient binding events mediated by E-selectin in a laminar shear flow. Biophys J 1993; 64 : 1922–33. 17. Alon R, Hammer DA, Springer TA. Lifetime of P-selectin-carbohydrate bond and its response to tensile force in hydrodynamic flow. Nature 1995; 374 : 539–42. 18. Chen S, Springer TA. Selectin receptor-ligand bonds : formation limited by shear rate and dissociation governed by the Bell model. Proc Natl Acad Sci USA 2001; 98 : 950–5. 19. Florin EL, Moy VT, Gaub HE. Adhesion forces between individual ligand-receptor pairs. Science 1994; 264 : 415–7. 20. Lee GU, Kidwell DA, Colton RJ. Sensing discrete streptavidin-biotin interactions with atomic force microscopy. Langmuir 1994; 10 : 354–7. 21. Pierres A, Benoliel AM, Bongrand P. Measuring the lifetime of bonds made between surface-linked molecules. J Biol Chem 1995; 270 : 26586–92. 22. Evans E, Merkel R, Ritchie K, Tha S, Zilker A. Picoforce method to probe submicroscopic actions in biomembrane adhesion. In : Bongrand P, Claesson M, Curtis ASG, eds. Studying cell adhesion. Heidelberg : Springer Verlag, 1994 : 125–39. 23. Merkel R, Nassoy P, Leung A, Ritchie K, Evans E. Energy landscapes of receptor-ligand bonds explored with dynamic force spectroscopy. Nature 1999; 397 :50–3. 24. Evans E, Leung A, Hammer D, Simon S. Chemically distinct transition states govern rapid dissociation of single L-selectin bonds under force. Proc Natl Acad Sci USA 2001; 98 : 3784–9. 25. Smith MJ, Berg EL, Lawrence MB. A direct comparison of selectin-mediated transient, adhesive events using high temporal resolution. Biophys J 1999; 77 : 3371–83. 26. Marshall B, McEver RP, Zhu C. Kinetic rates and their force dependence of the P-selectin/PSGL-1 interaction measured by atomic force microscopy. BED (Bioengeneering Conference ASME) 2001; 50 : 775–6. 27. Pierres A, Benoliel AM, Bongrand P, van der Merwe PA. Determination of the lifetime and force dependence of interactions of single bonds between surface-attached CD2 and CD48 adhesion molecules. Proc Natl Acad Sci USA 1996; 93 : 15114–8. 28. Izrailev S, Stepaniants S, Balsera M, Oono Y, Schulten K. Molecular dynamics study of unbinding of the avidin-biotin complex. Biophys J 1997; 72 : 1568–81. |