Depuis la mise en évidence du rôle des immunoglobulines (anticorps) dans la réponse immune, les scientifiques ont cherché à expliquer comment un répertoire limité d’anticorps peut interagir avec un nombre quasi infini d’antigènes. En effet, les sites antigéniques reconnus par les anticorps peuvent être portés par toutes les molécules du monde vivant: sucres, lipides, acides nucléiques, petites molécules chimiques (haptènes), peptides, protéines; il peut aussi s’agir de structures moléculaires complexes telles que des protéines glycosylées. Cette diversité de reconnaissance apparaît plus grande encore par le fait qu’une même macromolécule peut exposer plusieurs sites antigéniques à sa surface, et que des anticorps sont également capables de reconnaître des composés organiques synthétiques, ainsi que des molécules du soi (dans les maladies auto-immunes). Les travaux de S.Tonegawa [ 1], qui lui valurent de recevoir le Prix Nobel de Médecine en 1987, constituèrent une avancée décisive dans la compréhension de l’origine de la diversité du répertoire des anticorps. Au cours du processus de sélection clonale, la combinaison d’un nombre fini de gènes, associée à l’imprécision de la jonction de ces différents gènes, à la combinatoire chaîne lourde-chaîne légère, et enfin aux mutations somatiques ponctuelles disséminées le long des régions variables des chaînes lourdes et légères, constituait jusqu’à maintenant l’ensemble des mécanismes à l’origine de la diversité du répertoire des immunoglobulines.
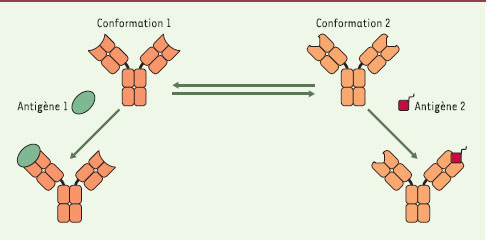
Toutefois, des travaux récents établissent qu’un niveau supplémentaire de variabilité du répertoire des anticorps existe! En effet, les résultats publiés dans la revue Science [ 2, 3] par D. S. Tawfik et al. démontrent qu’une même molécule d’anticorps peut pré-exister sous plusieurs conformations structurales, chacune étant susceptible de reconnaître un antigène spécifique (Figure 1).
| Figure 1.
Diversité structurale d’une molécule d’anticorps. La même molécule d’anticorps existe sous deux conformations structurales capables de lier deux antigènes structuralement distincts [3]. Les conformations 1 et 2 sont notées Ab1 et Ab2. dans le texte. |
Un tel scénario avait été envisagé par Linus Pauling dès 1940 [ 4], et repris plus récemment sur la base de données expérimentales sur la cinétique de formation de complexes anticorps-antigène [ 5, 6]. Ces derniers travaux suggéraient qu’un équilibre puisse exister entre plusieurs (au moins deux) conformations structurales (isomères) d’un anticorps unique, et que le ligand principal de cet anticorps reconnaissait préférentiellement un de ces isomères!
L’importance des travaux publiés par D.S. Tawfik et al. tient en ce qu’ils apportent des preuves expérimentales directes de l’existence d’un équilibre entre plusieurs isoformes de conformation différente d’un même anticorps, chaque isomère étant capable d’interagir spécifiquement avec des antigènes aussi différents qu’une petite molécule chimique (haptène) et une protéine globulaire. Cette démonstration repose sur l’analyse structurale par diffraction de rayons X de cristaux de l’anticorps SPE7, un antihaptène initialement, seul ou sous la forme de complexes anticorps-antigène. Le premier résultat de cette étude structurale est que l’anticorps non complexé cristallise sous deux conformations différentes notées Ab1 et Ab2 (Figure1): chaque formation présente une organisation spatiale tout à fait différente au niveau de régions importantes constituant son site de liaison ou paratope:les boucles hypervariables 3 des domaines variables des chaînes lourde et légère1. Ces différences aboutissent à deux types de paratopes:un paratope assez plat et régulier dans le cas d’Ab1 (une organisation typique d’anticorps qui lient des peptides ou des protéines), alors qu’Ab2 présente un paratope en forme d’entonnoir se terminant par une cavité profonde, aspect caractéristique d’un anticorps spécifique d’haptène. L’obtention des structures cristallographiques de l’anticorps SPE7 complexé à trois haptènes différents, à permis aux auteurs d’établir: (1) que, comme le laissait prévoir la structure de son paratope, c’est bien l’isomère Ab2 qui a été sélectionné pour lier de tels ligands; (2) que cet isomère particulier est par ailleurs capable de lier efficacement différents haptènes; et (3) que cette capacité de liaison fait intervenir des réarrangements structuraux induits (induced-fit) qui permettent à l’anticorps SEP7 de s’adapter aux caractéristiques structurales de chacun des trois haptènes qu’il reconnaît. Parallèlement, la structure d’un complexe entre SEP7 et une protéine (la thiorédoxine) était résolue, démontrant que c’était cette fois-ci l’isomère Ab1 qui avait été sélectionné comme la structure de paratope la mieux adaptée pour interagir efficacement avec l’épitope antigénique reconnu à la surface de la thiorédoxine.
Ce sont donc au total six structures différentes de l’anticorps SEP7 qui ont été caractérisées lors de cette étude! Une telle prodigalité d’organisations tridimensionnelles, à partir d’une séquence unique d’immunoglobuline, n’était certes pas envisagée, et appelle donc plusieurs commentaires. Tout d’abord, ces résultats établissent, et ce bien audelà de ce que l’on envisageait jusqu’à maintenant, le dynamisme structural des anticorps, ce qui pose la question de la diversité structurale d’un anticorps. Se limite-t-elle à un équilibre entre deux isomères ? Il est probable que non, car si les auteurs de ce travail ont expérimentalement établi l’existence de deux isomères conformationnels de l’anticorps SEP7, ils n’excluent pas que d’autres puissent exister. Si c’est le cas pour l’anticorps SEP7, en est-il de même pour l’ensemble des anticorps, et ce quelle que soit leur spécificité initiale: antihaptènes, anti-peptides, anti-protéine, etc. ? Quoi qu’il en soit, ces travaux identifient clairement un niveau supplémentaire de diversité du répertoire des immunoglobulines fondé sur la co-existence de plusieurs structures protéiques différentes et stables d’un point de vue énergétique. Une telle situation n’est pas sans rappeler les équilibres qui caractérisent les différents états des récepteurs allostériques qui sont reconnus et stabilisés par divers effecteurs physiologiques [ 7]. Après les récepteurs et les enzymes allostériques et la protéine prion, les anticorps constituent un nouvel exemple de la polyvalence structurale qui peut être associée à une séquence protéique unique !
Ces données expérimentales modifient aussi notre vision des évènements structuraux et énergétiques qui régissent la formation de complexes stables anticorps- antigènes. En effet, ces résultats suggèrent l’existence d’un équilibre entre plusieurs isoformes d’un même anticorps (2 au minimum), qui auraient chacune une propension particulière à lier des antigènes différents, la stabilisation des pré-complexes étant finalement assurée par des ajustements structuraux locaux et limités. La notion de spécificité des anticorps est également mise à mal ! En effet, si les exemples d’anticorps reconnaissant des motifs multiples ne manquent pas, il s’agissait jusqu’à maintenant d’une reconnaissance croisée entre des molécules qui étaient apparentées d’un point de vue structural. Ce que nous démontrent de manière inattendue D.S. Tawfik et al. c’est la capacité d’une molécule unique d’anticorps à interagir avec des antigènes totalement différents via des sites distincts. Une telle démonstration pose la question de l’avantage d’un tel potentiel, et de sa signification biologique ! Les deux sites de liaison aux antigènes que présente une molécule d’immunoglobuline sont-ils équivalents ? Peuventils reconnaître des antigènes différents et/ou deux épitopes distincts proches dans l’espace à la surface d’une molécule antigénique ? Fonctionnent-ils de manière coopérative ? S’agit-il là d’un moyen pour le système immunitaire d’assurer plus efficacement la neutralisation et l’élimination des molécules reconnues ?
Comme bien souvent, ces résultats expérimentaux posent plus de questions qu’ils n’apportent de réponses. Mais ce qui est certain, c’est que nous sommes en face d’une découverte importante, dont nous ne faisons que commencer à entrevoir les conséquences !