La méthylation des histones, quasiment inconnue il y a encore un an, est maintenant devenue la star des congrès dans le domaine de la régulation transcriptionnelle. Nous avions écrit une synthèse à ce propos il y a maintenant six mois [1], mais un certain nombre de développements récents méritent que l’on y revienne.
Les histones peuvent être méthy-ées sur des résidus lysines (K4, 9 et 27 de H3 et K20 de H4), mais aussi, comme cela vient tout juste d’être montré, sur des arginines [2]. Nous ne discuterons ici que la méthylation des histones sur les lysines.
La méthylation de la lysine 9 de l’histone H3 joue un rôle majeur dans le silencing (terme qui désigne la répression trans-criptionnelle qui s’exerce sur un large domaine chromatinien, en relation avec une structure particulière de la chromatine). Les histone méthyltransférases de la famille de Suv39H1, qui sont strictement spécifiques de la K9 de l’histone H3, sont en effet requises, chez la drosophile ou S. pombe, pour le silencing de gènes positionnés au voisinage d’un centromère (c’est-à-dire faisant partie de l’hétérochromatine péricentrique) [3]. De plus, on constate que la lysine 9 de l’histone H3 est méthylée dans des zones de chromatine soumises à un silencing : c’est le cas pour l’hétérochromatine péricentrique et le locus silencieux de type sexuel chez S. pombe [4], les centromères des chromosomes polytènes chez la drosophile [5], ou encore la région silencieuse adjacente au locus de la ß-globine chez le poulet [6]. Si on constate une corrélation entre la présence de cette méthyation et la structure de la chromatine, il est cependant très difficile de montrer qu’elle est indispensable, les mutants d’histones étant impossibles à réaliser dans les organismes eucaryotes supérieurs.
Selon l’hypothèse du « code des histones », la méthylation, tout comme d’ailleurs les autres modifications, modulerait la liaison de protéines avec les nucléosomes [7]. Ainsi, l’histone H3 méthylée sur la K9 est reconnue par les protéines de la famille HP1 (heterochromatin protein 1), protéines structurales de l’hétérochromatine.
Les méthyltransférases de la famille de Suv39H1 méthylent l’histone H3, permettant ainsi le recrutement des protéines de la famille HP1 et la répression transcriptionnelle qui lui est associée [3].
La méthylation de la lysine 9 de l’histone H3 joue donc principalement un rôle négatif sur la transcription. En est-il de même pour la méthylation des autres lysines ? De plus en plus de données suggèrent que la méthylation de K4 de l’histone H3 est au contraire associée à l’activation de la transcription. En effet, les méthyl-transférases capables de méthyler la lysine 4 de l’histone H3 sont généralement des activateurs transcriptionnels, comme Ash1 ou SET1 [2], ou bien sont physiquement associées à des co-activateurs [8]. Parailleurs, l’histone H3 méthylée sur la ysine 4 est enrichie dans les zones trans-criptionnellement actives [6 ,9]. La conséquence moléculaire de la méthylation de la lysine 4 n’est pas encore connue. L’hypothèse la plus probable serait que la méthylation de cette lysine favoriserait le recrutement d’un facteur activateur, ou au contraire inhiberait l’interaction d’un complexe répresseur.
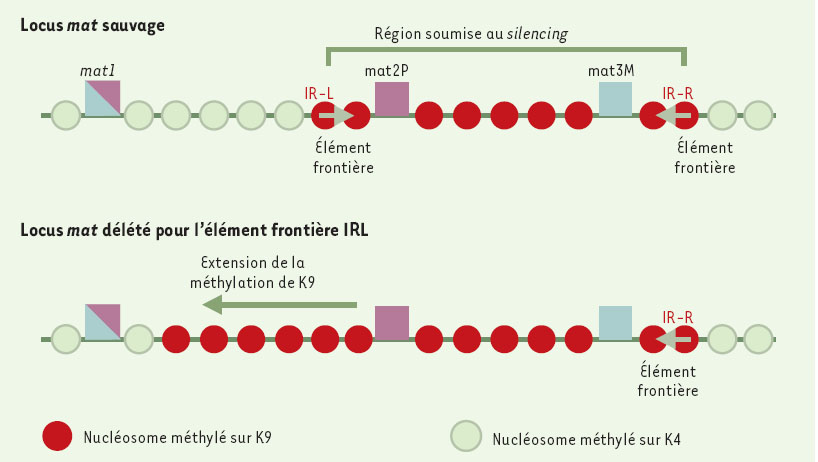
La méthylation des histones sur les ysines peut donc avoir des conséquences fonctionnelles diverses pour la chromatine. La méthylation présente toutefois une caractéristique particulière : sa longue durée de vie. En effet, bien qu’il paraisse maintenant de plus en plus probable qu’il existe un moyen de démé-thyler les histones (par réelle déméthy-ation ou par remplacement des histones méthylées), il est clair que c’est une modification beaucoup moins dynamique que l’acétylation ou la phospho-rylation. C’est une des raisons qui laissent penser que la méthylation des histones serait un moyen pour la cellule de « marquer » des domaines étendus de chromatine. En accord avec cette idée,’hétérochromatine centromérique, qui est « marquée » par la méthylation de la ysine 9 de l’histone H3, se conserve au cours du cycle mitotique de manière indépendante de la séquence primaire d’ADN [3]. La méthylation de la lysine 9 correspondrait ainsi à des zones silencieuses, alors que, au contraire, la méthylation de la lysine 4 définirait des régions permissives à la transcription. Cette hypothèse a été confirmée dans le cas du locus de type sexuel dans S. pombe [9] (Figure 1) ou du locus de la ß-globine chez le poulet [6]. Le rôle moléculaire des éléments barrières ou des isolateurs (insulators) qui fonctionnent comme frontières entre deux zones, serait d’empêcher la propagation de la méthylation de la lysine 9 [6,9] (Figure 1).
| Figure 1. Représentation schématique du locus de type sexuel (locus mat ) de la levure S. pombe. Les copies silencieuses mat2P (carré rouge) et mat3M (carré bleu) ainsi que le gène actif matl (qui est soit P, soit M, suivant le type sexuel de la levure) (carré moitié bleu, moitié rouge) sont indiqués. Les nucléosomes méthylés sur la lysine 9 ou sur la lysine 4 de l’histone H3 sont schématisés en rouge et en vert, respectivement. Notez que les deux modifications sont exclusives, et sont séparées, dans un locus sauvage (haut de la figure), par deux répétitions inversées (IR-L (inverted repeat left) et IR-R (inverted repeat right), flèches vertes), qui fonctionneraient donc comme des éléments frontières. La région soumise au silencing (région dans laquelle l’intégration d’un gène hétérologue conduit à la répression de ce gène) est indiquée : elle correspond exactement au domaine où les histones H3 sont méthylées sur la lysine 9. Le bas de la figure représente le même locus délété pour l’IR-L : noter l’extension de la zone où la lysine 9 est méthylée (indiquée par la flèche), ce qui confirme le rôle d’élément barrière pour l’IR-L. Le rôle de cet élément barrière serait ainsi d’empêcher la propagation de la méthylation de la lysine 9 de l’histone H3. Ces résultats sont tirés de [9]. |
Des études récentes indiquent cependant que la méthylation des his-tones pourrait être également impliquée dans la régulation de promoteurs spécifiques [10,11]. Dans ce cas-là, la modification ne concerne pas un domaine étendu, mais seulement un nombre limité de nucléosomes [11]. Ces résultats impliquent l’existence de mécanismes, encore inconnus, empêchant l’extension de la « marque » à tout le domaine chromatinien. Si ces résultats sont confirmés, alors la méthylation des histones pourrait, tout comme l’acétylation, être un intervenant majeur dans la régulation trans-criptionnelle de promoteurs spécifiques.
Quoi qu’il en soit, le consensus actuel confère à la méthylation des histones un rôle de «marque» de larges domaines chro-matiniens. Cette conclusion appelle un certain nombre de questions : que marquent les méthyla-tions de la K20 de l’histone H4 et de la K27 de l’histone H3 ? Quelles sont les relations fonctionnelles entre la méthylation des histones et les autres marques de la chromatine, comme l’acétylation des histones ou la méthylation de l’ADN ?