Le séquençage complet du génome du nématode a identifié 19282 gènes [1], mais la fonction de la grande majorité de ces gènes reste toujours inconnue. Six pour cent des gènes seulement ont été analysés par des études de génétique classique ou de biochimie, et 53 % d’entre eux présentent des homologies avec des gènes d’autres organismes. Au cours de ces deux dernières années, l’utilisation de puces à ADN contenant une partie ou la quasi-totalité du génome de C. elegans a entraîné une croissance exponentielle des expériences d’analyse d’expression de gènes à grande échelle. L’équipe de Stuart Kim à Stanford, en suivant le type d’approche qui avait été utilisé avec succès chez la levure [3], a analysé l’ensemble des résultats obtenus avec des puces à ADN pour identifier des corrélats d’expression [2].
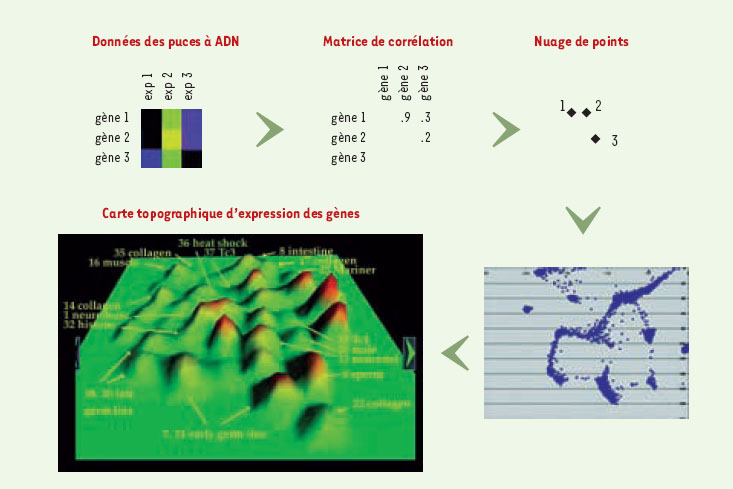
Kim et al. ont utilisé les résultats de 553 expériences, en provenance de 30 laboratoires différents, impliquant un total de 17 817 gènes, soit 94 % du génome de C.elegans (Tableau 1). Ces différentes expériences comparent les profils d’expression obtenus à partir de vers sauvages et de vers mutants (pour la voie de signalisation Ras, pour la détermination du sexe,..), ou ceux issus de l’analyse de vers à différents stades du développement ou encore élevés dans des conditions différentes (stress, irradiation…). Tous les gènes présents sur les puces, à l’exception d’une toute petite proportion (<1%), sont exprimés dans au moins une des conditions expérimentales. En combinant toutes ces expériences, les auteurs obtiennent ainsi une matrice où chaque ligne représente un gène et chaque colonne une expérience indépendante (Figure 1). Dans la matrice, le niveau relatif d’expression de chaque gène pour chaque expérience est indiqué. Un coefficient de corrélation est ensuite calculé de façon à définir le degré de liaison d’expression entre toutes les paires de gènes, et ce indépendamment des conditions expérimentales de départ. Chaque gène est ensuite placé sur une carte en deux dimensions, et sa position y est définie en fonction de la corrélation qui existe entre son profil d’expression et celui des autres gènes. Les distances entre gènes sont ainsi proportionnelles au degré de similitude qui existe entre leurs profils d’expression. Les gènes dont l’expression varie de façon similaire dans les différentes conditions expérimentales sont ainsi groupés sur la carte. Kim et al. ont ajouté un troisième axe traduisant la quantité de gènes dans chaque groupe. Ces groupes apparaissent alors en relief et les auteurs les comparent à des montagnes. Quarante-quatre montagnes ont été identifiées, numérotées de la plus grande, contenant le plus de gènes, à la plus petite. La quasi-totalité des gènes (17661 gènes, soit 93 %) peuvent être localisés sur la carte dans l’un ou l’autre des groupes d’expression. L’ensemble de ces données sont en libre accès (http://www.sciencemag.org/feature/ data/kim1061603/c._elegans_topomap.html).
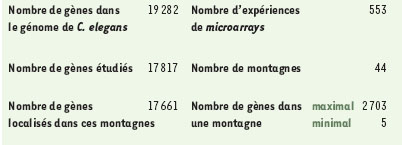
| Tableau 1. Étude du transcriptome de C. elegans. |
| Figure 1. Construction d’une carte topographique d’expression de gènes. Dans la matrice d’expression, le jaune représente l’augmentation relative de l’expression d’un gène et le bleu une diminution. Trois gènes et trois expériences sont données en exemple. Les données d’expression sont utilisées pour calculer un indice de corrélation entre chaque paire de gène. Les gènes avec le plus fort taux de corrélation sont ensuite utilisés pour construire un nuage de points en 2D. Ce graphe est alors converti en une carte topographique d’expression de gènes représentant les corrélations en 3D, où l’altitude correspond à la densité des gènes. |
Les auteurs ont analysé les gènes dans chaque montagne à la recherche de groupes de gènes associés à une propriété biologique. Ils ont ainsi identifié des montagnes correspondant à une expression spécifique d’un tissu donné (muscle, neurones, intestin…) bien qu’aucune des expériences de puces n’ait ciblé délibérément ces tissus, et des montagnes correspondant à une expression spécifique d’une fonction cellulaire. Par ailleurs, ils ont analysé la répartition de 56 groupes de gènes connus pour être impliqués dans un même processus : 46 de ces groupes sont enrichis dans une ou deux montagnes différentes. Cette analyse permet d’attribuer une importance physiologique à 30 des 44 montagnes.
Trente-neuf gènes ont une expression privilégiée dans es muscles. Ces derniers enrichissent les “montagnes” 1 et 16. La montagne 1 contient les gènes musculaires codant principalement pour des récepteurs, des protéines extracellulaires ainsi que des protéines associées aux récepteurs, comme egl-19 qui code pour un cana calcium, egl-30 qui code pour une sous-unité OC de protéine G ou unc-52 qui code pour un composant de la membrane basale. Les gènes codant pour les composants des fibres musculaires, comme la myosine, la paramyosine ou la troponine sont, eux, inclus dans la “montagne” 16. Ceci montre le niveau de précision de cette approche qui permet de distinguer, au sein d’un même tissu, les gènes associés à des fonctions différentes. Un autre point intéressant est que, dans cette même “montagne” 1, on trouve également des gènes neuronaux codant pour des récepteurs ou des protéines associées à ces récepteurs. Cette observation suggère aux auteurs que les gènes de la “montagne” 1 pourraient avoir des fonctions synaptiques au niveau de la jonction neuromusculaire.
Parmi les gènes groupés par analogie de fonction, ceux qui codent pour les protéines de choc thermique sont intéressants : 7 des 10 gènes composant la “montagne” 36 codent pour des protéines de ce type. Les 3 autres gènes n’avaient pas de fonction connue dans la réponse au choc thermique. Les résultats de cette analyse ont poussé les auteurs à analyser par puce à ADN la réponse de C. elegans au choc thermique, ce qui a permis de démontrer que ces trois gènes étaient effectivement réglés dans ces conditions.
Le bien-fondé de la corrélation de fonction ou de localisation tissulaire suggérée par l’analyse de la carte d’expression a été confirmé pour plusieurs groupes de gènes des différentes “montagnes”. Un atout supplémentaire de la carte d’expression est son aspect dynamique, puisque sa résolution s’affine à chaque inclusion de nouvelles données. Il apparaît donc qu’une telle carte d’expression peut permettre de prédire, dans une certaine mesure, la fonction d’un gène ou d’un groupe de gènes inconnus. Bien sûr, cette fonction hypothétique devra être confirmée par des données expérimentales. Mais ajoutée aux travaux systématiques d’obtention de mutants par le processus d’interférence ARN [4] et d’étude des interactions protéines-protéines par la technique du double hybride [5], l’analyse de ces données d’expression globale permettra d’avancer rapidement dans la compréhension de la fonction d’un gène ou d’un groupe de gènes et, à plus long terme, dans notre connaissance globale des processus biologiques d’un organisme entier.