| |
| Med Sci (Paris). 2002 April; 18(4): 439–447. Published online 2002 April 15. doi: 10.1051/medsci/2002184439.Systèmes de sécrétion des protéines de type IV et virulence bactérienne Vincent Foulongne, Sylvie Michaux-Charachon, David O’Callaghan, and Michel Ramuz* Inserm U. 431, Faculté de médecine, avenue Kennedy, 30900 Nîmes, France |
Dans de nombreux cas de bactéries pathogènes à Gram négatif, des protéines nécessaires à la virulence sont exportées dans l’environnement, qu’il soit ou non la cellule eucaryote elle-même. Pour pouvoir traverser la paroi bactérienne, ces protéines doivent franchir les deux obstacles que sont la membrane interne et la membrane externe, toutes deux hydrophobes et donc imperméables aux composés hydrophiles. Pour ce faire, elles ont besoin de systèmes de sécrétion. Quatre systèmes de sécrétion de protéines sont actuellement connus chez les bactéries à Gram négatif: les types I à IV [1]. Les types I sont responsables de la sécrétion d’enzymes extracellulaires par les bactéries à Gram négatif, comme la sécrétion d’hémolysine α chez Escherichia coli, de l’adé-nylate cyclase chez Bordetella pertussis, de protéases chez Pseudomonas aerugi-nosa [2]. La sécrétion par des systèmes de type II met en jeu un système particulier, dit système sec, comprenant différentes protéines, dont une ATPase, une protéine chaperon et une peptidase. Les protéines exportées par ces systèmes possèdent une séquence d’acides aminés hydrophobes, dite « séquence signal », qui est nécessaire à leur exportation mais sera éliminée par la peptidase dans l’espace périplasmique. Ces systèmes sont responsables de la sécrétion d’enzymes pectiques et de cellulases par Erwi-nia, d’élastase, de phospholipase C et d’exotoxine A par P . aeruginosa, de pullanase par Klebsiella oxytoca [3]. Les deux types suivants sont utilisés lors de la sécrétion de protéines au cours d’un contact avec les cellules. Le type III est certainement le mécanisme de sécrétion le plus étudié [4]. Il met en jeu une vingtaine de protéines, dont beaucoup sont des homologues des composants de la biosynthèse des flagelles. Ce système permet la sécrétion de protéines responsables de l’adhérence de la bactérie à la surface cellulaire (E. coli entéropathogènes), de l’entrée de la bactérie dans des cellules non phagocytaires (Salmonella typhimurium, Shigella spp., Yersinia spp.) ou au contraire de la résistance à la phagocytose par les macrophages (P. aeruginosa, Yersinia spp.), d’induction de l’apoptose des macrophages [5]. Les protéines bactériennes sécrétées interagissent avec des protéines cellulaires (protéines G, tyrosine kinases, adénylate cyclase, etc.), conduisant, entre autres effets, à la réorganisation de l’actine du cytosquelette et à la phagocytose, ou, au contraire, à la dépolymérisation de l’actine et à l’inhibition de la phagocytose, ou encore à l’apoptose. L’activité des systèmes de sécrétion de type III est réglée à la fois aux niveaux transcriptionnel et post-traductionnel. Le dernier système connu est la voie de sécrétion de type IV. Ce système permet la sécrétion de complexes nucléoprotéiques ou protéiques à travers la membrane des bactéries à Gram négatif [6]. Les plus connus jouent un rôle majeur dans l’échange de plasmides au cours de la conjugaison bactérienne, par exemple le système tra chez E. coli. Plus récemment, il a été démontré que ces systèmes de type IV jouent un rôle important dans la virulence de certaines bactéries, ce qui fait l’objet de cet article. Il est cependant possible que d’autres systèmes de sécrétion existent chez les bactéries, comme les systèmes dits « autotransporteurs » de Neisseria gonorrhoeæ ou d’Helicobacter pylori. |
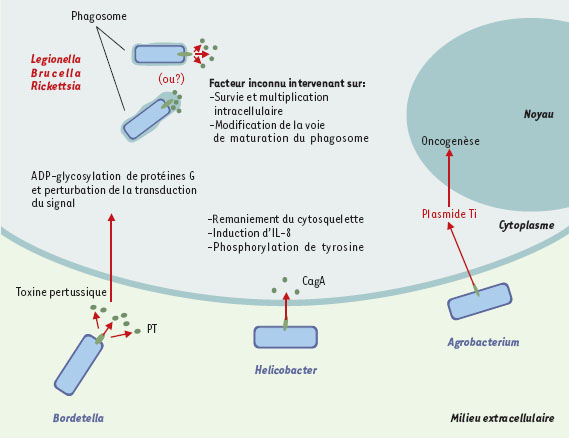
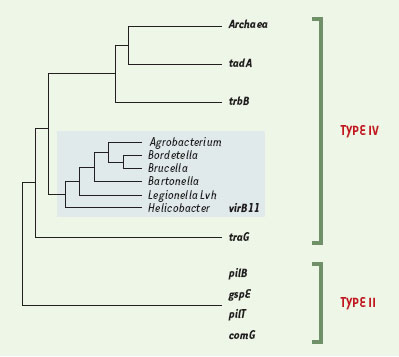
Les systèmes de sécrétion de protéines de type IV chez les bactéries pathogènes Le plus anciennement décrit et le mieux connu des systèmes de type IV impliqués dans la virulence est celui d’Agrobacterium tumefaciens, une bactérie pathogène pour les plantes (→). Récemment, de tels systèmes ont été retrouvés chez des bactéries pathogènes pour l’homme et les animaux (Figure 1, Tableau I) [7]. (→) m/s 2001, n°8/9, p. 856 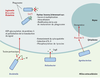 | Figure 1. Les systèmes de type IV chez les bactéries pathogènes. Les systèmes de sécrétion de type IV sont présents chez quelques bactéries pathogènes avec des comportements différents. Chez Agrobacterium et Helicobacter, la sécrétion de la molécule effectrice dans la cellule eucaryote receveuse nécessite un mécanisme dépendant du contact. Bordetella sécrète, dans le milieu extérieur, sa toxine (PT) par le système Ptl. Dans le cas des bactéries intracellulaires (Legionella, Rickettsia et Brucella), le système de sécrétion de type IV permet la survie de la bactérie dans la vacuole de phagocytose. La nature des protéines effectrices est encore inconnue chez ces bactéries, mais on peut supposer qu’elles interviennent sur le trafic intracellulaire de la vacuole d’endocytose, soit en modifiant la surface du phagosome, soit par interaction avec les cascades de signalisation des voies d’endocytose, pour diriger la bactérie vers une niche intracellulaire spécifique, favorable à sa survie. |
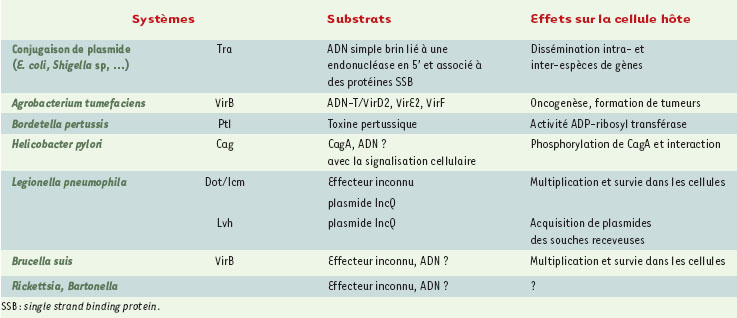
 | Tableau I. Les substrats des systèmes de type IV. |
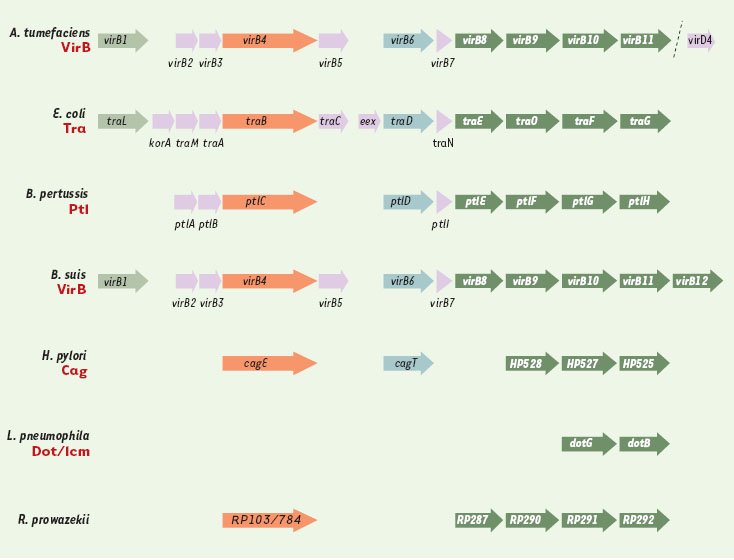
Agrobacterium tumefaciens L’infection d’une plante par A. tumefaciens se traduit par l’apparition de tumeurs du collet, appelées crown-galls. La bactérie infecte d’abord les cellules de la plante, puis lui transfère un plasmide, qui va s’intégrer dans son génome. Ce plasmide, nommé p Ti (pour tumor-inducing), contient à la fois des gènes responsables de son transfert et des gènes de synthèse de facteurs de croissance, déréglant la multiplication des cellules de la plante et conduisant à la formation de la tumeur [ 8]. Le transfert du plasmide se fait par l’intermédiaire d’un complexe protéique situé dans la paroi de la bactérie [ 9]. Ce complexe protéique, VirB, met en jeu onze protéines codées par onze gènes formant la région virB du plasmide et réunis en opéron (Figure 2). Plusieurs macromolécules sont exportées par VirB sous forme d’un complexe nucléoprotéique ( voir cidessous). Les protéines VirB de A. tumefaciens présentent une homologie importante avec les protéines d’ E. coli impliquées dans le transfert de plasmides, tels ceux des groupes IncP (protéines Tra) ou IncW (protéines Trw), au cours de la conjugaison. Enfin, la présence d’un deuxième système de type IV sur le plasmide Ti vient d’être démontrée.  | Figure 2. Organisation des systèmes de sécrétion de type IV. Alignement de différents systèmes de sécrétion illustrant la conservation de l’organisation entre les systèmes conjugatifs ou le système de transfert de l’ADN-T de Agrobacterium tumefaciens et les systèmes de sécrétion impliqués dans la virulence de bactéries pathogènes. |
Bordetella pertussis Le deuxième système de sécrétion de type IV participant à la virulence d’une bactérie a été ultérieurement décrit chez B. pertussis, responsable de la coqueluche [ 10]. Sa virulence est en grande partie déterminée par la synthèse et la sécrétion d’un complexe protéique, dit toxine pertussique. Cette toxine est formée de cinq sous-unités protéiques, codées par cinq gènes juxtaposés formant le locus ptx. Deux autres groupes de gènes, contigus à ptx, sont situés dans la même région chromosomique: (1) neuf gènes, appelés ptl (pour pertussis toxin liberation) (Figure 2); (2) deux gènes codant pour un système de régulation à deux composants, bvgA et bvgS. ptx et ptl forment un opéron, réglé par le système bvgA-bvgS suivant les caractéristiques du milieu (température, ions sulfates, acide nicotinique) [ 12]. Le complexe protéique Ptl, qui a une structure proche de celle de VirB, correspond à un système de sécrétion de type IV et permet la sécrétion de la toxine pentamérique [ 11]. Cette toxine, libérée dans le milieu extracellulaire, interagit avec des récepteurs glycoprotéiques des cellules cibles. Par ADP-glycosylation des protéines G, elle interfère avec la transduction du signal. Helicobacter pylori H. pylori est retrouvé chez 30 % à 100 % des adultes suivant la région géographique considérée (→). Cette bactérie a été incriminée dans l’apparition d’ulcères gastro-duodénaux, d’adénomes et de lymphomes gastriques, touchant à peu près 7 millions de personnes dans le monde et causant plusieurs centaines de milliers de morts. De nombreux facteurs de virulence ont été découverts chez H. pylori, tels qu’une uréase très active qui pourrait lui permettre de lutter contre l’acidité gastrique et une cytotoxine, VacA, provoquant la vacuolisation des cellules cibles. Cependant, l’étude de la virulence de divers isolats de H. pylori a montré qu’elle dépend aussi de la présence ou de l’absence dans le chromosome d’une région d’environ 40 kb appelée cag et contenant 31 gènes [ 13]. Six de ces gènes sont homologues avec les gènes du système de transport de type IV décrits ci-dessus (Figure 2). L’étude de mutants montre que cag induit plusieurs changements dans la cellule infectée, comme la sécrétion d’interleukine-8 (IL-8), le remodelage de la surface cellulaire avec formation de piédestaux, l’activation de facteurs de transcription, l’expression de proto-oncogènes [ 14]. Récemment, il a été démontré que le système de type IV permet l’exportation dans le cytoplasme de la cellule gastrique de CagA, une protéine de 141 kDa dont le gène est situé dans la région cag [ 15]. Après phosphorylation par les tyrosine kinases cellulaires, CagA participe à la formation du piédestal qui sert à l’adhérence de la bactérie [ 16, 17]. Enfin, il est à noter que les isolats de patients contiennent toujours un mélange de souches cag+ et de souches cag-, avec une fluctuation dans le temps du rapport entre les deux populations. Suivant le niveau de chacune, l’infection pourrait ou non s’accompagner de signes cliniques. H. pylori possède par ailleurs un locus impliqué dans la compétence naturelle de la bactérie, appelé locus comB, qui présente de fortes homologies avec des composants des systèmes de sécrétion de type IV, et dont le rôle semble limité au transport d’ADN lors de la transformation [ 18]. (→) m/s 2001, n°6/7, p. 712 Legionella pneumophila L. pneumophila est une bactérie ubiquitaire normalement présente dans l’eau où elle parasite diverses espèces d’amibes. L’inhalation d’aérosols contenant cette bactérie provoque chez l’homme la maladie des légionnaires, se traduisant par une pneumopathie souvent grave. L. pneumophila, en pénétrant dans le macrophage, inhibe l’acidification du phagosome et la fusion de celui-ci avec les lysosomes, lui permettant d’échapper aux mécanismes bactéricides du phagocyte. Le compartiment où elle se multiplie, appelé « phagosome réplicatif », est entouré de réticulum endoplasmique rugueux. Certains auteurs l’ont interprété comme un autophagosome. L. pneumophila paraît donc perturber le trafic membranaire intracellulaire. L’étude de mutants incapables de se multiplier dans les phagocytes a permis de localiser deux régions du chromosome impliquées dans la virulence, d’une vingtaine de kilobases chacune [ 19]. Ces gènes, au nombre d’une trentaine, ont reçu le nom de icm (pour intracellular multiplication) ou dot (pour defective for organelle trafficking). Le système dot/icm présente une remarquable homologie avec les composants du système conjugatif Tra du plasmide ColIb-P9 (différent du plasmide de virulence de cette bactérie) de Shigella flexneri. De plus, deux protéines (DotG et DotB) sont des homologues des protéines VirB10 et VirB11 de A. tumefaciens (Figure 2). Les mutants dotG et dotB ne peuvent plus se multiplier dans la cellule, ni inhiber l’acidification du phagosome et la fusion avec les lysosomes. Un système de sécrétion de type IV semble donc exister chez L. pneumophila, jouant un rôle dans la perturbation du trafic intracellulaire des membranes, et ceci dès les premières minutes suivant l’internalisation de la bactérie. On ignore encore la nature des protéines exportées responsables de cette activité. Un second système de type IV appelé Lvh, qui ne semble jouer aucun rôle dans la virulence, vient d’être mis en évidence chez L. pneumophila [ 20]. Brucella suis Brucella est l’agent d’infections affectant principalement des animaux domestiques ou sauvages (bovins, ovins, caprins, suidés, rongeurs, mammifères marins), et l’homme, chez qui il provoque la brucellose ou fièvre de Malte. Les deux caractéristiques principales de l’infection brucellienne sont la possibilité de passage à l’infection chronique et la capacité de la bactérie de se multiplier dans des cellules phagocytaires (principalement les macrophages) ou non (trophoblastes placentaires ou cellules endothéliales et épithéliales). L’infection de la cellule entraîne l’inhibition de la fusion phagosome-lysosome mais, contrairement à L. pneumophila, le phagosome reste acide [ 21]. Il est entouré de formations multilamellaires, le rapprochant d’un autophagosome [ 22]. Par ailleurs, l’infection des macrophages par Brucella provoque une stimulation de la synthèse d’IL-8 et une inhibition de la synthèse de TNFα ( tumor necrosis factor α) [ 23] et l’apoptose [ 24]. L’étude de mutants incapables de se multiplier dans des macrophages a permis de mettre en évidence la participation de nombreux gènes à la virulence de B. suis [ 25], en particulier un groupe de douze gènes formant un opéron, dont les onze premiers montrent une forte homologie avec l’opéron virB de A. tumefaciens et ptl de B. pertussis (Figure 2) [ 26]. Ce système de sécrétion, appelé VirB, existe aussi dans les autres espèces de Brucella [ 26, 27]. La mutation de ces gènes, y compris celle du douzième qui n’existe que chez Brucella, entraîne une totale incapacité de la bactérie de se multiplier dans le macrophage ou dans les cellules HeLa. Les mutants ne sont plus capables d’inhiber la fusion phago-lysosomiale, mais, en revanche, la synthèse d’IL-8 et l’inhibition de la synthèse de TNFα ne sont pas modifiées. Une étude récente a démontré que virB n’est pas induit au moment du contact entre la bactérie et la cellule, mais spécifiquement à l’intérieur du phagosome. De plus, le signal principal d’induction semble être le pH acide du phagosome, puisque sa neutralisation empêche alors l’induction. Il existe donc chez Brucella un système de sécrétion de type IV nécessaire à la virulence. Il est certainement responsable de la sécrétion de protéines, puisqu’il n’existe pas de plasmides naturels chez cette bactérie, mais la nature de la protéine ou des protéines exportée(s) n’est pas connue. Autres bactéries Le séquençage complet du génome de multiples bactéries a permis de retrouver chez certaines d’entre elles des équivalents des gènes de système de sécrétion de type IV. C’est le cas de Rickettsia prowazecki, l’agent du typhus exanthématique, de Bartonella henselae, qui provoque la maladie des griffes du chat, et de Actinobacillus actinomycetemcomitans, soupçonné d’être impliqué dans diverses infections du parodonte. Cependant, aucune preuve expérimentale n’est venue confirmer la réelle participation de ces gènes au pouvoir pathogène de ces bactéries. |
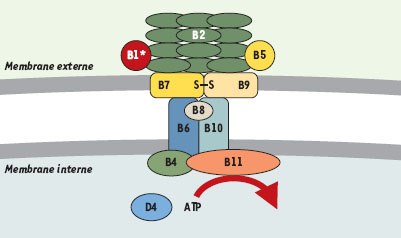
Structure des systèmes de sécrétion de protéines de type IV Les systèmes de sécrétion de type IV sont assemblés à partir des éléments constitutifs de la machinerie conjugative. L’analyse du système VirB de A. tumefaciens a permis d’identifier trois groupes fonctionnels de protéines VirB (Figure 3) [28, 29]. 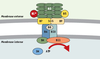 | Figure 3. Structure des systèmes de type IV. Localisation des composants VirB du système de transfert de l’ADN-T chez Agrobacterium tumefaciens. Les protéines VirB et VirD4 sont regroupées selon leur fonction probable: les protéines exocellulaires impliquées dans l’attachement à la cellule receveuse (VirB1*, VirB2 et VirB5), les protéines formant le pore (VirB3, VirB6, VirB7, VirB8, VirB9 et VirB10), et les ATPases de la membrane cytoplasmique (VirB4, VirB11 et VirD4). |
Le pilus La sous-unité principale du pilus est la protéine VirB2 [ 30]. Cette protéine est un homologue des protéines TraA et TrbC des plasmides F et RP4. La protéine, traduite sous forme de propiline (12 kDa), est transformée en une protéine mûre de 7 kDa capable de se polymériser pour former le pilus à la surface externe de la bactérie. La protéine VirB5 est également retrouvée comme constituant minoritaire du pilus T [ 31]. Le pilus pourrait permettre le contact entre la bactérie et la cellule végétale, induisant soit l’assemblage du système de transport dans la zone de contact, soit le recrutement dans cette même zone de contact des complexes assemblés au préalable. La protéine VirB1 possède des motifs caractéristiques des transglycosylases bactériennes: elle est donc susceptible de provoquer une lyse locale de la paroi de la bactérie permettant l’assemblage des autres constituants du pore. Elle peut également être transformée en un métabolite VirB1* (correspondant à la fraction carboxy-terminale) qui est sécrété à l’extérieur de la bactérie. VirB1* pourrait jouer un rôle soit dans le déclenchement de la formation du pilus, soit dans le contact avec la cellule receveuse [32]. Le canal transmembranaire Le canal transmembranaire, traversant les membranes interne et externe de la paroi, permet le transfert du complexe sécrété vers l’extérieur. Il est formé de nombreuses protéines. La structure prédite des protéines VirB6 à VirB10 permet d’en faire les constituants probables du pore transmembranaire. VirB6 et VirB10 sont des protéines de membrane interne alors que les protéines VirB7 et VirB9 sont des protéines de membrane externe. VirB6 représente le meilleur candidat comme protéine formant le pore. VirB7, une lipoprotéine de membrane externe, interagit avec VirB9: l’hétérodimère VirB7/VirB9 pourrait servir à stabiliser les autres protéines VirB [ 33]. D’autres interactions entre les protéines VirB8, VirB9 et VirB10 ont été récemment démontrées [ 30]. La protéine VirB10, qui présente un domaine périplasmique carboxy-terminal de grande taille, pourrait être le lien entre les éléments de la membrane externe et ceux de la membrane interne [ 30]. Les ATPases Deux protéines, VirB4 et VirB11, sont des ATPases associées à la membrane interne et apparaissent comme les moteurs du système de transport. Ces protéines utilisent l’énergie libérée par l’hydrolyse de l’ATP pour diriger soit le processus d’assemblage du système de transport, soit la translocation du substrat. |
Si de nombreux systèmes de type IV assurent le transport d’ADN, il faut toutefois noter que, dans ce cas, le substrat de conjugaison n’est jamais de l’ADN nu, mais del’ADN simple brin associé à des protéines. Les protéines ainsi co-transportées interagissent avec l’origine de transfert (oriT) pour former le complexe de transfert. Au cours de la conjugaison, les cellules donneuses engendrent un intermédiaire de transfert, comprenant l’ADN simple brin lié de façon covalente à son extrémité 5’ avec une endonucléase et, sur toute sa longueur, des protéines se liant à l’ADN simple brin. La reconnaissance de ce complexe nucléoprotéique par la machinerie de sécrétion est encore mal connue. L’hypothèse la plus vraisemblable est que la reconnaissance du substrat est conférée par les protéines associées, qui possèderaient des séquences peptidiques conservées ou encore des motifs de reconnaissance engendrés lors de leur repliement. L’appareil de conjugaison serait alors un système de sécrétion de protéines de type IV, permettant le transfert d’ADN associé [34]. Le transfert de l’ADN-T par A. tumefaciens ressemble beaucoup au processus de conjugaison. On observe en effet la formation du complexe T, qui est une association de l’ADN simple brin avec la protéine VirE2 et une endonucléase, la protéine VirD2, liée en 5’. Un argument confirmant l’hypothèse selon laquelle la machinerie de sécrétion reconnaît des protéines comme substrat est que la protéine VirE2 SSB (single strand binding protein) ou encore la protéine VirF de A. tumefaciens peuvent être sécrétées dans la cellule hôte de la plante indépendamment du complexe ADN-T/VirD2 [35, 36]. La machinerie de transfert de l’ADN-T de A. tumefaciens peut donc transporter les protéines VirE2, VirF, le complexe nucléoprotéique, mais elle est également capable de mobiliser des plasmides tels que le plasmide RSF1010. Ce transfert du plasmide RSF1010, comme dans le cas de Legionella, s’effectue au détriment des autres substrats, par un processus de compétition pour les systèmes de sécrétion disponibles [19, 37]. Une étude récente a en outre démontré le transfert possible de la protéine RecA par le système de conjugaison du plasmide RP4 chez Escherichia coli [34] et apporte un argument supplémentaire en faveur d’une composante protéique dans le processus de sécrétion. Dans le cas de sécrétion de protéines, des études récentes proposent l’intervention de protéines chaperons. En effet, chez A. tumefaciens, la protéine VirE1, nécessaire à la sécrétion de la protéine VirE2, est une petite protéine de type chaperon qui stabilise la protéine sécrétée VirE2 SSB [38]. Même si VirE1 en est à ce jour le seul exemple, la sécrétion de protéines par les systèmes de type IV requiert vraisemblablement des protéines annexes qui maintiennent le substrat dans un état de repliement compatible avec son transfert. Les processus de transport varient suivant la nature du substrat exporté. Dans le cas de la conjugaison ou dutransfert du complexe T chez A. tumefaciens, l’ADN doit rester intact pendant le processus. Le périplasme contenant de nombreuses nucléases susceptibles de dégrader l’ADN simple brin, la sécrétion d’ADN et des protéines associées est donc supposée se faire en une seule étape. Le système de sécrétion Ptl de la toxine de B. pertussis paraît différent sur plusieurs points. D’abord, un processus de sécrétion en deux étapes a été proposé. En effet, la toxine pertussique est formée de 5 sous-unités (S1-S5) qui possèdent chacune une séquence signal. Ces sous-unités doivent donc traverser la membrane interne par la voie générale de sécrétion dépendante de sec. La toxine est ensuite assemblée dans le périplasme avant d’être exportée à travers la membrane externe par le système de type IV. On a ensuite remarqué que, parmi les protéines VirB essentielles chez A. tumefaciens (VirB2-B11), seule VirB5 ne trouve pas son homologue chez B. pertussis. Une hypothèse a été avancée, selon laquelle la protéine VirB5 (ainsi que ses homologues Tra) serait nécessaire à la formation et au transfert du complexe protéine-ADN. Cependant, la région virB de B. suis possède un équivalent de virB5, ce qui ne confirme pas cette proposition, puisque, chez cette bactérie, le complexe VirB exporte très probablement une ou des protéines. Enfin, une autre différence importante caractérise B. pertussis. En effet, la toxine bactérienne est sécrétée dans le milieu extérieur et ne devrait pas nécessiter, contrairement aux autres substrats, un contact avec une cellule receveuse et donc n’exigerait pas la présence d’un pilus. Pourtant, il faut remarquer que la protéine PtlA de B. pertussis est l’homologue de la protéine VirB2 de A. tumefaciens, ce qui suggère que le système Ptl a conservé une trace de structure de type pilus. |
Évolution des systèmes de sécrétion de type IV Les systèmes de sécrétion de type IV ont donc comme fonctions connues soit le transfert de nucléoprotéines au cours de la conjugaison, soit celui de protéines pures, dans le cadre de la virulence. Actuellement, les premiers sont les mieux connus et les plus représentés dans le monde bactérien. En effet, la sécrétion d’holoprotéines n’a été démontrée que chez B. pertussis et H. pylori, et n’est que probable chez L. pneumophila et B. suis. Dans ces conditions, il est logique de penser que les systèmes de sécrétion d’holoprotéines sont dérivés des systèmes de conjugaison. Les arbres phylogénétiques construits à partir des séquences des différentes protéines formant l’opéron vont dans ce sens [39]. Il faut cependant noter que cette phylogénie moléculaire souffre, dans le cas des systèmes de type IV, de plusieurs insuffisances: d’une part, les séquences des gènes équivalents chez différentes bactéries sont trop divergentes pour permettre une interprétation statistiquement indiscutable de l’ordre des embranchements dans ces arbres ; d’autre part, la racine de ces arbres est le plus souvent placée de manière aléatoire. Ces deux inconvénients rendent difficile la désignation des lignées les plus anciennes. Une étude récente portant sur la phylogénie de 148 gènes de sécrétion de protéines homologues de VirB11, qui sont communes aux systèmes de type II et de type IV, vient de montrer que l’évolution de ces systèmes est beaucoup plus complexe qu’on ne l’a d’abord pensé [40]. L’arbre phylogénétique de ces différents gènes montre qu’ils forment une superfamille, divisée en deux familles correspondant aux systèmes de type II et de type IV, toutes deux dérivant d’un ancêtre commun (Figure 4). Les systèmes de type II ont pour unique fonction de sécréter des holoprotéines. Dans la famille dessystèmes de type IV, on observe une dispersion des systèmes engagés dans la conjugaison et de ceux responsables de la sécrétion d’holoprotéines, les deux étant regroupés ensemble dans plusieurs branches de l’arbre. Les conclusions de ces auteurs sont que: (1) il est difficile aujourd’hui de faire une proposition simple expliquant les relations évolutives entre les systèmes de conjugaison et de virulence; (2) comme le transfert d’ADN exige son couplage avec des protéines, la distinction entre les deux fonctions est peute tre artificielle. 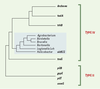 | Figure 4.Arbre phylogénétique des homologues de virB11 appartenant à des systèmes de sécrétion de type II et de type IV [ 40]. Les distances évolutives (longueur des branches) sont arbitraires, mais les embranchements sont respectés. La partie bleue correspond au groupe virB11 proprement dit, qui a été plus détaillé. Les gènes se distribuent en deux familles, correspondant aux systèmes de type II et de type IV. Les gènes de sécrétion de protéines pures ( Helicobacter, Bordetella et probablement Brucella) sont entremêlés avec des gènes de transfert de plasmides (gène lvh de Legionella, Agrobacterium) et donc ne dérivent pas d’un ancêtre commun. Le produit excrété par le complexe VirB de B. henselae n’est pas connu pour le moment. Enfin, une protéine sécrétée permettant l’adhérence de A. actinomycetemcomitans est codée par un gène du groupe tadA qui, par ailleurs, contient de nombreux systèmes de conjugaison. |
|
Grâce au séquençage systématique des génomes bactériens, le nombre des espèces chez qui un système de sécrétion de protéines de type IV est découvert ne cesse d’augmenter. Cependant, plusieurs inconnues demeurent. D’abord, il reste à déterminer si ces nouveaux systèmes jouent un rôle dans la conjugaison ou dans la virulence. Ensuite, si la structure du pore commence à être assez bien connue chezA. tumefaciens, elle est peu étudiée chez les autres bactéries. Il serait intéressant que cette connaissance soit élargie à ces autres exemples. En effet, chez les espèces dont la virulence dépend d’un tel système de sécrétion, on peut espérer découvrir des molécules capables de bloquer le transport des protéines de virulence et donc l’expression du pouvoir pathogène de ces bactéries. De telles substances pourraient servir de médicaments antibactériens non antibiotiques, permettant d’améliorer la thérapeutique des maladies correspondantes pour diverses raisons: soit en relais d’antibiotiques dont l’efficacité est amoindrie par les conditions environnantes (par exemple, le pH acide du contenu gastrique ou du phagosome), soit en bloquant de manière précoce la sécrétion de toxines responsables principales du tableau clinique (comme dans le cas de la coqueluche). |
1. Cossart P, Boquet P, Normark S, Falkow S. Cellular microbiology emerging. Science 1996; 271 : 315–6. 2. Wandersman C. Secretion across the bacterial outer membrane. In: Neidhardt C, ed. Escherichia coli and Salmonella: cellular and molecular microbiology. Washington, DC: ASM Press, 1996 : 955–66. 3. Hobbs M, Mattick JS. Common components in the assembly of type 4 fimbriae, DNA transfer systems, filamentous phages and proteinsecretion apparatus: a general system for the formation of surfaceassociated protein complexes. Mol Microbiol 1993; 10 : 233–43. 4. Hueck CJ. Type III secretion systems in bacterial pathogens of animals and plants. Microbiol Rev 1998; 62 : 379–433. 5. Anderson DM, Schneewind O. Type III secretion of Gram-negative pathogens: injecting virulence factors into host cells and more. Curr Opin Microbiol 1999; 2 : 18–24. 6. Christie PJ, Covacci A. Bacterial type IV secretion systems: DNA conjugation machines adapted for export of virulence factors. In: Cossart P, Boquet P, Normark S, Rappuoli R. eds. Cellular microbiology. Washington DC: ASM Press, 2000 : 265–73. 7. Christie PJ, Vogel JP. Bacterial type IV secretion: conjugation systems adapted to deliver effector molecules to host cells. Trends Microbiol 2000; 8 : 354–60. 8. Zambryski P. Basic processes underlying Agrobacterium-mediated DNA transfer to plant cells. Ann Rev Genet 1998; 22 : 1–30. 9. Christie PJ. The Agrobacterium tumefaciens T-complex transport apparatus: a paradigm for a new family of multifunctional transporters in eubacteria. J Bacteriol 1997; 179 : 3085–94. 10. Weiss AA, Johnson FD, Burns DL. Molecular characterization of an operon required for pertussis toxin secretion. Proc Natl Acad Sci USA 1993; 90 : 2970–4. 11. Covacci, A, Rappuoli R. Pertussis toxin export requires accessory genes located downstream from the pertussis toxin operon. Mol Microbiol 1993; 8 : 429–34. 12. Scarlato V, Ariço B, Domenighini M, Rappuoli R. Environmental regulation of virulence factor in Bordetella species. Bioassays 1993; 15 : 99–104. 13. Akopyants NS, Clifton SW, Kersulyte D, et al. Analyses of the cag pathogenicity island of Helicobacter pylori. Mol Microbiol 1998; 28 : 37–53. 14. Covacci A, Telford JL, Del Guidice G, Parsonnet J, Rappuoli R. Helicobacter pylori virulence and genetic geography. Science 1999; 284 : 1328–33. 15. Segal ED, Cha J, Lo J, Falkow S, Tompkins LS. Altered states: involvment of the phosphorylated CagA in the induction of the host cellular growthchanges by Helicobacter pylori. Proc Natl Acad Sci USA 1999; 96 : 14559–64. 16. Stein M, Rappuoli R, Covacci A. Tyrosine phosphorylation of the Helicobacter pylori CagA antigen after cag-driven host cell translocation. Proc Natl Acad Sci USA 2000; 97 : 1263–8. 17. Odenbreit S, Pull J, Sedlmaier B, Gerland E, Fisher W, Haas R. Translocation of Helicobacter pylori CagA into gastric epithelial cells by type IV secretion. Science 2000; 287 : 1497–500. 18. Hofreuter D, Odenbreit S, Puls J, Schwan D, Haas R. Genetic competence in Helicobacter pylori: mechanisms and biological implications. Res Microbiol 2000; 151 : 487–91. 19. Vogel JP, Andrews HL, Wong SK, Isberg RR. Conjugative transfer by the virulence system of Legionella pneumophila. Science 1998; 279 : 873–6. 20. Segal G, Russo JJ, Shuman HA. Relationships between a new type IV secretion system and the icm/dot virulence system of Legionella pneumophila. Mol Microbiol 1999; 34 : 799–809. 21. Porte F, Liautard JP, Köhler S. Early acidification of phagosomes containing Brucella suis is essential for intracellular survival in murine macrophages. Infect Immun 1999; 67 : 4041–7. 22. Pizarro-Cerda J, Meresse S, Parton RG, et al. Brucella abortus transits through the autophagocytic pathway and replicates in the endoplasmic reticulum of nonprofessionnal phagocytes. Infect Immun 1998; 66 : 5711–24. 23. Caron E, Gross A, Liautard JP, Dornand J. Brucella species release a specific, protease-sensitive inhibitor of TNF-alpha expression, active on human macrophage-like cells. J Immunol 1996; 156 : 2885–93. 24. Gross A, Terraza A, Ouahrani-Bettache S, Liautard JP, Dornand J. In vitro Brucella suis infection prevents the programmed cell death of human monocytic cells. Infect Immun 2000; 68 : 342–51. 25. Foulongne V, Bourg G, Cazevieille C, Michaux-Charachon S, O’Callaghan D. Identification of Brucella suis gene affecting intracellular survival in an in vitro human macrophage infection model by signature-tagged transposon mutagenesis. Infect Immun 2000; 68 : 1297–303. 26. O’Callaghan D, Cazevieille C, Allardet-Servent A, et al. A homologue of the Agrobacterium tumefaciens VirB and Bordetella pertussis Ptl type IV secretion systems is essential for intracellular survival of Brucella suis. Mol Microbiol 1999; 33 : 1210–20. 27. Sierra R, Comerci DJ, Sanchez DO, Ugalde RA. A homologue of an operon required for DNA transfer in Agrobacterium is required in Brucella abortus for virulence and intracellular multiplication. J Bacteriol 2000; 182 : 4849–55. 28. Burns DL. Biochemistery of type IV secretion. Curr Opin Microbiol 1999; 2 : 25–9. 29. Beijersbergen A, Smith SJ, Hooykass PJJ. Localization and topology of VirB proteins of Agrobacterium tumefaciens. Plasmid 1994; 32 : 212–8. 30. Lai EM, Kado CI. The T-pilus of Agrobacterium tumefaciens. Trends Microbiol 2000; 8 : 361–9. 31. Schmidt-Eisenlohr H, Domke N, Angerer C, Wanner G, Zambryski PC, Baron C. Vir proteins stabilize VirB5 and mediates its association with the Tpilus of Agrobacterium tumefaciens. J Bacteriol 1999; 181 : 7485–92. 32. Baron C, Llosa M, Zhou S, Zambryski PC. VirB1, a component of the Tcomplex transfer machinery of Agrobacterium tumefaciens, is processed to a C-terminal secreted product, VirB1*. J Bacteriol 1997; 179 : 1203–10. 33. Spudich GM, Fernadez D, Zhou ZR, Christie PJ. Intermolecular disulfide bonds stabilize VirB7 homodimers and VirB7/VirB9 heterodimers during biogenesis of the Agrobacterium tumefaciens T-complex transport apparatus. Proc Natl Acad Sci USA 1996; 93 : 7512–7. 34. Heinemann J. Genetic evidence of protein transfer during bacterial conjugation. Plasmid 1999; 41 : 240–7. 35. Citovsky V, Zupan J, Warnick D, Zambryski PC. Nuclear localization of Agrobacterium VirE2 protein in plant cells. Science 1992; 256 : 1802–5. 36. Regensburg-Tuink AJG, Hooykass PJJ. Transgenic N. glauca plants expressing bacterial virulence gene virF are converted into hosts for nopaline strains of A. tumefaciens. Nature 1993; 363 : 69–71. 37. Stahl LE, Jacobs A, Binns AN. The conjugal intermediate of plasmid RSF1010 inhibits Agrobacterium tumefaciens virulence and VirB-dependent export of VirE2. J Bacteriol 1998; 180 : 3933–9. 38. Deng W, Cheng L, Peng WT, et al. VirE1 is a specific molecular chaperone for the exported single stranted DNA binding protein VirE2 in Agrobacterium. Mol Microbiol 1999; 31 : 1795–807. 39. Winans SC, Burns DL, Christie PJ. Adaptation of a conjugal transfer system for the export of pathogenic macromolecules. Trends Microbiol 1996; 4 : 64–8. 40. Planet PJ, Kachlany SC, De Salle R, Figurski DH. Phylogeny of genes for secretion NTPases: identification of the widespread subfamily and development of a diagnostic key for gene classification. Proc Natl Acad Sci USA 2001; 98 : 2503–8. |