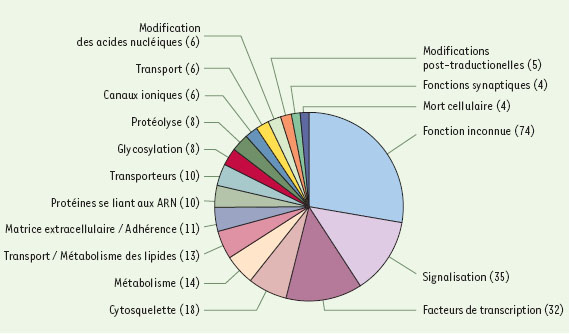
Les photorécepteurs (bâtonnets et cônes) sont les cibles cellulaires de nombreuses maladies rétiniennes [1]. Ces affections sont très hétérogènes sur le plan clinique, se distinguant par l’âge du patient lors de l’apparition des troubles visuels et, par ailleurs, par la sévérité ainsi que la distribution spatiale au sein de la rétine de la perte fonctionnelle des photorécepteurs. Jusqu’à présent, 132 locus mis en cause lors de maladies rétiniennes ont été localisés (cf. http://www.sph.uth.tmc.edu/RetNet/). Cependant, il est fort probable que le nombre de ces locus ne fera que s’accroître dans l’avenir. Sur ces 132 locus, 82 gènes ont été clonés, et environ 50 % d’entre eux sont spécifiquement exprimés par les photorécepteurs, confirmant qu’il existe une grande corrélation entre la spécificité tissulaire de l’expression génique et d’éventuelles manifestations cliniques (Figure 1). Une stratégie logique pour comprendre ces maladies consiste à identifier les gènes spécifiquement exprimés par les photorécepteurs et dont les mutations seraient susceptibles d’expliquer ces maladies. Au cours de ces dernières années, l’expression différentielle de gènes est devenue possible grâce à différentes techniques, dont l’hybridation soustractive, l’analyse des banques d’EST (expressed sequence tags), le differential display, les techniques de puces à ADN, et enfin le SAGE (serial analysis of gene expression). Cependant, les deux paramètres primordiaux que sont le seuil de sensibilité et la normalisation ont donné l’avantage aux techniques de puces à ADN et au SAGE, qui, toutes deux, se développent considérablement. Ainsi, afin de déterminer le profil d’expression des ARN exprimés spécifiquement par les photorécepteurs, l’équipe de recherche dirigée par le Dr C.L. Cepko (Department of Genetics, Howards Hugues Medical Institute, Havard Medical School, Boston, USA) a fait appel à la technique du SAGE. Ces travaux viennent de faire l’objet d’une publication dans Cell [2]. Avant de décrire les résultats obtenus, rappelons brièvement les principes de la technique du SAGE.
 |