Chez les mammifères, l’existence de chromosomes sexuels hétéromorphes implique la mise en place d’un mécanisme compensateur permettant d’aboutir à une équivalence de dose entre le mâle XY et la femelle XX. Touchant aléatoirement l’un des deux chromosomes X chez la femelle, ce processus, connu sous le nom d’inactivation du chromosome X, prend place au cours du développement embryonnaire précoce et se caractérise, in fine, par l’extinction transcriptionnelle de la quasi-totalité des quelque deux mille gènes portés par le gonosome touché. Proposé dès 1961, ce mécanisme derégulation génique concertée laisse encore la part belle au mystère. Cependant, les mécanismes moléculaires qui sous-tendent les phases précoces de ce phénomène sont aujourd’hui de plus en plus décortiqués et plusieurs éléments clés ont été caractérisés [1].
Le centre d’inactivation (Xic), région unique du chromosome X, est impliqué dans les étapes précoces de l’inactivation, en particulier le comptage du nombre de chromosomes X présents dans la cellule et le choix du chromosome à inactiver. Le Xic est également le site à partir duquel l’inactivation se propage en cis le long du chromosome X. Identifié au sein du Xic, le gène Xist engendre un transcrit nucléaire de grande taille, issu exclusivement du chromosome X inactif (Xi), lequel s’en trouve entièrement recouvert. Des expériences d’invalidation ont démontré le rôle majeur de ce gène dans l’inactivation, mais d’autres acteurs entrent également en jeu [2]. C’est le cas de Tsix, un gène antisens de Xist qui semble impliqué dans la régulation de ce dernier [3] (→). L’ARN Xist pourrait ensuite recruter des facteurs de nature encore inconnue, responsables de l’extinction transcriptionnelle des gènes associés en cis et de l’hétérochromatinisation conditionnelle qui caractérise l’inactivation. Le chromosome X inactif se distingue en effet de son homologue actif par un certain nombre de modifications chromatiniennes, dont la plupart se mettent en place tardivement et semblent jouer un rôle plus important dansle maintien que dans l’amorçage de l’inactivation.
(→) m/s 2000, n°6-7, p. 818
Ainsi, la nature des changements épigénétiques précoces de la chromatine du futur X inactif reste à découvrir. Très récemment, plusieurs publications ont montré le rôle de la lysine 9 de l’histone H3 (H3 Lys-9) et de ses modifications dans l’inactivation [4–6] et en particulier au niveau de son amorçage [4].
Les modifications covalentes des histones - méthylation, acétylation ou encore phosphorylation - sont déterminantes pour l’organisation de la chromatine des cellules eucaryotes [7]. Ces changements épigénétiques ont notamment pour conséquence de contrôler la transcription en empêchant ou, au contraire, en favorisant, l’accessibilitéde la chromatine à des facteurs régulateurs. Récemment observée comme marque de l’hétérochromatine chez de nombreux organismes (→), la méthylation de H3 Lys-9 par l’enzyme Suv39 engendre un site de fixation pour des protéines d’hétérochromatine, comme HP1 chez la souris. Grâce à la combinaison des techniques d’immunofluorescence couplée à l’ARN-FISH (fluorescent in situ hybridization) et d’immunoprécipitation de la chromatine (ChIP), réalisées sur des cellules souches embryonnaires, modèle de choix pour l’inactivation, Heard et al. [4] révèlent le rôle de la méthylation de H3 dans l’amorçage et la propagation de l’inactivation. Le Xi est en effet enrichi en histone H3 Lys-9 méthylée chez la souris comme chez l’homme [5], et des études cinétiques montrent que cet enrichissement est très précoce : mis en place en même temps que - ou juste après - la propagation de l’ARN Xist le long du chromosome X ; il précède donc l’extinctiontranscriptionnelle des gènes. La méthylation de H3 Lys-9 est associée à une hypoacétylation du même résidu ainsi qu’à l’hypométhylation de la lysine 4. Ces changements sont les plus précoces observés à ce jour et montrent combien les modifications de l’histone H3 sont déterminantes pour le déclenchement de l’inactivation.
(→) m/s 2001, n°4, p. 478
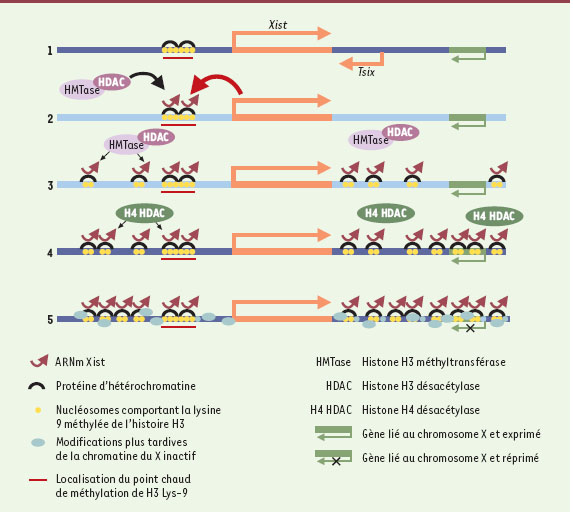
La fonction précoce de la méthylation de H3 Lys-9 est également illustrée par la pré-existence d’un point chaud (hotspot) de méthylation en 5’ de Xist [4], qui pourrait jouer le rôle d’un centre de nucléation à partir duquel l’hétérochromatine s’étendrait le long du chromosome à inactiver, et ce sous le contrôle de Xist. L’association de ce point chaud avec l’ARN Xist permettrait le recrutement privilégié d’un complexe multiprotéique incluant des histones désacétylases/méthyltransférases ainsi que des protéines d’hétérochromatine. Ces complexes étendraient leur action progressive à la chromatine adjacente, puis à l’ensemble du chromosome X, probablement par le biais d’éléments relais (Figure 1).
Si la mise en évidence d’un hotspot de méthylation de H3 Lys-9 souligne encore une fois l’importance des séquences cis dans l’inactivation, elle suppose également l’intervention de facteurs trans, comme les enzymes modificatrices d’histones (méthyltransférases) et les protéines d’hétéro-chromatine associées. En effet, ni Suv39 ni HP1 ne semblent impliquées dans la méthylation de H3 Lys-9 sur le Xi [6]. Des facteurs trans sont également supposés agir à d’autres étapes de l’inactivation, comme le comptage des chromosomes X et le choix de celui qui sera inactivé. Récemment, l’équipe de J. Lee a suggéré, sur la base de données expérimentales quelque peu préliminaires, l’implication de CTCF (CCCTC-binding factor) dans ce choix [8].
L’analyse in silico de DXPas34, une région de quelques kilobases en aval du gène Xist et impliquée dans l’étape du choix ayant révélé la présence de multiples sites de fixation de CTCF, les auteurs ont montré la fixation spécifique de cette protéine au niveau de certains de ces sites in vitro par gelretard, et in vivo par ChIP. Sur la base de ces données, et dans la mesure où la délétion de la région contenant les sites CTCF conduit à une inactivation systématique du chromosome délété, les auteurs postulent que CTCF est impliqué dans le choix du chromosome X à inactiver. Selon leur modèle, la fixation de CTCF sur l’un des deux X déterminerait le futur X actif (Xa) enempêchant l’activation de la transcription de Xist par un enhancer potentiel et en favorisant, a contrario, l’action répressive de Tsix sur l’expression de ce même gène. CTCF et Tsix agiraient donc de façon coordonnée pour la régulation de Xist. Cependant, les auteurs n’ont étudié la fixation de CTCF in vivo que dans des fibroblastes femelles, dans lesquels Tsix ne s’exprime plus et où le choix est déjà fait. De plus, les auteurs postulent que, sur le futur Xi, la fixation de CTCF serait abolie par la méthylation de la région. Or, la méthylation de cette région, du moins au niveau des CpG, est spécifique du Xa [9]. Ainsi, si CTCF est un candidat intéressant pour jouer un rôle dans l’inactivation, son rôle dans le choix du X à inactiver reste à démontrer.
| Figure 1. Cinétique de modification de la chromatine, en particulier au niveau de H3 Lys-9, durant la mise en route de l’inactivation du chromosome X. 1. Avant le début de l’inactivation, les gènes Xist et Tsix sont exprimés à bas niveau. Un point chaud de méthylation de H3 Lys-9 est présent en 5’ de Xist et serait associé à des facteurs protéiques. 2. A un moment critique du développement embryonnaire, l’expression de Tsix sur le futur Xi s’efface et les transcrits de Xist sont stabilisés. L’accumulation de ces nouveaux transcrits stables permettrait le recrutement d’un complexe comportant des protéines modificatrices des queues d’histones (HDAC/HMTase), ces dernières s’associant préférentiellement aux facteurs déjà présents sur le hotspot. 3. Propagation de la méthylation de H3 Lys-9 tout au long du chromosome X par recrutement, dépendant de l’ARN Xist, à la fois de facteurs d’hétérochromatine et de complexes HDAC/HMTase. Certaines régions du X pourraient jouer le rôle d’éléments relais et être la cible première de ce complexe ribonucléoprotéique. 4. Le remodelage de la chromatine (hétérochrosmatinisation) consécutif à la méthylation de H3 Lys-9 conduit à l’extinction transcriptionnelle des gènes liés au X ciblé et exprimés jusqu’ici. 5. Apparition des modifications plus tardives de la chromatine, telles que la désacétylation de l’histone H4 et l’accumulation de macroH2A. |