| |
| Med Sci (Paris). 2002 May; 18(5): 577–584. Published online 2002 May 15. doi: 10.1051/medsci/2002185577.Activation et modifications post-traductionnelles de p53 après dommage de l’ADN Virginie Leblanc and Pierre May* Laboratoirede Cancérogenèse moléculaire,UMR 217 CEA-Cnrs, DRR, DSV,CEA, BP n° 6,92265 Fontenay-aux-Roses Cedex,France |
Le suppresseur de tumeur p53 est l’une des plus importantes molécules intervenant dans la régulation de la croissance cellulaire et la protection des cellules contre la croissance tumorale. Elle joue un rôle central dans la réponse cellulaire au stress génotoxique et la perte de ce « gardien du génome » représente une étape importante de la carcinogenèse. Des mutations inactivatrices de p53 ont été observées dans plus de 50 % des tumeurs humaines (voir revue dans [1]). De telles mutations facilitent probablement la carcinogenèse en abrogeant les activités oncosuppressives de la protéine p53 sauvage, quoique certaine formes de mutants p53 peuvent contribuer à des activités oncogéniques (mutants à gain de fonction). p53 se comporte en suppresseur de tumeur en induisant l’arrêt de la croissance cellulaire ou l’apoptose en réponse à un grand nombre de stress cellulaires (exposition aux agents endommageant l’ADN, hypoxie, déficit en nucléotides, activation oncogénique inappropriée, etc.). Ces réponses empêcheraient la propagation des cellules comportant un ADN endommagé et présentant un potentiel oncogénique. Ce rôle de p53 comme « gardien du génome » est probablement la raison pour laquelle elle est la cible de mutations dans plus de 50 % de tous les cancers humains. Des travaux récents ont contribué de manière essentielle à la connaissance des mécanismes d’activation en réponse aux stress environnementaux et à l’expression inappropriée de signaux oncogéniques. L’activation de p53 peut être modulée à plusieurs niveaux : (1) une augmentation de la concentration en protéine ; (2) une augmentation de la demi-vie ; (3) la conversion de p53 d’une forme latente en une conformation active ; et (4) la translocation de p53 du cytoplasme au noyau. Il découle de la meilleure compréhension du mécanisme d’activation de p53 que des modifications post-traductionnelles de la protéine p53 sont un facteur essentiel de la régulation de p53 dans les cellules sressées et/ou non stressées. Nous résumons dans cet article les données récentes concernant les modifications post-traductionnelles de p53 après dommages de l’ADN (phosphorylations, déphosphorylations, acétylations et désacétylations). La concentration accrue de p53, modifiée de façon covalente (voir plus haut), permet à la protéine d’exercer sa fonction majeure qui est de se lier à des séquences particulières d’ADN et d’activer la transcription de gènes adjacents. L’expression de ces gènes conduit finalement, directement ou indirectement, à la mort cellulaire ou à l’inhibition de la division cellulaire. |
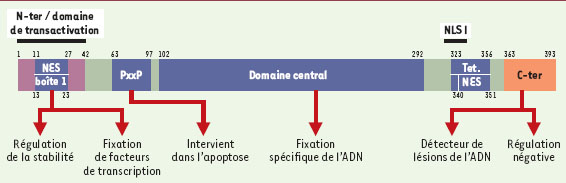
Des études récentes montrent que la protéine p53 sauvage est impliquée dans la transcription de gènes, la synthèse de l’ADN, la réparation, la sénescence, la plasticité du génome, l’apoptose et l’angiogenèse. p53 est une protéine fixant l’ADN d’une manière spécifique. Elle reconnaît une séquence d’ADN contenant deux copies d’un motif de 10 paires de bases (appelé ER pour élément de réponse à p53). p53 active l’expression des gènes contenant un ER. p53 peut aussi réprimer la transcription d’autres gènes en interagissant avec l’appareil cellulaire de transcription. La fixation spécifique de p53 à l’ADN est nécessaire à sa fonction onco-suppressive. En effet, toutes les formes mutées de p53 provenant de tumeurs présentent une affinité réduite pour l’ADN [1]. L’activité transcriptionnelle de p53 est impliquée dans la régulation de processus tels que le cycle cellulaire, l’apoptose et la réparation de l’ADN. Comme le montre la Figure 1, la protéine p53 est composée : (1) d’un domaine N-terminal (résidus 1-42) nécessaire à l’interaction avec les composants de l’appareil transcriptionnel ; ce domaine inclut une séquence très conservée (résidus 13-23, boîte 1) (Figure 1) ; (2) d’une région riche en résidus proline, contenant plusieurs copies du motif PxxP (résidus 63-97) [2] ; (3) d’un domaine central, très conservé, de fixation à l’ADN (résidus 102-292), contenant la plupart des mutations inactivatrices retrouvées dans différents types de cancers humains ; (4) d’un domaine de tétramérisation (résidus 323-356), qui facilite la fixation spécifique de p53 à l’ADN ; (5) et d’un domaine de régulation négative, C-terminal (résidus 363-393), qui inhibe la fonction de fixation spécifique à l’ADN. Enfin, la présence de séquences d’exportation vers le cytoplasme (NES, nuclear export signal) et aux extrémités N- et C-ter-minales, ainsi que des séquences de localisation nucléaire (NLS, nuclear localization signal) à l’extrémité C-terminale, permettent la régulation de la localisation subcellulaire de p53 [3].  | Figure 1. Domaines structuraux et fonctionnels de la protéine p53. La protéine p53 compte 393 acides aminés. Elle comprend un domaine acide ou domaine de transactivation (acides aminés 1-42), un domaine PxxP riche en pro-line, supposé nécessaire pour régler la stabilisation de p53 et l’apoptose, un domaine central (acides aminés 102-292) de fixation spécifique à des séquences d’ADN, un domaine de tétramérisation (acides aminés 335-356) et un domaine extrême C-terminal (acides aminés 363-393) de régulation négative de la fixation spécifique à l’ADN et fonctionnant comme détecteur de lésions de l’ADN. Les domaines acides aminés 300-323 et acides aminés 323-363 correspondent respectivement à une séquence de localisation nucléaire (NLS) et à une séquence d’exportation nucléaire (NES). La protéine p53 possède 5 régions très conservées. Seule la région conservée « boîte 1 » (acides aminés 13-23) est indiquée, ce sous-domaine est impliqué dans la régulation de la stabilité de p53. Une séquence coïncidant à peu près avec la boîte 1 (acides aminés 11-27, NES) correspond à un signal d’exportation nucléaire. Les fonctions de chaque domaine sont indiquées sous les flèches verticales. NES : signal d’exportation nucléaire ; Tet : domaine de tétramérisation ; NLS I : signal de localisation nucléaire ; N-ter : domaine N-terminal ; C-ter : domaine C-terminal. |
|
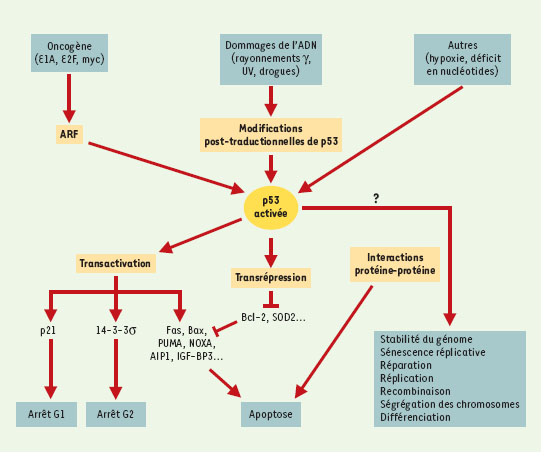
Réponse dépendante de p53 après un stress génotoxique En réponse à un dommage de l’ADN, à l’hypoxie, à un déficit en nucléotides ou à l’action d’agents chimiothérapeutiques, p53 subit des modifications post-traductionnelles ayant pour conséquence l’accumulation d’une protéine active sur le plan de la transcription (Figure 2). L’activation de p53 faisant suite aux dommages causés à l’ADN après radiation γ ou UV a été particulièrement étudiée. Nous nous limiterons dans cet article à ces deux types de stress. Il a été montré que p53, activée après radiation γ, provoque l’arrêt du cycle cellulaire ou l’apoptose. Ces deux processus éliminent les cellules génétiquement altérées et assurent l’intégrité structurale du génome cellulaire, inhibant de ce fait le développement tumoral. L’activation de p53, notamment par radiation, permet d’induire la fixation spécifique de p53 à un ER et l’expression des gènes contenant cet ER (gènes impliqués dans le contrôle du cycle cellulaire et/ou de l’apoptose). De nombreuses études suggèrent que la régulation des activités transcriptionnelles de p53 dépend de l’état phosphorylé et acétylé de cette protéine. La phosphorylation de p53 régit également sa stabilité en contrôlant ses interactions avec MDM2, une protéine qui facilite la dégradation protéolytique de p53 par la voie de l’ubiquitine. Lorsque l’activité protéolytique du protéasome est bloquée par des inhibiteurs spécifiques, l’excès de MDM2 favorise l’accumulation des formes ubiquitinylées de p53, suggérant que MDM2 facilite l’ubiquitinylation. Cette conclusion est renforcée par l’observation selon laquelle MDM2 peut fonctionner directement in vitro en tant qu’ubiquitine-protéine ligase E3, spécifique de p53. 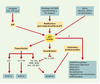 | Figure 2. Modèle de l’activation de p53 par dommages de l’ADN et par une expression inappropriée d’oncogènes. Un dommage causé à l’ADN peut augmenter l’abondance de p53, provoquant une rapide activation des kinases qui modifient p53. Il en résulte une rupture de l’association entre p53 et MDM2. Les kinases employées et les sites modifiés sur p53 peuvent différer selon les types d’irradiation ou de stimulus responsables du dommage (par exemple, l’irradiation γ active la kinase ATM, contrairement à l’irradiation UV). Par ailleurs, les oncogènes peuvent activer p53 par une voie différente. Une expression inappropriée d’oncogènes peut produire l’induction du facteur de transcription E2F (non représenté sur la figure) qui peut induire l’expression du gène ARF. Le gène ARF se fixe sur MDM2, inhibant la fonction ubiquitine-ligase de MDM2, ce qui protège p53 de la dégradation protéolytique par la voie MDM2. Il en résulte l’accumulation de p53 et son activation, induisant l’expression de gènes en aval, gènes impliqués dans l’arrêt de croissance ou l’apoptose des cellules. La fonction principale de p53 est de stimuler la transcription de nombreux gènes. Cependant, p53 agit également en réprimant d’autres gènes et en provoquant des mécanismes indépendants de la transcription tels que des interactions protéine/protéine. |
L’activation de p53 peut conduire à l’arrêt du cycle cellulaire aux frontières G1/S ou G2/M [4]. L’activation de l’expression du gène p21WAF1 est en grande partie responsable de l’arrêt en G1/S. Ce gène code pour la protéine p21WAF1 qui est un inhibiteur de presque tous les complexes y compris les cyclines D-Cdk4/6, E-Cdk2 et A-Cdk2. Lorsque p53 est activée par un dommage de l’ADN, p21WAF1 augmente considérablement et arrête le cycle cellulaire en inhibant les complexes cycline D/Cdk4/6 et cycline A,E/Cdk2. La stimulation par p53 de l’expression du gène codant pour la protéine 14-3-3σ est en partie responsable de l’arrêt en G2. Par ailleurs, il a été montré que 14-3-3σ est fortement induite par les radiations γ ainsi que par d’autres agents endommageant l’ADN [5]. Le mécanisme par lequel p53 stimule l’apoptose n’est pas encore complètement établi. Les gènes liés à l’apoptose et transactivés par p53 incluent Bax (dont la protéine est antagoniste de Bcl-2), NOXA, p53AIP-1 (apoptosis induced protein 1). Il existe d’autres médiateurs de l’apoptose comme Fas/APO de la famille des récepteurs du TNF (tumor necrosis factor), PIG3 (p53 inducible gene), impliqué dans la régulation de la production d’espèces réactives de l’oxygène. Outre sa fonction d’activateur, p53 réprime aussi l’expression de gènes en interagissant physiquement avec d’autres composants de la machinerie transcriptionnelle, comme TBP (TATA box binding protein) ou d’autres facteurs régulateurs positifs ou négatifs de transcription [6]. Parmi les gènes réprimés par p53 se trouvent le gène anti-apoptotique Bcl-2, les gènes c-fos, MnSOD2, cycline A, interleukine-6 [7]. La fonction de trans -répression de p53 semble particulièrement importante pour son activité apoptotique. Le rôle biologique de p53 ne se limite pas à l’arrêt de croissance et à la mort cellulaire programmée : elle participe également au maintien de la stabilité du génome en étant impliquée à la fois dans la réparation de l’ADN, la recombinaison et la ségrégation chromosomique [8]. |
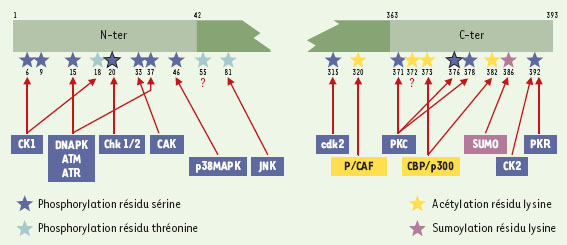
Modifications post-traductionnelles de p53 (phosphorylation et acétylation) La phosphorylation et l’acétylation sur de multiples sites de la protéine p53 se produisent après exposition des cellules à des agents génotoxiques. Des anticorps qui reconnaissent spécifiquement des sites phosphorylés ou acétylés sur p53 ont été utilisés. Les sites de phosphorylation sont regroupés aux extrémités N-(sérines 6, 9, 15, 20, 33, 37, 46 et thréonines 18, 55 et 81) et C-terminales (sérines 315, 371, 376, 378 et 392) tandis que l’acétylation et l’ubiquitinylation ne concernent que l’extrémité C-terminale au niveau du bloc des lysines 370, 372, 373, 381 et 382 (Figure 3). Il a également été montré que les sérines 20, 315 et 376 pouvaient être déphosphorylées [9](→). (→) m/s2001, n°11, p. 1242 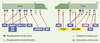 | Figure 3. Modifications post-traductionnelles de p53. Représentation schématique de la protéine p53 et des résidus spécifiques des acides aminés qui sont sujets à une phosphorylation, une déphosphorylation, une acétylation ou une sumoylation (SUMO fait partie d’un nombre croissant de protéines de type ubiquitine qui modifient de façon covalente leurs protéines cibles) après traitement génotoxique. La localisation des sites de phosphorylation, d’acétylation et de sumoylation est indiquée, ainsi que les kinases et les acétylases qui modifient ces sites in vitro et, dans la plupart des cas, in vivo. Les co-activateurs et la protéine SUMO sont indiqués dans leur site d’association. Les chiffres se rapportent aux résidus acides aminés. Bien que les modifications post-traductionnelles de la plupart des sites se produisent en réponse à un stress donné, des différences dans des sites individuels, en réponse à divers types de stress, ont été observées. |
Phosphorylations de la région amino-terminale et rôle dans l’activation de p53 Les protéine kinases connues pour répondre au stress cellulaire et pouvant phosphoryler p53 in vitro sont d’excellents candidats pour la phosphorylation de p53 in vivo. Parmi elles, les membres de la famille des kinases phosphatidylinositol 3’ (DNA-PK pour DNA-activated protein kinase ; ATM kinase pour ataxia telangiectasia mutated kinase ; ATR kinase pour A-T related kinase) phosphorylent p53 in vitro [10], et certains membres de la famille des SAK (stress-activated kinases) telles que JNK-1 et p38MAPK [11, 12]. DNA-PK est une sérine-thréonine kinase nucléaire pouvant phosphoryler p53 sur les sérines 15 et 37. ATM, produit du gène ATM muté chez les patients présentant une ataxie télangiectasie, peut phosphoryler p53 sur la sérine 15 in vitro. ATR peut phosphoryler les sérines 15 et 37 in vitro. La phosphorylation sur la sérine 15 stimule l’interaction de p53 avec ses co-activateurs transcriptionnels, les histone-acétyltransférases p300 et CBP (CREB binding protein) [13]. En outre, la sérine 20 est phosphorylée in vitro par Chk1 et Chk2 (check point kinases), qui sont activées par ATM et ATR après un dommage de l’ADN [10]. Une étude in vivo montre qu’en réponse aux UV et aux radiations γ, la phosphorylation de p53 sur la sérine 20 a pour effet d’augmenter la transactivation des gènes p21 et mdm2 [14]. En outre, comme pour le résidu 15, la phosphorylation du résidu 20 peut activer p53 en inhibant son interaction avec MDM2 et en participant à la stabilisation du complexe p53/p300 [15]. Par ailleurs, on a montré que p53, phosphorylée au résidu 15 après exposition des cellules aux UV, n’était pas exportée vers le cytoplasme. Ainsi, la phosphorylation de ces deux résidus participerait à la stabilisation de p53 et à l’inhibition de l’exportation de p53 vers le cytoplasme [16]. P38MAPK1 peut phosphoryler p53 sur le résidu 46. Cette phosphorylation favorise la transactivation du gène pro-apoptotique p53AIP-1 [17]. JNK1 appartient également à la famille des kinases activées par le stress. La phosphorylation de la thréonine 81 par JNK1 intervient dans la stabilisation de p53 et dans son activité transcriptionnelle en réponse à un stress [11]. Les sérines 33 et 37 sont également phosphorylées in vivo après irradiation UV par JNK1 [18]. Des études in vitro et in vivo suggèrent que la fixation de JNK à p53 cause l’ubiquitinylation et la dégradation de p53. La sérine 33 est le substrat in vitro de CAK (cycline dependent kinase activating kinase). De même, les résidus sérines 6 et 9 de p53 peuvent être phoshorylés in vitro par la kinase CKI (caséine kinase I). La phosphorylationdu résidu thréonine 18 par CKI peut activer p53 en stabilisant le complexe p300-p53. Par ailleurs, cette phosphorylation peut inhiber la fixation de p53 à MDM2 [15]. Enfin, il a été mis en évidence le fait que la thréonine 55 peut être phosphorylée in vitro. De l’ensemble de ces études, il ressort que, lorsque le domaine N-terminal est la cible de kinases, la protéine p53 est stabilisée par perte d’interaction avec MDM2 et est rendue compétente pour la transcription par interaction avec CBP/p300. Phosphorylations de la région C-terminale et rôle dans la fixation spécifique de p53 à l’ADN Le domaine C-terminal est modifié par plusieurs kinases comme la kinase dépendante des cyclines (Cdk2), la PKC (protéine kinase C), la CKII (caséine kinase II) ou encore la PKR ( double-stranded-RNA-activated protein kinase), mais également par des phosphatases et des histones acétyltransférases (HAT) telles que p300, PCAF ( p300/CBP associated factor) ou CBP. Le résidu sérine 315 est une cible de p34 cdc2 et de Cdk2 [ 19, 20]. In vivo, la phosphorylation de la sérine 315 provoque l’augmentation de l’activité de fixation à l’ADN et stimule l’activation transcriptionnelle de p53. Les sérines 376 et 378 sont des sites de phosphorylation par la PKC. Ces deux résidus sont constitutivement phosphorylés dans des cellules non irradiées (MCF7) [ 21]. Selon certains auteurs, il semblerait que, dans des cellules non stressées, la phosphorylation de l’extrémité C-terminale par la PKC augmenterait l’ubiquitinylation et la dégradation de p53 [ 22]. La sérine 392 est phosphorylée in vitro par la caséine kinase II ; in vivo, ce site est phosphorylé après irradiation UV mais pas après irradiation γ [ 23]. Le résidu 392 peut également être le substrat in vitro de la PKR [ 24]. I n vitro, la phosphorylation des sérines 315, 378, 392 augmente la fixation spécifique de p53 à l’ADN. Enfin, la phosphorylation de la sérine 392 favoriserait la tétramérisation de p53, ce qui activerait p53 en tant que facteur transcriptionnel [ 25]. Il apparaît donc que la phosphorylation de l’extrémité C-terminale joue un rôle déterminant dans la fixation spécifique de p53 à l’ADN. Déphosphorylation Une déphosphorylation de la sérine 20 est coordonnée à une augmentation de la phosphorylation du résidu sérine 392, après irradiation non ionisante [ 26]. Par ailleurs, il a été décrit que la phospho-sérine 315 pouvait être le substrat d’une phosphatase, Cdc-14, intervenant dans le contrôle de la progression du cycle cellulaire [ 27]. Les sérines 376 et 378 sont phosphorylées de façon constitutive dans les cellules non irradiées. La déphosphorylation de la sérine 376 favorise la fixation de la protéine 14-3-3 (impliquée dans le point de contrôle G2 dépendant de p53) et contribue à l’activation de p53 en réponse aux radiations ionisantes [ 28]. Acétylation L’acétylation de la lysine 320 par PCAF et/ou les lysines 373 et 382 par p300/CBP contribue également à l’activation de p53. Il a été suggéré que la phosphorylation des sérines 15, 33 et 37 permet l’acétylation de résidus lysines de la région distante C-terminale, au moyen d’un recrutement accru de p300/CBP/PCAF [ 29] causant ainsi une augmentation de la capacité de fixation spécifique à l’ADN ( Figures 3 et 4). L’acétylation a été initialement supposée favoriser la fixation de p53 à l’ADN. Cependant, de nombreux résultats ont amené à rejeter cette hypothèse (voir revue dans [30]). En outre, des analyses in vitro de la fixation de p53 à la chromatine ont confirmé que l’acétylation en C-terminal de p53 n’est pas requise pour que p53 se fixe à l’élément de réponse ER, bien que l’activité de p300/CBP histone acétylase soit nécessaire à la transactivation induite par p53. Ces résultats suggèrent un modèle dans lequel le recrutement de p300/CBP dans le domaine N-terminal aboutirait à l’acétylation dans le domaine C-terminal qui affecterait l’ubiquitinylation induite par MDM2, avec pour conséquence une stabilité accrue de p53. En outre, p300/CBP lié à p53 pourrait acétyler des histones dans les éléments de réponse de p53, conduisant à des modifications de la chromatine nécessaires à la transactivation de p53 [30]. |
Détection par la cellule d’ADN endommagé et famille ATM Après l’identification des sites de phosphorylation, les efforts se sont concentrés pour élucider les voies en aval déclenchant les événements de phosphorylation [31]. Des différents types de dommages causés à l’ADN dans la cellule, le plus létal est le DSB (double strand break). Dès que des DSB se produisent dans la cellule, le signal de dommage à l’ADN est activé par une cascade de protéine kinases. Les protéine kinases se trouvant en amont de cette cascade ont des similitudes avec la famille des phosphatidyl-inositol-kinases. Deux kinases de cette famille, ATM et ATR (ATM-rad3-related), sont cruciales pour la détection de DBS chez les mammifères. Le gène ATM code pour une protéine de masse moléculaire relative de 350 kDa, contenant un domaine de fixation à l’ADN et un domaine catalytique phosphatidyl inositol 3 kinase. Il a été prouvé qu’ATM se fixe directement à des extrémités libres de l’ADN. Ces protéine kinases phosphorylent et activent les protéine kinases en aval, Chk2 et Chk1. Il reste cependant beaucoup à apprendre sur les gènes et les facteurs impliqués dans la stabilisation et l’activation de p53. |
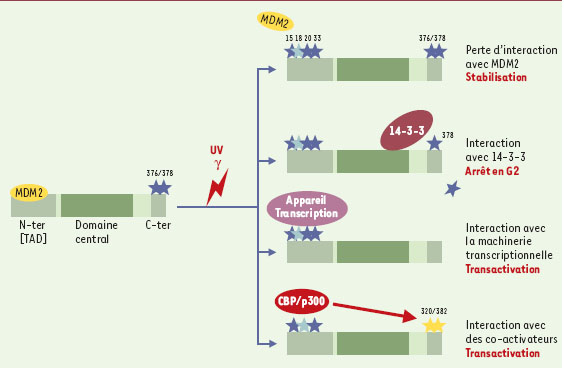
Stabilisation de p53 : contribution et interdépendance des modifications post-traductionnelles et des interactions protéines-protéines La phosphorylation de résidus dans la région N-terminale (sérines 15, 20, 33 et thréonine 18) de p53 est nécessaire pour régler sa fixation à MDM2 ainsi que sa localisation nucléaire [15, 16]. Dans les cellules non stressées, p53 est latente et maintenue à faible concentration par une dégradation dépendante de MDM2. Ce régulateur négatif fonctionne comme une ubiquitine ligase et déclenche à la fois un transport du noyau vers le cytoplasme et une dégradation de p53 par le protéasome [32]. Le ciblage de la dégradation impliquerait également p300 qui pourrait fixer à la fois MDM2 et p53. Cela suggère l’existence d’un double rôle de p300 dans la fonction transactivatrice de p53 ainsi que dans sa dégradation [33]. La suppression des 30 derniers acides aminés donne une molécule p53 encore capable de fixer MDM2, mais réfractaire à la dégradation contrôlée par MDM2 [34]. La protéine ARF, codée par le locus INK4a/ARF, et induite par l’expression inappropriée d’oncogènes, est capable, en se fixant à MDM2, d’inhiber la dégradation de p53 causée par MDM2 [35] (Figure 2). Le rôle majeur de MDM2 dans la régulation de la stabilité de p53 n’empêche pas la contribution d’autres protéines, notamment JNK qui intervient de manière indépendante de MDM2 [36]. |
Nous avons montré dans cet article que la protéine p53 humaine est modifiée de façon post-traductionnelle sur plus d’une dizaine de sites. Des anticorps polyclonaux et monoclonaux recon-naissant spécifiquement les sites modifiés ont permis de mettre en évidence - pour la plupart des sites de modifications - une augmentation de la phosphorylation en réponse à des dommages de l’ADN. Sept sérines et deux thréonines dans le domaine N-terminal de p53, à savoir Ser6, 9, 15, 20, 33, 46 et Thr18, 81, sont phosphorylées à la suite d’un stress. Dans le domaine de régulation C-terminal, Ser315 et 392 sont phosphorylées, Lys320, 373 et 382 sont acétylées, alors que Ser376 et 378 sont phosphorylées constitutivement dans des cellules non stressées. La phosphorylation de la région N-terminale, induite en réponse à un stress génotoxique, augmente la stabilité de p53 et sa capacité d’interagir avec l’appareil cellulaire de transcription, alors que la phosphorylation du domaine C-terminal facilite la fixation de p53 à des séquences spécifiques de l’ADN (Figure 4). Toutefois, les modifications post-traductionnelles de p53 ne sont pas les seuls éléments de la régulation de la fonction de p53. Rappelons que la protéine MDM2 constitue un composant majeur d’une voie de régulation négative. La protéine p300, adaptateur transcriptionnel, forme le composant d’une voie régulatrice positive qui facilite l’induction d’expression de gènes dépendants de p53. L’équilibre entre la capacité de p300 et de MDM2 d’entrer en compétition pour se fixer sur le même domaine N-terminal (boîte 1) permet de moduler l’activité spécifique de p53 comme suppresseur de tumeur. Il faut rappeler le rôle prédominant de la phosphorylation de la thréonine 18 et de la sérine 20, occasionnant la dissociation du complexe p53-MDM2 au profit du complexe p53-p300 (Figure 4), ce qui montre que la phosphorylation de ces deux résidus est un événement associé à l’activation de p53. Par ailleurs, une étude a montré que les radiations γ produisent la déphosphorylation de la sérine 376. Cette modification a pour conséquence la création d’un site de fixation pour la protéine 14-3-3 (Figure 4) et l’association de p53 avec cette protéine augmente l’affinité de p53 pour les séquences spécifiques d’ADN. Il est concevable que les modifications post-traductionnelles puissent modifier la conformation de la protéine p53, son affinité pour ses protéines partenaires ainsi que pour ses séquences cibles. 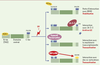 | Figure 4. Stabilisation et activation de p53 par modification des interactions protéine-protéine. Dans les cellules en croissance normale, la protéine p53 est associée, à son extrémité N-terminale (transactivation domain, TAD), à la protéine MDM2 ; les sérines 376 et 378 sont phosphorylées. Après irradiation UV ou γ, les sérines 15, 20 et 33 (étoiles bleu foncé) et la thréonine 18 (étoile bleu clair) deviennent phosphorylées provoquant une perte d’interaction avec MDM2 et une stabilisation de p53 ; p53 s’accumule dans le noyau et n’est pas dégradée. La déphosphorylation de la sérine 376 crée un site d’association à la protéine 14-3-3 σ, ce qui a pour effet d’augmenter l’affinité de p53 pour les sites spécifiques d’ADN. Différents facteurs de l’appareil de transcription interagissent avec la région N-terminale (TAD). Les co-activateurs de transcription p300/CBP se fixent sur les sites phosphorylés 15, 18, 20, induisant à distance l’acétylation des Lys 320 et des Lys 382 (étoiles jaunes), et renforçant l’activité transcriptionnelle de p53. |
Dans cette étude, nous avons concentré notre attention sur les effets des radiations UV et γ. Élargissant notre point de vue à d’autres types de stress, l’activité transcriptionnelle de p53 est induite par de nombreux agents importants comme signaux suppresseurs de tumeur dans les cancers en évolution. Ces agents comprennent les oxydants chimiques, la perturbation du pool des nucléotides, un pH extracellulaire bas, une hypoxie, etc. En outre, des composés anti-tumoraux classiques, notamment l’adriamycine, le 5-fluorouracile et l’étoposide peuvent activer la fonction de p53. Bien que de nombreux agents de stress distincts et divers types de cellules partagent des caractéristiques similaires dans la régulation de p53, aucun type cellulaire, ni aucun agent de stress ne peut être utilisé pour généraliser toutes les étapes connues de la voie d’activation de p53. Les profils très détaillés de gènes obtenus en utilisant des microfiltres d’oligonucléotides ont clairement montré des différences et des similitudes dans les produits de gènes induits ou réprimés par p53 dans différents types cellulaires et dans diverses conditions [37]. La liste des gènes dont l’expression est réglée en réponse à p53 est caractérisée par une grande hétérogénéité. Celle-ci dépend de la nature du stress imposé pour activer p53 et du type cellulaire. Il serait intéressant de développer une étude systématique pour établir un catalogue des gènes réglés dans chaque situation expérimentale. Cette hétérogénéité pourrait expliquer les différences entre les réponses aux chimiothérapies et aux radiations pour les divers types de cellules ou de tissus tumoraux. |
1. May P, May E. Twenty years of p53 research: structural and functional aspects of the p53 protein. Oncogene 1999; 18 : 7621–36. 2. Venot C, Maratrat M, Dureuil C, et al. The requirement for the p53 proline-rich functional domain for mediation of apoptosis is correlated with specific PIG3 gene transactivation and with transcriptional repression. EMBO J 1998; 17 : 4668–79. 3. Gottifredi V, Prives C. Molecular biology: getting p53 out of the nucleus. Science 2001; 292 : 1851–52. 4. North S, Hainaut P. p53 and cell-cycle control: a finger in every pie. Pathol Biol 2000; 48 : 255–70. 5. Hermeking H, Lengauer C, Polyak K, et al. 14-3-3 sigma is a p53-regulated inhibitor of G2/M progression. Mol Cell 1997; 1 : 3–11. 6. Ko LJ, Prives C. p53: puzzle and paradigm. Genes Dev 1996; 10 : 1054–72. 7. Drane P, Bravard A, Bouvard V, May E. Reciprocal downregulation of p53 and SOD2 gene expression: implication in p53 mediated apoptosis. Oncogene 2001; 20 : 430–9. 8. Vogelstein B, Lane D, Levine AJ. Surfing the p53 network. Nature 2000; 408 : 307–10. 9. Appella E, Anderson CW. Post-translational modifications and activation of p53 by genotoxic stresses. Eur J Biochem 2001; 268 : 2764–72. 10. Caspari T. How to activate p53. Curr Biol 2000; 10 : R315–7. 11. Buschmann T, Potapova O, Bar-Shira A, et al. Jun NH2-terminal kinase phosphorylation of p53 on Thr-81 is important for p53 stabilization and transcriptional activities in response to stress. Mol Cell Biol 2001; 21 : 2743–54. 12. Bulavin DV, Saito S, Hollander MC, et al. Phosphorylation of human p53 by p38 kinase coordinates N-terminal phosphorylation and apoptosis in response to UV radiation. EMBO J 1999;18 : 6845–54. 13. Dumaz N, Meek DW. Serine15 phosphorylation stimulates p53 transactivation but does not directly influence interaction with HDM2. EMBO J 1999; 18 : 7002–10. 14. Jabbur JR, Huang P, Zhang W. DNA damage-induced phosphorylation of p53 at serine 20 correlates with p21 and Mdm-2 induction in vivo. Oncogene 2000; 19 3–8. 15. Dornan D, Hupp TR. Inhibition of p53-dependent transcription by BOX-I phospho-peptide mimetics that bind to p300. EMBO Rep 2001; 2 : 139–44. 16. Zhang YP, Xiong Y. A p53 amino-terminal nuclear export signal inhibited by DNA damage-induced phosphorylation. Science 2001; 292 : 1910–5. 17. Oda K, Arakawa H, Tanaka T, et al. p53AIP1, a potential mediator of p53-dependent apoptosis, and its regulation by Ser-46-phosphorylated p53. Cell 2000; 102 : 849–62. 18. Bean LJH, Stark GR. Phosphorylation of serines 15 and 37 is necessary for efficient accumulation of p53 following irradiation with UV. Oncogene 2001; 20 : 1076–84. 19. Price BD, Hughesdavies L, Park SJ. cdk2 kinase phosphorylates serine 315 of human p53 in vitro. Oncogene 1995; 11 : 73–80. 20. Bischoff JR, Friedman PN, Marshak DR, Prives C, Beach D. Human p53 is phosphorylated by p60-cdc2 and cyclin B-cdc2. Proc Natl Acad Sci USA 1990; 87 : 4766–70. 21. Jimenez GS, Bryntesson F, Torres-Arzayus MI, et al. DNA-dependent protein kinase is not required for the p53-dependent response to DNA damage. Nature 1999; 400 : 81–3. 22. Chernov MV, Bean LJH, Lerner N, Stark GR. Regulation of ubiquitination and degradation of p53 in unstressed cells through C-terminal phosphorylation. J Biol Chem 2001; 276 : 31819–24. 23. Kapoor M, Lozano G. Functional activation of p53 via phosphorylation following DNA damage by UV but not gamma radiation. Proc Natl Acad Sci USA 1998; 95 : 2834–7. 24. Cuddihy AR, Wong AH, Tam NW, Li S, Koromilas AE. The double-stranded RNA activated protein kinase PKR physically associates with the tumor suppressor p53 protein and phosphorylates human p53 on serine 392 in vitro. Oncogene 1999; 18 : 2690–702. 25. Sakaguchi K, Sakamoto H, Lewis MS, et al. Phosphorylation of serine 392 stabilizes the tetramer formation of tumor suppressor protein p53. Biochemistry 1997; 36 : 10117–24. 26. Craig AL, Blaydes JP, Burch LR, Thompson AM, Hupp TR. Dephosphorylation of p53 at Ser20 after cellular exposure to low levels of non-ionizing radiation. Oncogene 1999; 18 : 6305–12. 27. Li L, Ljungman M, Dixon JE. The human Cdc14 phosphatases interact with and dephosphorylate the tumor suppressor protein p53. J Biol Chem 2000; 275 : 2410–4. 28. Stavridi ES, Chehab NH, Malikzay A, Halazonetis TD. Substitutions that compromise the ionizing radiationinduced association of p53 with 14-3-3 proteins also compromise the ability of p53 to induce cell cycle arrest. Cancer Res 2001; 61 : 7030–3. 29. Lambert PF, Kashanchi F, Radonovich MF, Shiekhattar R, Brady JN. Phosphorylation of p53 serine 15 increases interaction with CBP. J Biol Chem 1998; 273 : 33048–53. 30. Prives C, Manley JL. Why is p53 acetylated? Cell 2001; 107 : 815–8. 31. Gottifredi V, Shieh SY, Prives C. Regulation of p53 after different forms of stress and at different cell cycle stages. Cold Spring Harbor Symp Quant Biol 2000; 65 : 483–8. 32. Momand J, Wu HH, Dasgupta G. MDM2 - master regulator of the p53 tumor suppressor protein. Gene 2000; 242 : 15–29. 33. Grossman SR, Perez M, Kung AL, et al. p300/MDM2 complexes participate in MDM2-mediated p53 degradation. Mol Cell 1998; 2 : 405–15. 34. Kubbutat MH, Ludwig RL, Ashcroft M, Vousden KH. Regulation of Mdm2-directed degradation by the C terminus of p53. Mol Cell Biol 1998; 18 : 5690–8. 35. Pomerantz J, Schreiberagus N, Liegeois NJ, et al. The Ink4a tumor suppressor gene product, p19(Arf), interacts with MDM2 and neutralizes MDM2’s inhibition of p53. Cell 1998;92 : 713–23. 36. Woods DB, Vousden KH. Regulation of p53 function. Exp Cell Res 2001; 264 : 56–66. 37. Zhao RB, Gish K, Murphy M, et al. Analysis of p53-regulated gene expression patterns using oligonucleotide arrays. Genes Dev 2000; 14 : 981–93. |