| |
| Med Sci (Paris). 2002 December; 18(12): 1276–1281. Published online 2002 December 15. doi: 10.1051/medsci/200218121276.Éléments mobiles SINE en phylogénie Dorothée Huchon, Masato Nikaido, and Norihiro Okada Tokyo Institute of Technology, Faculty of Bioscience and Biotechnology, 4259 Nagatsuta-cho, Midori-ku, Yokohama 226-8501, Japon |
Depuis Darwin, les classifications biologiques ont pour but de refléter la phylogénie, c’est-à-dire les relations évolutives entre les espèces. Les premières classifications étaient fondées sur des caractères morpho-anatomiques, paléontologiques ou embryologiques. Cependant, ces caractères semblent limités lorsque les espèces présentent des morphologies très différentes, donc difficilement comparables, comme dans le cas des eucaryotes unicellulaires (→), ou lorsqu’elles ont développé des adaptations similaires de façon indépendante (convergence), comme dans le cas des rongeurs [1]. Depuis le début des années 1960, l’analyse des caractères moléculaires, et en particulier des séquences d’ADN, est apparue comme une approche complémentaire des travaux morphologiques. Presque révolutionnaires, ces études moléculaires ont permis de suggérer de nouvelles hypothèses et de faire progresser les phylogénies précédentes (→→ dans le cas des eucaryotes). (→) m/s 1995, n° 8, p. 1 (→→) m/s 1995, n° 8, p. 1 et 1996, n° 2, p. 1 Cependant, après une phase euphorique au cours de laquelle les études moléculaires semblaient être la solution à toutes les questions phylogénétiques en suspens, certaines contradictions sont apparues. Les phylogénies moléculaires se sont en effet révélées être sensibles à l’échantillonnage taxonomique choisi, ainsi qu’au gène utilisé (→). Si de nombreux progrès ont été réalisés pour comprendre et détecter les artéfacts, des approches phylogénétiques complémentaires apparaissent nécessaires. Récemment, l’étude des sites d’insertion d’éléments transposables tels que les SINE (short interspersed repetitive elements) est apparue comme une méthode phylogénétique puissante, ayant permis d’améliorer considérablement la phylogénie de nombreux groupes taxonomiques tels que les poissons salmonidés, les mammifères cétacés et artiodactyles (revue dans [2, 3]), les primates [4] ou bien encore les plantes crucifères du genre Brassica [5]. Les SINE ont également été utilisés en phylogéographie pour étudier l’origine des populations humaines [6–8]. (→) m/s 1995, n° 8, p. 1 |
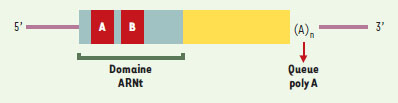
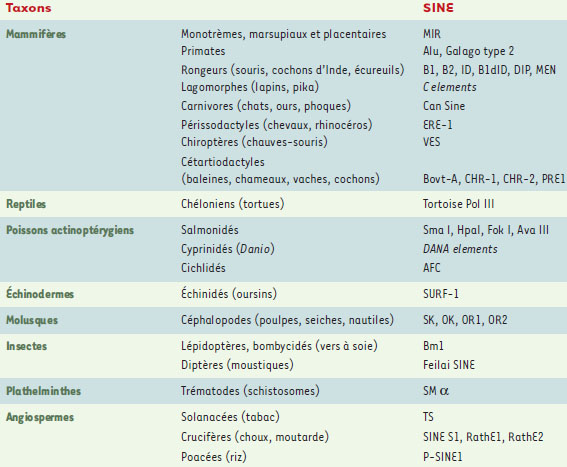
Dynamique des éléments transposables et intérêt phylogénétique Les éléments transposables représentent une composante importante du génome des eucaryotes, puisqu’ils constituent environ 45 % du génome humain, par exemple [9]. Aucun rôle précis n’a pu leur être attribué jusqu’à présent, et ils sont souvent considérés comme des parasites du génome (ADN « égoïste ») ou comme de l’ADN « poubelle ». Toutefois, ces éléments participent activement à l’évolution des génomes en facilitant, par exemple, les remaniements chromosomiques (revue dans [10]). Les éléments transposables sont divisés en deux groupes: les rétrotransposons, ou éléments de classe I, ont un mode de transposition nécessitant le passage par un intermédiaire ARN, qui est ensuite rétrotranscrit en ADN avant d’être inséré dans le génome. Les transposons, ou éléments de classe II, utilisent en revanche un intermédiaire ADN. Les SINE sont des rétrotransposons dérivés principalement des ARN de transfert ou de l’ARN cytoplasmique 7SL (Figure 1). Des familles de SINE ont été identifiées dans de nombreux organismes, invertébrés, vertébrés ou plantes (Tableau I).  | Figure 1. Structure simplifiée d’un SINE. A et B : promoteurs de l’ARN polymérase III qui peuvent être utilisés pour identifier les SINE présents dans un génome, soit en transcrivant in vitro l’ADN génomique par l’ARN polymérase III, soit en effectuant des PCR avec des amorces spécifiques du promoteur de l’ARN polymérase III (voir texte). |
 | Tableau I.Exemples de taxons pour lesquels une ou plusieurs familles de SINE ont été identifiées (d’après [ 2, 11– 13]). |
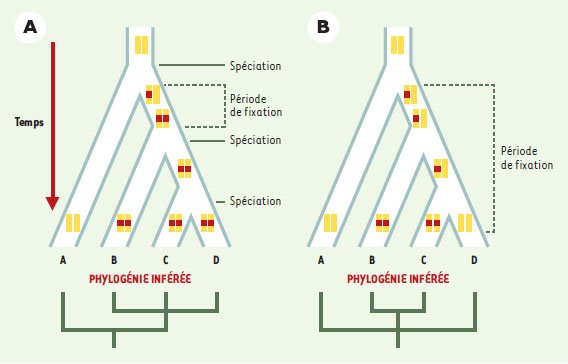
Le mécanisme de transposition des SINE n’est pas encore élucidé: dans la mesure où ils ne codent pas pour les fonctions nécessaires à leur rétrotransposition, les SINE pourraient utiliser la rétrotranscriptase des éléments LINE (long interspersed repetitive elements) (revue dans (→) et [14]). Bien que les mécanismes de multiplication des SINE soient, eux aussi, relativement mal connus, leur distribution dans les génomes révèle deux caractéristiques essentielles pour leur utilisation en phylogénie. Premièrement, les SINE présentent une spécificité d’insertion très faible, puisqu’elle dépend de combinaisons nucléotidiques variées et fréquentes [9, 13, 15, 16]: ils peuvent donc s’insérer à peu près partout dans le génome. Par ailleurs, étant donné la taille importante des génomes, la probabilité qu’un SINE s’insère exactement au même endroit dans le génome de deux espèces différentes peut être considérée comme négligeable [3]. Deuxièmement, il n’existe aucun processus d’élimination des rétrotransposons [2, 4]: les SINE se maintiennent de façon irréversible dans leur site d’insertion, où ils subissent les forces de mutation et de sélection propres à cette région du génome. Pour l’ensemble de ces raisons, la présence d’un SINE à un site donné est un événement qui n’est intervenu qu’une fois au cours de l’évolution. (→) m/s 2001, n° 1, p. 103 Un SINE, à un locus donné, est fixé dans une population (ou une espèce) quand il est présent chez tous les individus de cette population. Sa fixation dépend principalement du hasard qui intervient dans la transmission d’un allèle d’une génération à une autre: selon les conditions, l’allèle peut totalement disparaître de la population (élimination d’un allèle) ou être présent chez tous les individus (fixation d’un allèle). D’après la théorie neutraliste de l’évolution, le temps de fixation dépend de la taille de la population, les fixations étant concentrées aux moments où l’espèce compte peu d’individus, comme à son commencement. L’étude des sites d’insertion des SINE permet d’apporter une information phylogénétique lorsque la fixation d’un SINE à un site donné se situe entre deux événements de spéciation (Figure 2A): dans ce cas, les individus partageant le même SINE à un locus précis ont tous une origine commune. Ainsi dans la Figure 2A, les taxons B, C et D, possédant un SINE commun, sont phylogénétiquement plus proches entre eux qu’ils ne le sont du taxon A. La présence d’un SINE à un locus est un caractère binaire qui ne connaît ni convergence, ni réversion. Elle apparaît donc comme un caractère phylogénétique « parfait » dont la polarité est connue: l’absence de SINE dans un site est le caractère ancestral, et sa présence le caractère dérivé. 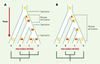 | Figure 2. Impact sur la phylogénie inférée du temps de fixation d’un SINE dans un locus par rapport aux événements de spéciation. L’évolution des allèles à un locus est représentée le long d’un arbre phylogénétique. Le locus considéré est indiqué par une barre verticale jaune, la présence d’un SINE est caractérisée par un rectangle rouge sur le locus. Les événements de spéciation sont indiqués par des flèches et le temps de fixation des allèles par une accolade. A. Le SINE se fixe entre deux événements de spéciation, et sa distribution reflète la phylogénie vraie. B. Le SINE n’est pas fixé lors de la séparation des espèces, et l’étude de sa distribution conduit à une phylogénie erronée. |
|
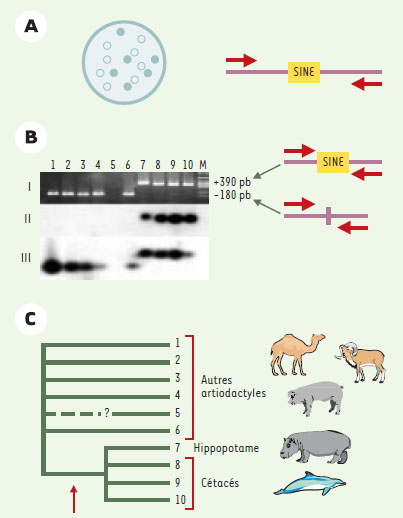
Les principales étapes de la méthode SINE La première étape de toute étude phylogénétique fondée sur l’utilisation des SINE consiste à identifier les éléments pertinents pour répondre à la question phylogénétique posée. Il faut, en effet, trouver une famille ou une sous-famille de SINE dont la multiplication par transposition dans le génome est contemporaine de la diversification des espèces étudiées (Figure 2A). Par exemple, il a été estimé que les éléments MIR (mammalian-wide interspersed repeats), présents dans le génome des mammifères, avaient cessé de se transposer il y a environ 80-100 millions d’années [9]: les MIR ne sont donc pas adaptés pour résoudre les relations de parenté au sein des rongeurs qui se sont diversifiés il y a environ 58 millions d’années. Les données bibliographiques peuvent guider le choix de la famille de SINE à considérer. Ainsi, les SINE associés à un taxon particulier (Tableau I) doivent pouvoir aider à clarifier la phylogénie de ce taxon. En l’absence d’information bibliographique, plusieurs méthodes ont été proposées, comme la transcription in vitro de l’ADN génomique par l’ARN polymérase III: cette approche se fonde sur le fait que les SINE sont dérivés des ARN de transferts et possèdent donc les promoteurs spécifiques de l’ARN polymérase III (Figure 1). L’ARN obtenu par transcription est ensuite utilisé comme sonde pour cribler une banque génomique de l’espèce concernée [17]. Une seconde méthode consiste à effectuer des PCR en utilisant des amorces spécifiques du promoteur de l’ARN polymérase III (A box et B box) présent dans la séquence des SINE (Figure 1); les produits de PCR obtenus sont utilisés par la suite pour cribler une banque génomique [12]. Une fois la famille de SINE pertinente caractérisée, la mise en évidence de leurs sites d’insertion se déroule en cinq étapes (Figure 3). Pour contenir le maximum d’information phylogénétique, les espèces choisies doivent être les moins divergentes au sein du groupe étudié: ainsi, la recherche de SINE dans le génome de l’espèce A (Figure 2) ne permettra pas de résoudre les relations entre B, C, et D, et il vaut mieux choisir l’espèce C ou D. Une connaissance a priori de la phylogénie est donc préférable. 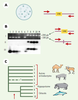 | Figure 3. Différentes étapes de la méthode SINE. A. Construction d’une banque de gènes, criblage de la banque grâce à une sonde caractéristique de la famille de SINE étudiée, séquençage des clones positifs et définition d’amorces (représentées par des flèches) dans les régions flanquant les SINE identifiés. B. Données confirmant le regroupement de l’hippopotame avec les cétacés (locus HIP4, [18]). I. Réaction de PCR avec les amorces créées, afin de déterminer la distribution du SINE dans le groupe étudié. Les bandes de grandes taille (taxons 7-10) correspondent à des fragments contenant un SINE; celles de petite taille (taxons 1-4 et 6) à des fragments sans SINE. II. Validation du résultat de la PCR par Southern blot utilisant la séquence du SINE comme sonde. III. Southern blot utilisant la séquence des régions flanquant le SINE comme sonde. Ces deux dernières étapes permettent de vérifier que les produits de PCR obtenus sont bien homologues et ne correspondent pas à des artéfacts. C. Arbre phylogénétique inféré à partir du locus HIP4. Les taxons 7 à 10 sont regroupés car ils partagent un SINE, la position du taxon 5 ne peut être inférée, l’absence de produit de PCR ne permettant pas de déterminer l’absence ou la présence de SINE à ce locus. Taxons considérés: 1: Camelus bactrianus (chameau); 2: Sus scrofa (cochon); 3: Axis axis (cerf axis); 4: Giraffa camelopardalis (girafe); 5: Ovis aries (mouton); 6: Bos taurus (vache); 7: Hippopotamus amphibius (hippopotame); 8: Megaptera novaeangliae (baleine à bosses); 9: Berardius bairdii (baleine à bec); 10: Globicephala macrorhynchus (globicéphale); M: Marqueur de taille. |
Pour élucider complètement les relations de parenté entre plusieurs espèces, il est nécessaire d’identifier au moins un SINE caractéristique pour chaque nœud de l’arbre phylogénétique. |
Avantages et limites de la méthode SINE L’intérêt de cette approche est qu’elle ne connaît pas d’artéfacts lors de la reconstruction des relations phylogénétiques. En revanche, le recours à l’analyse de séquences conduit les méthodes de reconstruction à être sensibles aux hétérogénéités de taux de substitution ou de composition en bases entre espèces. Dans le premier cas, les espèces ayant des taux de mutations élevés seront placées de façon artificielle à la base des arbres phylogénétiques: c’est le phénomène d’attraction des longues branches (→) [19]. Dans le second cas, les taxons ayant des compositions en bases similaires auront tendance à être regroupés artificiellement [20]. Un autre avantage de la méthode SINE est qu’elle ne nécessite pas l’utilisation de groupe externe pour polariser l’arbre phylogénétique obtenu; or les groupes externes sont parfois sources de problèmes dans l’enracinement des arbres [21]. Le fait que l’absence de SINE soit le caractère ancestral permet à lui seul d’orienter l’ordre de branchement des espèces. La méthode SINE possède cependant ses limites. Tout d’abord, quand les séquences deviennent trop divergentes (à partir de 20-30 % de divergence), il devient difficile de définir des amorces de PCR communes à l’ensemble des espèces étudiées: cela se traduit par une absence de bande dans le produit de réaction obtenu. C’est le cas du mouton (espèce 5) dans la Figure 3B. Il est remarquable que l’absence de données n’affecte pas les conclusions obtenues: ainsi, dans la Figure 3B, le regroupement de l’hippopotame (espèce 7) avec les cétacés (espèces 8, 9 et 10) n’est pas remis en cause. Seule la position du mouton reste ambiguë, et il est impossible de savoir, à travers l’étude de ce locus, si le mouton appartient ou non au groupe cétacés + hippopotame. Toutefois, l’utilisation d’autres SINE a montré que le mouton se regroupait avec les ruminants (cerf et vache), et non avec l’hippopotame ou les cétacés [18]. Les problèmes de divergence entre séquences limitent la méthode SINE à des questions phylogénétiques concernant des spéciations ayant eu lieu après 75-100 millions d’années (ce qui correspond au crétacé supérieur), ou plus récemment [3] si les taxons considérés présentent de forts taux de substitution, comme dans le cas des divergences entre familles de rongeurs. La seconde limite de la méthode SINE trouve sa source dans la possibilité d’un polymorphisme ancestral au locus considéré. En effet, lorsque la divergence des taxons étudiés se produit avant la fixation du SINE à ce locus, la reconstruction peut conduire à une phylogénie erronée. Ainsi, dans la Figure 2B, les populations à l’origine des espèces B, C et D étaient polymorphes. L’allèle contenant le SINE s’est fixé dans les espèces B et C, mais pas dans l’espèce D chez laquelle il a été éliminé. La phylogénie reconstruite par la méthode SINE isole donc l’espèce D de B et C, alors qu’en réalité C est plus proche de D que de B. De tels problèmes peuvent se produire lorsque les espèces se sont diversifiées dans un laps de temps très court, pendant la période de fixation de l’élément SINE. Le moyen de détecter cet artéfact est d’identifier plusieurs SINE caractéristiques de chaque branchement de l’arbre phylogénétique. En cas de polymorphisme ancestral, il est attendu que les locus obtenus soient incongruents entre eux, c’est-à-dire qu’ils conduisent à des images phylogénétiques différentes. L’ordre de branchement des espèces se traduit alors par une multifurcation. (→) m/s 2002, n° 11, p.1146 |
Exemples d’applications phylogénétiques chez les cétartiodactyles Les premiers résultats obtenus avec les SINE ont permis de confirmer des résultats moléculaires inattendus, comme le placement des cétacés au sein des artiodactyles [22] ou le surprenant groupement de l’hippopotame avec les baleines [18]. Les études morphologiques considéraient quant à elles que les cétacés et les artiodactyles formaient deux ordres distincts de mammifères [23], et regroupaient les hippopotames avec les cochons [24]. Récemment, la méthode SINE a au contraire permis de conforter les résultats morphologiques divisant les cétacés en odontocètes (cétacés à dents) et mysticètes (cétacés à fanons), alors que les analyses de séquences ne vérifiaient pas l’origine unique des odontocètes [25]. Il y a donc fort à attendre de l’application future de cette méthode à d’autres groupes taxonomiques dont la phylogénie reste énigmatique. |
D.H. remercie l’association Sciencescope pour lui avoir suggéré l’écriture de cet article, Frédéric Delsuc pour ses commentaires pendant la rédaction et l’expert de médecine/ sciences pour ses remarques. Le travail de D.H. était financé par une bourse Lavoisier attribuée par le ministère des Affaires étrangères.
|
1. Jaeger JJ. Rodent phylogeny: new data and old problems. In: Benton MJ, ed. The phylogeny and classification of the Tetrapods. Oxford: Clarendon Press, 1988 : 177–99. 2. Shedlock AM, Okada N. SINE insertions: powerful tools for molecular systematics. BioEssays 2000; 22: 148–60. 3. Shedlock AM, Milinkovitch MC, Okada N. SINE evolution, missing data, and the origine of whales. Syst Biol 2000; 49: 808–17. 4. Hamdi H, Nishio H, Zielinski R, Dugaiczyk A. Origin and phylogenetic distribution of Alu DNA repeats: irreversible events in the evolution of primates. J Mol Biol 1999; 289: 861–71. 5. Tatout C, Warwick S, Lenoir A, Deragon JM. SINE insertions as clade markers for wild crucifer species. Mol Biol Evol 1999; 16: 1614–21. 6. Batzer MA, Stoneking M, Alegria-Hartman M, et al. African origin of human-specific polymorphic Alu insertions. Proc Natl Acad Sci USA 1994; 91: 12288–92. 7. Novick GE, Novick CC, Yunis J, et al. Polymorphic alu insertions and the Asian origin of native American populations. Hum Biol 1998; 70: 23–39. 8. De Pancorbo MM, Lopez-Martinez M, Martinez-Bouzas C, et al. The Basques according to polymorphic Alu insertions. Hum Genet 2001; 109: 224–33. 9. International Human Genome Sequencing Consortium. Initial sequencing and analysis of the human genome. Nature 2001; 409: 860–921. 10. Makalowski W. SINEs as a genomic scrap yard: an essay on genomic evolution. In: Maraia RJ, ed. The impact of short interspersed elements (SINEs) on the host genome. Austin: R.G. Landes Company, 1995 : 81–104. 11. Okada N. SINEs: short interspersed repeated elements of the eukaryotic genome. Trends Ecol Evol 1991; 6: 358–61. 12. Borodulina OR, Kramerov DA. Wide distribution of short interspersed elements among eukaryotic genomes. FEBS Lett 1999; 457: 409–13. 13. Lenoir A, Lavie L, Prieto JL, et al. The evolutionary origin and genomic organization of SINEs in Arabidopsis thaliana. Mol Biol Evol 2001; 18: 2315–22 14. Okada N, Hamada M, Ogiwara I, Ohshima K. SINEs and LINEs share common 3’ sequences: a review. Gene 1997; 205: 229–43. 15. Jurka J. Sequence patterns indicate an enzymatic involvement in integration of mammalian retroposons. Proc Natl Acad Sci USA 1997; 94: 1872–7. 16. Tatout C, Lavie L, Deragon JM. Similar target site selection occurs in integration of plant and mammalian retroposons. J Mol Evol 1998; 47: 463–70. 17. Endoh H, Okada N. Total DNA transcription in vitro: a procedure to detect highly repetitive and transcribable sequences with tRNA-like structures. Proc Natl Acad Sci USA 1986; 83: 251–5. 18. Nikaido M, Rooney AP, Okada N. Phylogenetic relationships among cetartiodactyls based on insertions of short and long interspersed elements: hippopotamuses are the closest extant relatives of whales. Proc Natl Acad Sci USA 1999; 96: 10261–6. 19. Philippe H, Laurent J. How good are deep phylogenetic trees? Curr Opin Genet Dev 1998; 8: 616–23. 20. Mooers AØ, Holmes EC. The evolution of base composition and phylogenetic inference. Trends Ecol Evol 2000; 15: 365–9. 21. Sullivan J, Swofford DL. Are guinea pigs rodents? The importance of adequate models in molecular phylogenetics. J Mammal Evol 1997; 4: 77–86. 22. Shimamura M, Yasue H, Ohshima K, et al. Molecular evidence from retroposon that whales form a clade within even-toed Ungulata. Nature 1997; 388: 666–70. 23. McKenna MC, Bell SK. Classification of mammals above the species level. New York: Columbia University Press, 1997 : 632 p. 24. Montgelard C, Ducroq S, Douzery E. What is a suiforme (Artiodactyla)? Mol Phylogenet Evol 1998; 9: 528–32. 25. Nikaido M, Matsuno F, Hamilton H, et al. Retroposon analysis of major cetacean lineages: the monophyly of toothed whales and the paraphyly of river dolphins. Proc Natl Acad Sci USA 2001; 98: 7384–9. |