Le maintien de l’intégrité du génome est essentiel à la transmission fidèle du patrimoine génétique. Les rayonnements ultraviolets (UV) contenus dans la lumière solaire modifient la structure de l’ADN et peuvent conduire à l’introduction de mutations. Ainsi, plus de la moitié des cancers de la peau d’origine kératinocytaire (les carcinomes baso- et spinocellulaires, CBC, CSC) se développant dans les zones corporelles photo-exposées et portent des mutations caractéristiques des rayons UV (transitions C vers T, et mutations en tandem CC vers TT) dans le gène suppresseur de tumeur P53 [ 1]. Les CBC et les CSC occupent le premier rang (environ 30 %) de toutes les tumeurs humaines et leur incidence augmente d’au moins 10 % chaque année [ 2]. L’étude des mécanismes moléculaires dédiés à la protection de l’ADN est donc essentielle pour que progressent la prévention et le traitement des cancers cutanés photo-induits.
Les lésions introduites dans l’ADN par les UVB (280-315 nm) sont en majorité des cis-syn dimères de cyclobutane pyrimidine (CPD), et des pyrimidine 6-4 pyrimidone photoproduits (6-4 PP). Ces lésions sont formées sur deux pyrimidines [cytosine (C) ou thymine (T)] adjacentes (sur le même brin d’ADN). À dose d’UVB égale, huit CPD (ciblant surtout les séquences TT) sont formés pour un 6-4 PP (ciblant les séquences CC) [ 3]. CPD et 6-4 PP distordent la double hélice d’ADN d’un angle de 7 à 30° et 44°, respectivement [ 4]. La courbure de l’ADN constitue le signal d’alarme conduisant à l’élimination des CPD et des 6-4 PP. La réparation de l’ADN par excision de nucléotide (nucleotide excision repair, NER) est le mécanisme majeur de l’élimination de ces lésions. Au cours des dix dernières années, les différentes étapes participant à la NER ont été très étudiées et la plupart des gènes et des protéines constituant les complexes de réparation ont été identifiés [ 5]. L’importance de la NER est attestée par l’existence de maladies humaines graves dites « de réparation » dont le prototype est le xeroderma pigmentosum (XP), une maladie rare (1/500 000 naissances dans notre région du globe), à transmission autosomique et récessive. Tous les patients atteints de XP présentent une forte sensibilité aux UV, accompagnée dès le plus jeune âge de nombreuses lésions cancéreuses (CBC et CSC). Chez certains patients, ces manifestations cutanées s’associent à des anomalies du développement et neurologiques de sévérité variable. L’hétérogénéité clinique de la maladie est liée à l’existence de sept groupes de complémentation XP « classiques » (XP-A à XP-G), caractérisés chacun par l’altération d’une étape distincte de la NER. L’étude des cellules de patients atteints de xeroderma pigmentosum a fortement contribué à la dissection des différentes étapes de la NER [ 6].
Le schéma classique de la NER est constitué de l’enchaînement des étapes suivantes.
- Reconnaissance de la distorsion de l’ADN ; les lésions du brin transcrit d’un gène actif sont réparées « préférentiellement » par transcription coupled repair (TCR) suivant l’arrêt de l’ARN polymérase II en cours de progression. Les lésions du brin non transcrit des gènes actifs, ou celles des régions inactives du génome sont réparées par global genome repair (GGR). En fonction du type de lésion dans l’ADN et des systèmes d’étude, les protéines intervenant dans cette étape peuvent être soit XPA et RPA (replication protein A), soit XPC, soit XPC et XPE.
- L’étape de reconnaissance est suivie par le recrutement d’un complexe protéique contenant le facteur de transcription de base TFIIH. Les hélicases XPB et XPD contenues dans TFIIH déroulent la double hélice d’ADN en présence d’ATP pour former une « bulle ».
- L’identité de la lésion est ensuite vérifiée, le complexe stabilisé.
- L’intervention de deux endonucléases simple brin, XPG en 3’ de la lésion, puis XPF-ERCC1 en 5’ de la lésion permet l’incision puis l’élimination d’un oligonucléotide simple brin de 27 à 34 bases.
- La réplication fidèle de l’ADN simple brin par les ADN polymérases e et d comble la brèche de façon stable.
- Finalement, l’ADN néosynthétisé est ligaturé par l’ADN ligase.
L’ordre séquentiel (ou non, selon les auteurs) dans lequel les différentes protéines de la NER interviennent à partir de l’étape de reconnaissance a fait l’objet de nombreux travaux. En particulier, l’identité des protéines responsables de la reconnaissance des différentes lésions dans l’ADN est le sujet d’un débat, parfois lié aux conditions expérimentales [ 7].
Le type de lésion, CPD ou 6-4PP, dans l’ADN semble jouer un rôle primordial dans le recrutement des protéines de reconnaissance et le début de la NER. Le rôle des protéines XPA, RPA, XPC et XPE a été plus particulièrement étudié. On a longtemps pensé que XPA, une protéine à doigts de zinc présentant une forte affinité pour les lésions dans l’ADN, pouvait être « la » protéine de reconnaissance [ 8]. Des expériences in vitro ont en effet indiqué que la vitesse de réparation in vitro d’un ADN double brin synthétique porteur d’une lésion 6-4 PP (T<>T) est 5 fois plus rapide si les protéines XPA et RPA sont ajoutées avant XPC que dans la situation inverse [ 9]. Pour les tenants de ce modèle XPA-RPA first, XPC, dont l’affinité pour la lésion est médiocre dans ces conditions expérimentales, jouerait un rôle secondaire ou accessoire dans la reconnaissance.
D’autres travaux ont montré que la présence d’un excès d’ADN « compétiteur », dépourvu de lésion accroît très fortement l’affinité de XPC pour les 6-4 PP [ 10]. Ces conditions sont plus proches de la réalité physiologique puisque, dans la cellule, le nombre de lésions induites par un flux d’UV de 1 joule/m2 est seulement de 2,4 x 10- 5 lésions par 1 000 pb d’ADN [ 11] et n’est pas, comme in vitro, d’une lésion par fragment d’ADN d’une centaine de paires de bases. En présence d’ADN compétiteur, l’analyse par reconstruction séquentielle in vitro des différentes étapes de NER, a permis de proposer XPC comme « la » protéine de reconnaissance des 6-4 PP. La pertinence du modèle in vitro XPC first a été étayée in vivo par la technique d’irradiation locale à travers un filtre de polycarbonate porteur de pores de 5 mm de diamètre. De cette façon, seule une petite surface cellulaire en regard des pores est irradiée. XPC est alors recrutée au niveau des sites d’irradiation, y compris en l’absence de XPA (dans des cellules du groupe de complémentation XP-A). En miroir, XPA ne se concentre pas au niveau des sites d’irradiation en l’absence de XPC (dans des cellules du groupe de complémentation XP-C), indiquant le caractère primordial et obligatoire de l’intégrité de XPC pour la formation du complexe d’incision [ 12]. Mais d’autres expériences in vitro ont montré une mauvaise affinité de la protéine XPC pour les CPD (de type T<>T), suggérant l’intervention du complexe protéique XPE (composé des protéines DDB1/DDB2). En effet, DDB2 stimule in vitro la réparation des lésions CPD (T<>T), à l’origine de moins de distorsions que les 6-4 PP [ 13]. DDB2 pourrait contribuer au remaniement de la chromatine et aider à la reconnaissance par XPC des lésions source de peu de distorsions, probablement en amplifiant localement l’altération de la structure de l’ADN. Le modèle XPC first s’en trouve ainsi affiné. XPC serait la protéine de reconnaissance, agissant seule et en premier pour les 6-4 PP, et de concert avec le complexe XPE pour les CPD.
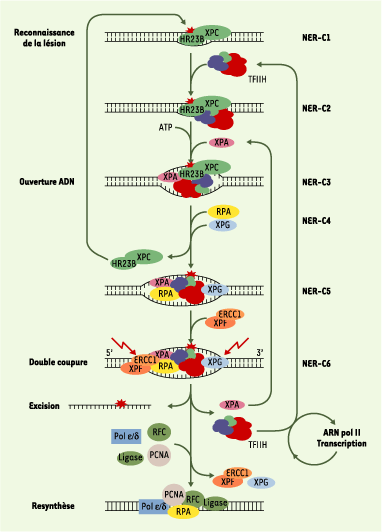
| Figure 1. XPC, complexé à la protéine HR23B, interagit avec la structure formée par l’ADN endommagé (complexe NER-C1). L’entrée de TFIIH forme le complexe NER-C2. En présence d’ATP, les hélicases XPD et XPB contenues dans TFIIH déroulent la molécule d’ADN, permettant les associations séquentielles de XPA, RPA et XPG et conduisant à la formation du complexe NER-C5. L’entrée de XPG catalyse le départ et le recyclage de XPC-HR23B. Avec l’entrée de l’endonucléase XPF-ERCC1 est formé le complexe d’incision NER-C6 qui catalyse l’excision de l’oligonucléotide portant la lésion, le relargage de XPA et TFIIH. TFIIH est ainsi rendu disponible pour la formation de nouveaux complexes d’incision. En l’absence de dommage dans l’ADN, TFIIH peut aussi être re-associé avec l’ARN polymérase II. RPA, XPG et XPF-ERCC1 restent associés à l’ADN simple brin restant jusqu’à l’arrivée des facteurs de re-synthèse de l’ADN avec lesquels RPA reste associé (d’après [
15]). |