Il y a maintenant dix ans, le groupe de V. Ambros décrivait lin-4, un gène de C. elegans ne codant pas pour une protéine mais pour un ARN d’environ 21-23 nucléotides (nt) dont l’invalidation affectait de façon très importante le développement du ver [ 1]. D’abord perçu comme « une bizarrerie » propre à C. elegans, des petits ARN apparentés à lin-4 sont maintenant décrits aussi bien chez A. thaliana (~ 20), D. melanogaster (~ 70), C. elegans (~ 100), M. musculus
(~ 120), H. sapiens (~ 130). De façon remarquable, certains d’entre eux sont très conservés au cours de l’évolution, suggérant une fonction biologique importante. Les membres de cette nouvelle classe d’ARN n’ayant pas de fonction codante ont été collectivement appelés microARN ou miARN. Bien que notre connaissance de l’étendue de ce microcosme d’ARN soit encore balbutiante, on estime actuellement qu’il en existe au minimum 250 chez l’homme, un nombre correspondant à ~ 1 % de l’ensemble des gènes codant pour des protéines et identifiés à ce jour (pour revue, voir [ 2, 3]).
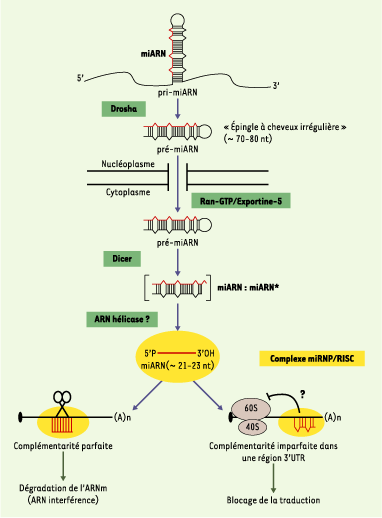
Les mécanismes moléculaires de la biosynthèse des miARN sont relativement bien compris. Apparemment, tous les miARN sont synthétisés sous forme d’ARN précurseurs de grande taille dont la maturation fait intervenir au moins deux protéines apparentées à l’ARNase III : Drosha et Dicer [ 4, 5] (→). Drosha intervient initialement dans le nucléoplasme, où elle convertit un transcrit primaire (ou pri-miARN) en une structure en épingle à cheveux irrégulière d’environ 70 nt (ou pré-miARN). Ce pré-miARN est par la suite pris en charge par Dicer qui façonne un intermédiaire de maturation transitoire, nommé miARN:miARN*, dont un seul des deux brins formera le microARN mature (généralement c’est le brin dont l’extrémité 5’ est la plus faiblement appariée) [ 6, 7]. D’autres facteurs participent vraisemblablement à la biosynthèse des miARN, notamment l’exportine 5 qui assure le transport nucléocytoplasmique du pré-miARN (Figure 1). La biosynthèse des miARN n’est donc pas sans rappeler celle des petits ARN interférents (ou siARN) impliqués dans les phénomènes d’ARN interférence (ou ARNi) qui, eux aussi, sont produits par Dicer à partir de longs ARN double-brins [ 8].
(→) m/s 2004, n° 1, p. 26
| Figure 1.
Biosynthèse et modalités d’action des miARN. La maturation des miARN fait intervenir deux enzymes, Drosha et Dicer, qui, à partir d’un ARN précurseur en tige boucle imparfaite produisent un ARN simple brin d’environ 21-23 nt: le microARN. Ce dernier s’associe alors avec des protéines pour former le complexe RISC/miRNP. Ce complexe RISC/miRNP - dont la composition protéique commence à être élucidée (eiF2C2, Gemin3, Gemin4, FMRP…) - s’apparie avec des ARNm via le miARN. Si la complémentarité est imparfaite, il y a blocage de la traduction alors que si la complémentarité est parfaite, il y a destruction de l’ARNm cible (c’est l’ARN interférence). |
Les miARN, une fois libérés des pré-miARN, ou au cours de leur processus de maturation à partir du pré-miARN, interagissent avec des protéines spécifiques pour former un complexe ribonucléoprotéique stable nommé RISC-miRNP (RNA-induced silencing complex-micro-ribonucleoprotein). Au sein de ce complexe, le miARN, par un jeu d’appariement de bases, interagit avec des ARNm. Les modalités d’action de RISC-miRNP sont relativement mal comprises, mais semblent contrôlées par la qualité du duplex miARN:ARNm mis en jeu. Si cette complémentarité est parfaite, RISC-miRNP induit alors un clivage au milieu de l’hybride miARN:ARNm entraînant la dégradation rapide de l’ARNm. Ainsi, non seulement les miARN partagent la même voie de biosynthèse que les siARN mais, tout comme eux, ils peuvent aussi promouvoir l’ARN interférence. En revanche, si cette complémentarité est irrégulière (présence dans le duplex de nucléotides non appariés), on observe alors l’inhibition de la traduction de l’ARNm cible sans pour autant que sa stabilité soit affectée. De tels appariements imparfaits prennent place généralement dans la région 3’ non traduite des ARNm (ou 3’UTR) et les mécanismes moléculaires du blocage de la traduction restent à élucider (Figure 1).
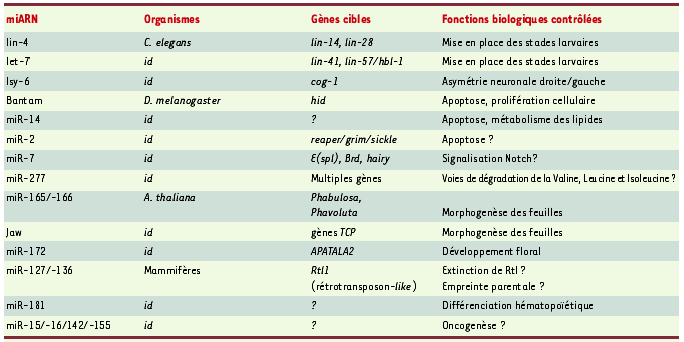
Alors que leur nombre ne cesse de croître, la fonction biologique des miARN est très mal comprise. Chez les plantes, nombreux sont les miARN présentant des complémentarités parfaites avec des ARNm ; c’est notamment le cas de ceux codant pour des facteurs de transcription impliqués dans le développement [ 9]. En revanche, chez les métazoaires, la recherche des gènes que contrôlent les miARN reste plus problématique. À l’exception de miR-127 et miR-136, dont on soupçonne l’implication dans l’extinction de l’expression d’un gène soumis à l’empreinte génomique parentale [ 10] (Tableau I), aucun des miARN identifiés à ce jour ne présente de complémentarité parfaite avec des ARNm cellulaires. Il est donc probable que la majorité de ces miARN interviennent dans l’inhibition de la traduction plutôt que pour promouvoir l’ARNi, au contraire de ce qui se passe chez les plantes. C’est par exemple le cas pour lin-4 et let-7 qui inhibent la traduction des ARNm des gènes hétérochroniques 1 (lin-14, lin-28, lin-41 et lin-57), qui sont requis pour les transitions temporelles des stades larvaires au cours du développement du ver [ 11]. De même, la mise en place de l’asymétrie neuronale (droite-gauche) chez C. elegans fait aussi intervenir le miARN lsy dont la cible est l’ARNm cog [ 12]. Chez la drosophile, plusieurs miARN (miR-14, bantam, miR-2) contrôlent la prolifération et/ou la différenciation cellulaires en modulant notamment l’expression de gènes proapoptotiques comme hid (Tableau I) [ 13]. Chez les mammifères, bien que sa cible reste à identifier, miR-181 interviendrait lors du processus de différenciation hématopoïétique [ 14]. Il ne faut pas non plus écarter l’hypothèse d’un dérèglement de l’expression des miARN dans les processus d’oncogenèse, et certaines translocations chromosomiques associées à des leucémies affectent des locus géniques comportant les gènes miR-142, miR-15, miR-16, miR-155 (pour revue, voir [ 15, 16]).
| Tableau I.
Exemples de microARN dont la fonction biologique et/ou les cibles ARNm sont caractérisées.
|
Identifier le répertoire complet des ARNm cibles des miARN représente un réel défi - à la fois expérimental et conceptuel - car les appariements imparfaits mis en jeu lors du blocage de la traduction sont difficiles à caractériser, et même si certains ARNm cibles ont été récemment prédits par des approches in silico [ 17– 19], il convient maintenant de développer des approches biochimiques et génétiques pour valider formellement ces interactions. De plus, un même miARN peut intervenir sur des ARNm différents ; l’inverse est également vrai, une même région 3’UTR peut se lier à plusieurs miARN différents. Appréhender cette combinatoire d’interactions ARN:ARN et les réseaux de régulation sous-jacents ajoute un autre niveau de complexité qu’il faudra décrypter pour apprécier pleinement la fonction biologique de cette nouvelle famille d’ARN non codants. Des phénomènes épigénétiques de « mise en veille » de gènes faisant intervenir des mécanismes apparentés à l’ARNi ont été récemment décrits, notamment chez les plantes et S. pombe [ 20, 21]. L’intervention de miARN dans de tels processus chez les vertébrés reste une question ouverte qu’il conviendra aussi d’éclaircir. En conclusion, la découverte des miARN - et plus généralement celle de la machinerie de l’ARNi - a révolutionné notre vision du contrôle de l’expression des gènes et nous rappelle, s’il en était encore besoin, que l’étude du monde fascinant des ARN non codants ne fait que commencer…