| |
| Med Sci (Paris). 2004 August; 20(8-9): 767–772. Published online 2004 August 15. doi: 10.1051/medsci/2004208-9767.Régulation de l’expression des séquences répétées et interférence par l’ARN Valérie Robert and Alain Bucheton* Institut de Génétique humaine, CNRS, 34396 Montpellier Cedex 5, France |
Une partie importante du génome des eucaryotes est constituée de séquences répétées. Celles-ci résultent, en majorité, de l’invasion du génome par des éléments transposables et des rétrovirus endogènes, séquences ayant la capacité de se multiplier. L’expression de ces séquences peut provoquer leur transposition qui est souvent délétère pour l’hôte. Ainsi, des cancers et des maladies génétiques peuvent être associés à l’insertion de ces éléments à proximité ou dans certains gènes. La transposition de ces séquences doit donc être maintenue à un taux compatible avec la survie des espèces. L’expression des éléments transposables est étroitement contrôlée aussi bien au niveau transcriptionnel que post-transcriptionnel. Ce n’est que très récemment que les mécanismes de ce contrôle ont commencé à être découverts. Conservés au cours de l’évolution, ils ont été mis en évidence chez les plantes, le champignon Neurospora crassa, le nématode Caenorhabditis elegans (C. elegans), la mouche du vinaigre Drosophila melanogaster (D. melanogaster), et dans des systèmes cellulaires dérivés des mammifères. Nous limiterons cette synthèse à C. elegans et D. melanogaster, organismes de référence chez lesquels de nombreuses études ont été réalisées ces dernières années. |
Régulation de l’expression des séquences répétées au niveau transcriptionnel Extinction transcriptionnelle des séquences multiples chez D. melanogaster La répression de transgènes multiples chez D. melanogaster a été étudiée, entre autres, grâce à miniwhite [
1], un transgène dont l’expression peut être détectée en observant directement la couleur de l’œil. Les individus porteurs d’un allèle white sauvage ont les yeux rouges, alors que ceux qui en sont dépourvus ont les yeux blancs. L’insertion d’une copie unique du transgène miniwhite dans le génome de mouches mutantes pour le gène white produit des mouches aux yeux rouges, ce qui indique que miniwhite est normalement exprimé. Mais lorsque plusieurs copies du transgène sont présentes, les mouches ont les yeux blancs. Le transgène n’est donc plus exprimé dans ces conditions. Au moins deux gènes de D. melanogaster sont impliqués dans l’extinction des copies multiples de miniwhite. Le premier code pour la protéine Enhancer of zeste (E[z]) qui appartient au groupe Polycomb regroupant des répresseurs qui agissent en complexes multimoléculaires maintenant la chromatine dans un état transcriptionnellement inactif [
2]. Récemment, trois études indépendantes (pour revue, voir [ 2]) ont montré que E(z) a une activité méthylase. De telles activités enzymatiques sont essentielles au cours de la mise en place des états réprimés ou déréprimés de la chromatine. Elles agissent en modifiant l’extrémité aminoterminale des histones [
3], protéines associées à la chromatine. Le second gène requis pour l’extinction des copies multiples de miniwhite code pour la protéine HP1, dont on observe, sur des chromosomes polytènes de glandes salivaires de larves, qu’elle se fixe sur les transgènes miniwhite éteints. HP1 est un composant de la chromatine. Chez les mammifères, on a démontré que HP1 est nécessaire à la méthylation de la lysine 9 de l’histone H3, une modification chimique associée à un état chromatinien transcriptionnellement inactif. L’identification de gènes impliqués dans la répression des copies multiples du transgène miniwhite suggère que le contrôle des séquences répétées a lieu, au moins en partie, au niveau transcriptionnel. Cette observation est supportée par d’autres études. Ainsi, lorsque des copies multiples du transgène w-Adh, contenant le gène Adh (alcohol dehydrogenase) sous le contrôle de séquences régulatrices du gène white, sont insérées dans le génome de la mouche, l’expression du gène endogène Adh (normalement non répété dans le génome) et du gène Adh transgénique est réprimée [
4]. Cette extinction est d’autant plus forte que le nombre de copies du transgène w-Adh augmente. L’efficacité de cette régulation est affectée chez les individus portant à l’état hétérozygote des mutations dans les gènes Polycomb ou polycomblike, deux membres du groupe Polycomb. On observe, sur les chromosomes polytènes, que les protéines codées par ces deux gènes sont liées aux sites où sont insérés les transgènes éteints [4]. Chez C. elegans, l’extinction des transgènes a également une composante transcriptionnelle L’injection d’ADN chez C. elegans conduit à la formation de molécules extrachromosomiques composées de plusieurs centaines de copies de la séquence injectée répétées en tandem [
5]. L’expression des séquences portées par les molécules extrachromosomiques est, en général, éteinte dans la lignée germinale, alors qu’elle ne l’est que rarement dans le soma. Les modifications des histones au niveau de ces molécules extrachromosomiques éteintes ont été étudiées par des expériences d’immunochimie [
6]. Ces molécules fixent des anticorps capables de reconnaître spécifiquement la forme de l’histone H3 méthylée en position lysine 9. L’extinction des transgènes semble donc avoir une composante transcriptionnelle, comme le confirme l’identification d’un certain nombre de gènes impliqués dans ce mécanisme. Ainsi, par analogie avec ce qui est connu chez D. melanogaster, le rôle de HP1 dans l’extinction des transgènes a été étudié. Après inactivation du gène codant pour cette protéine, les séquences présentes dans les molécules extra-chromosomiques sont exprimées. HP1 est donc nécessaire à l’extinction des transgènes [
7]. Cette répression est aussi dépendante de gènes appelés mes-2 et mes-6, qui appartiennent au groupe Polycomb [
8]. |
Régulation de l’expression des séquences répétées au niveau post-transcriptionnel Chez C. elegans, la régulation des éléments transposables et l’extinction des transgènes sont apparentées entre elles et à l’interférence par l’ARN Chez C. elegans, les éléments transposables peuvent s’amplifier dans le soma, mais pas dans la lignée germinale [
9,
10]. Cette observation suggère fortement qu’une régulation existe, maintenant le nombre de leurs copies à un niveau réduit dans la lignée germinale. Certains gènes impliqués dans cette régulation ont été identifiés par mutagenèse [
11]. Ils peuvent être classés en deux catégories : ceux qui ne sont impliqués que dans la régulation des éléments transposables, et ceux qui sont également impliqués dans l’extinction des transgènes [
12,
13] et dans un phénomène de régulation de l’expression des gènes connu sous le nom d’interférence par l’ARN (voir Encadré). Chez C. elegans, la caractérisation du mécanisme de l’ARNi a permis de commencer à comprendre comment les éléments transposables sont contrôlés. Ainsi, des ARN double brin et des ARN guides correspondant aux séquences des éléments transposables ont été mis en évidence dans les souches de C. elegans chez lesquelles les éléments transposables sont régulés dans la lignée germinale [
21]. Les ARN double brin caractérisés correspondent pour l’essentiel à des séquences longues d’une dizaine de paires de bases localisées aux extrémités des éléments transposables. Lorsque ces séquences sont insérées dans un transgène s’exprimant normalement dans la lignée germinale, l’expression de ce transgène est éteinte. Cette extinction est dépendante des gènes impliqués dans le contrôle des éléments transposables, ce qui suggère que les ARN double brin identifiés sont effectivement actifs dans le mécanisme de ce contrôle. L’interférence par l’ARN En 1998, Fire et al. [
14] ont montré qu’il est possible, chez C. elegans, d’inhiber post-transcriptionnellement l’expression d’un gène en injectant dans l’animal des molécules d’ARN double brin ayant une séquence commune avec le gène ciblé [
15]. Cette inhibition est spécifique puisque seuls les gènes présentant de longues séquences communes avec l’ARN double brin sont éteints. Cette régulation a été appelée interférence par l’ARN (ARNi). L’étude intensive de l’ARNi chez C. elegans et D. melanogaster a permis de réaliser des progrès remarquables et un modèle peut maintenant être proposé [
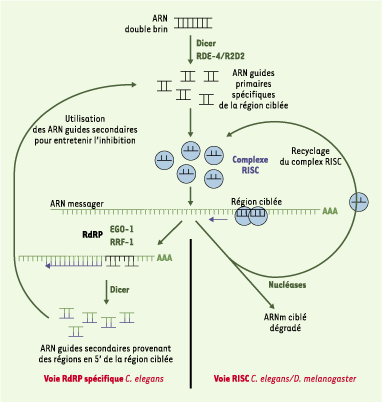
16]. L’ARN double brin est d’abord découpé en courtes molécules interférentes, appelées ARN guides. Celles-ci interagissent avec l’ARN messager ciblé et provoquent sa dégradation par une, ou deux voies distinctes, selon les organismes (Figure 1). De nombreuses protéines impliquées dans le mécanisme de l’ARNi ont été identifiées (Tableau I). Un certain nombre d’entre elles sont conservées. Ainsi la ribonucléase III Dicer qui dégrade les molécules d’ARN double brin et produit des ARN guides (Figure 1) est présente aussi bien chez C. elegans [
17–
19] que chez D. melanogaster [
20]. En revanche, d’autres protéines comme les ARN polymérases dépendantes de l’ARN sont spécifiques de C. elegans [16].  | Figure 1. Mécanisme hypothétique de l’interférence par l’ARN chez C. elegans. Après son incorporation dans l’animal, l’ARN double brin est découpé en molécules d’ARN de 21 nucléotides [45]. Ces molécules sont considérées comme l’agent interférant à proprement parler [25] et ont été appelées ARN guides (short interfering RNA (siRNA)). Elles sont incorporées au sein d’un complexe ribonucléoprotéique, appelé RISC (RNA-induced silencing complex). Cette incorporation permet la localisation du complexe RISC au niveau de l’ARNm cible qui va être dégradé par Tudor-SN, une nucléase présente au sein du complexe (voie RISC) [34]. D’autres études ont permis d’établir l’existence potentielle de la voie RdRP (RNA-dependant RNA polymerase), une seconde voie de dégradation de l’ARNm cible [16]. Après interaction des ARN guides avec l’ARNm cible, un second brin d’ARN complémentaire à l’ARN messager serait synthétisé. La molécule d’ARN double brin, ainsi formée, serait à son tour, clivée en ARN guides par l’enzyme Dicer, et les ARN guides néo-formés (ou secondaires, par opposition aux ARN guides primaires qui proviennent directement de la dégradation du signal) seraient utilisés pour entretenir l’inhibition. Les ARN polymérases dépendantes de l’ARN (EGO-1, RRF-1) nécessaires à la synthèse du brin d’ARN complémentaire ainsi que les ARN guides secondaires n’ont été mis en évidence que chez C. elegans, ce qui suggère que la voie RdRP est spécifique de cet organisme (d’après [16]). |
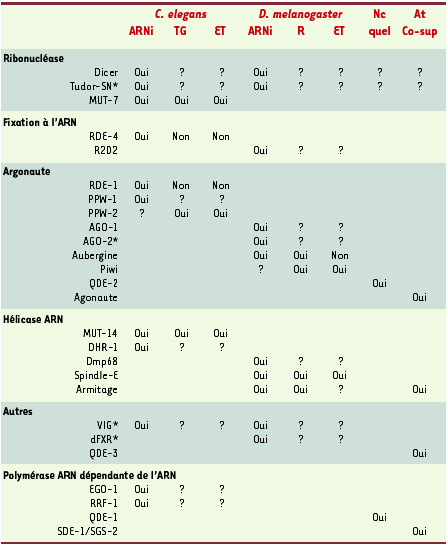
 | Tableau I Gènes impliqués dans le contrôle post-transcriptionnel des séquences répétées et l’interférence par l’ARN chez C. elegans et D. melanogaster. |
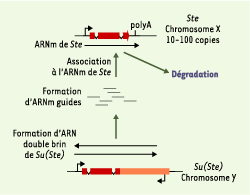
Contrôle post-transcriptionnel de l’expression des séquences répétées chez D. melanogaster L’expression des séquences Stellate (Ste), naturellement répétées dans le génome de D. melanogaster, semble régulée par un mécanisme semblable à l’ARNi [
22,
23]. Le locus Ste, localisé sur le chromosome X, est composé de la répétition en tandem d’un cadre ouvert de lecture dont le nombre de copies varie, selon les souches, de quelques unités à plus d’une centaine. L’expression de Ste est réprimée par Su(Ste), un locus indépendant localisé sur le chromosome Y et composé de répétitions en tandem très fortement apparentées à celles composant Ste. La délétion de ce locus se traduit par une stérilité des mâles associée à une surexpression de Ste. Des études successives ont permis de montrer que : (1) les gènes sting/aubergine, spindle-E/homeless et armitage (Tableau 1) impliqués dans l’ARNi sont également nécessaires au contrôle de Ste par Su(Ste) [ 22,
24] ; (2) les répétitions Su(Ste) produisent deux types de molécules d’ARN simple brin complémentaires, susceptibles de s’associer pour former une molécule double brin (Figure 2) ; (3) l’extinction de Ste s’accompagne, dans les testicules, de la production de molécules d’ARN dont la taille varie entre 25 et 27 nucléotides, et qui sont complémentaires de régions communes à Ste et Su(Ste). La relation entre production de molécules d’ARN double brin de Su(Ste) et extinction de Ste a été établie en utilisant un système cellulaire. Lorsque des cellules porteuses des répétitions Ste fusionnées avec un gène rapporteur sont transfectées avec une molécule d’ARN double brin du type de celle produite par Su(Ste), l’expression du gène rapporteur est supprimée.  | Figure 2. Répression de Ste par Su(Ste) chez D. melanogaster. L’existence de deux promoteurs en orientation inverse dans les unités de Su(Ste) conduit à la synthèse de molécules d’ARN complémentaires et à la formation d’ARN double brin permettant l’induction de la dégradation de l’ARNm de Ste par un mécanisme similaire à l’ARNi (voir texte). Les régions communes à Ste et Su(Ste) sont en rouge. |
Chez D. melanogaster, des mécanismes similaires à l’ARNi pourraient également intervenir dans le contrôle des éléments transposables. Ainsi, la recherche systématique d’ARN guides dans des embryons de D. melanogaster a montré qu’un grand nombre d’entre eux correspondent à des éléments transposables [
25]. De plus, en contexte mutant pour spindle-E/homeless, l’expression d’un certain nombre d’éléments transposables est augmentée [23]. « Oui » et « Non » indiquent que le gène est impliqué ou non dans le phénomène considéré (ARNi, extinction des transgènes (TG), contrôle des séquences répétées (R), contrôle des éléments transposables (ET)). « ? » signifie que l’implication du gène dans le phénomène considéré n’a pas été étudiée. Les protéines concernées respectivement dans le quelling (quel) chez Neurospora crassa (Nc) et la co-suppression (Co-sup) chez Arabidopsis thaliana (At), deux processus de contrôle de l’expression des séquences répétées, sont indiquées. Les protéines participant au complexe RISC (voir Figure 2) sont indiquées par une astérisque. Spindle-E est aussi connue sous le nom de Homeless, et Aubergine sous le nom de Sting. Plusieurs types de ribonucléase semblent intervenir dans les mécanismes d’extinction des gènes. Comme les membres de la famille Dicer (voir texte), les nucléases Tudor-SN ont été conservées au cours de l’évolution. Il s’agit de composants du complexe RISC. Elles pourraient intervenir dans la dégradation de l’ARNm ciblé [
34]. MUT-7 est une ribonucléase potentielle, identifiée exclusivement chez C. elegans [11], qui pourrait intervenir dans la stabilisation des ARN guides primaires [
35]. RDE-4 et R2D2 possèdent des motifs de fixation à l’ARN double brin et interagissent avec Dicer. Elles pourraient jouer un rôle dans le transfert des ARN guides primaires de l’enzyme Dicer au complexe RISC. Les membres de la famille Argonaute sont largement impliqués dans les mécanismes d’extinction des gènes. Leur(s) rôle(s) est (sont) encore mal défini(s) mais RDE-1 semble nécessaire à la stabilisation des ARN guides primaires chez C. elegans [35]. Cette protéine peut se fixer à RDE-4 [
36]. Chez D. melanogaster, Argonaute-1 (AGO-1) agit en aval de Dicer [
37] et Argonaute-2 (AGO-2) est associée aux ARN guides [
38] au sein du complexe RISC. Des mutants du gène aubergine sont défectifs pour l’ARNi [
39] et piwi est impliqué dans le contrôle des séquences répétées et des éléments transposables [
40,
41]. Des hélicases ARN putatives sont également impliquées dans l’ARNi. Chez C. elegans, mut-14 code pour une hélicase ARN potentielle [35], qui pourrait être nécessaire à l’initiation de la transcription inverse à l’origine de la formation de l’ARN double brin. DHR-1 est une autre hélicase ARN putative de C. elegans interagissant avec RDE-4 [36]. L’hélicase ARN Dmp68 de D. melanogaster est requise dans le mécanisme de l’ARNi [
42]. Enfin, chez D. melanogaster, Armitage, qui est impliquée dans l’ARNi et le contrôle des séquences répétées [
43], semble nécessaire à la formation d’un complexe RISC actif [24]. Cette protéine est très proche de SDE-3 présente chez Arabidopsis thaliana où elle joue un rôle dans la co-suppression. Chez D. melanogaster, VIG (vasa intronic gene) et dFXR, semblable à une protéine humaine dont l’expression est affectée chez les patients souffrant du syndrome de l’X fragile [
44], font partie du complexe RISC. Dicer, AGO-2 et Dmp68 interagissent in vivo avec dFXR. Chez C. elegans, seule VIG a été identifiée. EGO-1 et RRF-1 sont deux protéines ressemblant à des ARN polymérases dépendantes de l’ARN [16] reconnues chez C. elegans mais absentes chez D. melanogaster. On pense qu’elles utilisent l’ARNm comme matrice pour synthétiser une molécule d’ARN double brin qui sera dégradée en ARN guides secondaires. En accord avec cette hypothèse, aucun ARN guide secondaire n’a pu être détecté dans des mutants de RRF-1. |
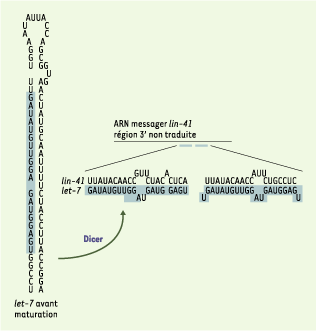
Des molécules de type ARN guide jouent un rôle essentiel dans le développement Outre leur rôle dans le contrôle des éléments transposables et des séquences virales [
26], les mécanismes que nous venons de décrire sont également impliqués dans la régulation du développement. De nombreux travaux ont ainsi permis de montrer que de très nombreux ARN d’une taille similaire à celle des ARN guides sont codés par le génome. Ils sont appelés microARN et leur conservation au cours de l’évolution permet d’envisager qu’ils jouent un rôle essentiel dans le développement [
27]. Leurs modes de production et d’action sont intensivement étudiés. Ainsi, on a montré que le microARN let-7 (Figure 3), conservé chez C. elegans, D. melanogaster, et chez l’homme, subit un processus de maturation dans lequel la protéine Dicer est impliquée [17,
18]. Cette observation est en accord avec l’hypothèse selon laquelle les microARN sont des ARN guides naturels. let-7 interagit ensuite avec la région 3’ non traduite des ARNm des gènes cibles dont il bloque la traduction, régulant ainsi leur expression. Parmi les cibles potentielles de let-7, on trouve lin-41. Il a été montré in vivo que ce gène, qui contrôle le passage de l’état larvaire à l’état adulte chez le nématode, est effectivement une cible biologique de let-7 [
28].  | Figure 3. Structure de l’ARN let-7 et de son précurseur et mécanisme de régulation de l’expression de ses cibles. Avant maturation par la protéine Dicer, l’ARN let-7 a une longueur de 70 nucléotides et forme une structure en tige-boucle. La maturation produit une molécule de 22 nucléotides partiellement complémentaire à la région 3’ non traduite de certains ARN messagers, dont celui de lin-41. L’interaction entre les deux séquences bloque la traduction de l’ARN messager (voir texte). |
Les efforts se portent aujourd’hui vers l’identification des cibles potentielles des microARN présents dans les génomes et sur la validation biologique des interactions ARN messagers-microARN prédites par bio-informatique. |
Deux études récentes montrent que chez C. elegans et D. melanogaster, les composantes transcriptionnelles et post-transcriptionnelles de la régulation de l’expression des séquences répétées sont probablement connectées, comme cela avait déjà été mis en évidence chez les plantes. Ainsi, les gènes mes de C. elegans interviendraient dans le contrôle de l’ARNi [
29], et le gène piwi (Tableau I) de D. melanogaster, impliqué dans la régulation post-transcriptionnelle, jouerait également un rôle dans la régulation transcriptionnelle des séquences répétées [
30]. L’étude plus approfondie de cette relation devrait mieux faire comprendre dans quelle mesure deux niveaux de régulation entrent en jeu pour, d’une part contrôler strictement la mise en place du plan d’organisation des organismes, d’autre part leur permettre de résister à l’envahissement de séquences parasites et de virus. La conservation du mécanisme d’ARNi et la facilité de l’appliquer sur le plan expérimental font de ce phénomène un outil particulièrement utile et performant dans le cadre d’études génomiques. Ainsi, l’ARNi est largement utilisée aujourd’hui dans des cellules humaines en culture pour éteindre l’expression des gènes et en élucider la fonction (pour revue, voir [
31]) (‹). (‹) m/s 2004, n° 8-9, p. 815
On a récemment montré que cette technique pouvait également être utilisée pour inhiber la prolifération virale dans des cellules de mammifères [
32,
33]. Bien que préliminaires en termes d’applications médicales, ces données permettent d’envisager l’utilisation de l’ARNi à des fins thérapeutiques et d’entrevoir que ce mécanisme de régulation, apparemment universel et encore inconnu il y a quelques années, pourrait bientôt être largement utilisé dans le domaine de la santé. |
Nous remercions Damien Coudreuse et Alain Pélisson d’avoir lu et critiqué ce manuscrit. Le travail de l’équipe d’Alain Bucheton est financé par des crédits du CNRS, de l’ARC et de l’Union Européenne.
|
1. Dorer DR, Henikoff S. Expansions of transgene repeats cause heterochromatin formation and gene silencing in Drosophila. Cell 1994; 77 : 993–1002. 2. Orlando V. Polycomb, epigenomes, and control of cell identity. Cell 2003; 112 : 599–606. 3. Strahl BD, Allis CD. The language of covalent histone modifications. Nature 2000; 403 : 41–5. 4. Pal-Bhadra M, Bhadra U, Birchler JA. Cosuppression in Drosophila : gene silencing of alcohol dehydrogenase by white-Adh transgenes is Polycomb dependent. Cell 1997; 90 : 479–90. 5. Stinchcomb DT, Shaw JE, Carr SH, Hirsh D. Extrachromosomal DNA transformation of Caenorhabditis elegans. Mol Cell Biol 1985; 5 : 3484–96. 6. Kelly WG, Schaner CE, Dernburg AF, et al. X-chromosome silencing in the germline of C. elegans. Development 2002; 129 : 479–92. 7. Couteau F, Guerry F, Muller F, Palladino F. A heterochromatin protein 1 homologue in Caenorhabditis elegans acts in germline and vulval development. EMBO Rep 2002; 3 : 235–41. 8. Kelly WG, Fire A. Chromatin silencing and the maintenance of a functional germline in Caenorhabditis elegans. Development 1998; 125 : 2451–6. 9. Emmons SW, Yesner L. High-frequency excision of transposable element Tc 1 in the nematode Caenorhabditis elegans is limited to somatic cells. Cell 1984; 36 : 599–605. 10. Emmons SW, Roberts S, Ruan KS. Evidence in a nematode for regulation of transposon excision by tissue-specific factors. Mol Gen Genet 1986; 202 : 410–5. 11. Ketting RF, Haverkamp TH, van Luenen HG, Plasterk RH. Mut-7 of C. elegans, required for transposon silencing and RNA interference, is a homolog of Werner syndrome helicase and RNaseD. Cell 1999; 99 : 133–41. 12. Ketting RF, Plasterk RH. A genetic link between co-suppression and RNA interference in C. elegans. Nature 2000; 404 : 296–8. 13. Dernburg AF, Zalevsky J, Colaiacovo MP, Villeneuve AM. Transgene-mediated cosuppression in the C. elegans germ line. Genes Dev 2000; 14 : 1578–83. 14. Fire A, Xu S, Montgomery MK, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature 1998; 391 : 806–11. 15. Montgomery MK, Xu S, Fire A. RNA as a target of double-stranded RNA-mediated genetic interference in Caenorhabditis elegans. Proc Natl Acad Sci USA 1998; 95 : 15502–7. 16. Sijen T, Fleenor J, Simmer F, et al. On the role of RNA amplification in dsRNA-triggered gene silencing. Cell 2001; 107 : 465–76. 17. Ketting RF, Fischer SE, Bernstein E, et al. Dicer functions in RNA interference and in synthesis of small RNA involved in developmental timing in C. elegans. Genes Dev 2001; 15 : 2654–9. 18. Grishok A, Pasquinelli AE, Conte D, et al. Genes and mechanisms related to RNA interference regulate expression of the small temporal RNAs that control C. elegans developmental timing. Cell 2001; 106 : 23–34. 19. Knight SW, Bass BL. A role for the RNase III enzyme DCR-1 in RNA interference and germ line development in C. elegans. Science 2001; 2 : 2. 20. Bernstein E, Caudy AA, Hammond SM, Hannon GJ. Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature 2001; 409 : 363–6. 21. Sijen T, Plasterk RH. Transposon silencing in the Caenorhabditis elegans germ line by natural RNAi. Nature 2003; 426 : 310–4. 22. Schmidt A, Palumbo J, Bozetti MP, et al. Genetic and molecular characterization of sting, a gene involved in crystal formation and meiotic drive in the male germ line of Drosophila melanogaster. Genetics 1999; 151 : 749–60. 23. Aravin AA, Naumova NM, Tulin AV, et al. Double-stranded RNA-mediated silencing of genomic tandem repeats and transposable elements in the D. melanogaster germline. Curr Biol 2001; 11 : 1017–27. 24. Tomari Y, Du T, Haley B, et al. RISC assembly defects in the Drosophila RNAi mutant armitage. Cell 2004; 116 : 831–41. 25. Elbashir SM, Lendeckel W, Tuschl T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes Dev 2001; 15 : 188–200. 26. Plasterk RH. RNA silencing : The genome’s immune system. Science 2002; 296 : 1263–5. 27. Ambros V. MicroRNAs : Tiny regulators with great potential. Cell 2001; 107 : 823–6. 28. Lin SY, Johnson SM, Abraham M, et al. The C elegans hunchback homolog, hbl-1, controls temporal patterning and is a probable microRNA target. Dev Cell 2003; 4 : 639–50. 29. Dudley NR, Labbe JC, Goldstein B. Using RNA interference to identify genes required for RNA interference. Proc Natl Acad Sci USA 2002; 99 : 4191–6. 30. Pal-Bhadra M, Bhadra U, Birchler JA. RNAi related mechanisms affect both transcriptional and posttranscriptional transgene silencing in Drosophila. Mol Cell 2002; 9 : 315–27. 31. McManus MT, Sharp PA. Gene silencing in mammals by small interfering RNAs. Nat Rev Genet 2002; 3 : 737–47. 32. Hu WY, Myers CP, Kilzer JM, et al. Inhibition of retroviral pathogenesis by RNA interference. Curr Biol 2002; 12 : 1301–11. 33. Gitlin L, Karelsky S, Andino R. Short interfering RNA confers intracellular antiviral immunity in human cells. Nature 2002; 418 : 430–4. 34. Caudy AA, Ketting RF, Hammond SH, et al. A micrococcal nuclease homologue in RNAi effector complexes. Nature 2003; 425 : 411–4. 35. Tijsterman M, Ketting RF, Okihara KL, et al. RNA helicase MUT-14-dependent gene silencing triggered in C. elegans by short antisense RNAs. Science 2002; 295 : 694–7. 36. Tabara H, Yigit E, Siomi H, et al. The dsRNA binding protein RDE-4 interacts with RDE-1, DCR-1, and a DExH-box helicase to direct RNAi in C. elegans. Cell 2002; 109 : 861–71. 37. Williams RW, Rubin GM. ARGONAUTE1 is required for efficient RNA interference in Drosophila embryos. Proc Natl Acad Sci USA 2002; 99 : 6889–94. 38. Hammond SM, Boettcher S, Caudy AA, et al. Argonaute2, a link between genetic and biochemical analyses of RNAi. Science 2001; 293 : 1146–50. 39. Kennerdell JR, Yamaguchi S, Carthew RW. RNAi is activated during Drosophila oocyte maturation in a manner dependent on aubergine and spindle-E. Genes Dev 2002; 16 : 1884–9. 40. Pal-Bhadra M, Bhadra U, Birchler JA. Cosuppression of nonhomologous transgenes in Drosophila involves mutually related endogenous sequences. Cell 1999; 99 : 35–46. 41. Sarot E, Payen-Groschene G, Bucheton A, Pelisson A. Evidence for a piwi-dependent RNA silencing of the gypsy endogenous retrovirus by the Drosophila melanogaster flamenco gene. Genetics 2004; 166 : 1313–21. 42. Ishizuka A, Siomi MC, Siomi H. A Drosophila fragile X protein interacts with components of RNAi and ribosomal proteins. Genes Dev 2002; 16 : 2497–508. 43. Cook HA, Koppetsch BS, Wu J, Theurkauf WE. The Drosophila SDE3 homolog armitage is required for oskar mRNA silencing and embryonic axis specification. Cell 2004; 116 : 817–29. 44. audy AA, Myers M, Hannon GJ, Hammond SM. Fragile X-related protein and VIG associate with the RNA interference machinery. Genes Dev 2002; 16 : 2491–6. 45. Zamore PD, Tuschl T, Sharp PA, Bartel DP. RNAi : Double-stranded RNA directs the ATP-dependent cleavage of mRNA at 21 to 23 nucleotide intervals. Cell 2000; 101 : 25–33. |