| |
| Med Sci (Paris). 2005 March; 21(3): 273–278. Published online 2005 March 15. doi: 10.1051/medsci/2005213273.Rôle de RIP140 dans la signalisation hormonale Audrey Castet, Sophie Carascossa, Vanessa Duong, Patrick Augereau, Stéphan Jalaguier, and Vincent Cavailles* Inserm U.540, Endocrinologie moléculaire et cellulaire des cancers, et Université de Montpellier I, 60, rue de Navacelles, 34090 Montpellier, France |
Récepteurs nucléaires et cofacteurs transcriptionnels Les récepteurs nucléaires (RN) forment une grande famille de facteurs de transcription impliqués dans de nombreux processus physiologiques fondamentaux tels que le développement, l’homéostasie, le métabolisme ou la reproduction. Ces récepteurs contrôlent l’expression de gènes cibles en recrutant différents types de corégulateurs transcriptionnels qui agissent, généralement sous forme de complexes multiprotéiques, en modulant l’efficacité d’initiation de la transcription ou la structure de la chromatine [
1]. Le remodelage chromatinien par ces cofacteurs implique différents types d’activités enzymatiques (ATPases, acétylases/désacétylases, méthylases…) capables de modifier le positionnement des nucléosomes ou de produire des signaux de reconnaissance pour différentes protéines régulatrices (code des histones). Le rôle physiologique d’un certain nombre de ces corégulateurs transcriptionnels a été analysé par invalidation du gène correspondant chez la souris, révélant selon les cas une létalité embryonnaire ou une diminution de la réponse hormonale dans certains tissus cibles. Par ailleurs, dans certaines situations pathologiques, en particulier lors de la transformation cancéreuse, ces gènes subissent différents types d’altérations (amplification génique, mutations, translocations…) qui peuvent participer au dérèglement de fonctions cellulaires contrôlées par les RN. |
Le cofacteur RIP140 : clonage et structure de la protéine RIP140 (receptor interacting protein of 140 kDa), également connu sous l’appellation NRIP1 (nuclear receptor interacting protein 1), a été l’un des premiers cofacteurs transcriptionnels humains des récepteurs hormonaux nucléaires à être caractérisé. L’ADNc correspondant a été isolé il y a dix ans dans le laboratoire de Malcolm Parker à Londres (Royaume-Uni) [
2]. L’approche utilisée reposait sur un criblage d’une banque d’expression d’ADNc établie à partir de cellules de cancer du sein, l’hybridation ayant été réalisée à l’aide d’une sonde protéique radiomarquée correspondant au domaine de liaison du ligand du récepteur αdes œstrogènes (REα), en présence d’œstrogènes. Quelques années plus tard, l’ADNc de RIP140 a également été isolé chez la souris par une approche de double hybride dans la levure [
3]. À l’heure actuelle, on peut obtenir, dans les banques de données, les séquences des ADNc de RIP140 de plusieurs espèces (rat, poulet, xénope, poisson-zèbre et fugu). L’analyse des séquences nucléotidiques a malheureusement été peu informative sur les différents domaines structuraux présents dans la protéine RIP140. On savait que plusieurs régions de la protéine permettaient son interaction in vitro avec les RN dans leur forme activée par l’hormone [
4]. L’identification de la séquence LxxLL comme motif consensus minimal d’interaction spécifique avec les RN [
5] a, par la suite, révélé que RIP140 contenait 9 de ces motifs, riches en leucine, répartis sur toute la molécule (Figures 1A et B). Ce nombre particulièrement élevé de motifs LxxLL est une première particularité de ce cofacteur, lui offrant ainsi une grande diversité dans le mode de recrutement par les RN. RIP140 interagit en effet avec de nombreux récepteurs, tels que le REalpha, le récepteur des hormones thyroïdiennes (TR), des rétinoïdes et réxinoïdes (RAR, RXR) [2–4] ou des glucocorticoïdes [
6], et il est également capable de se fixer à d’autres facteurs de transcription, comme le récepteur de l’aryl-hydrocarbone (AhR) [
7] et, plus récemment, c-jun [
8].
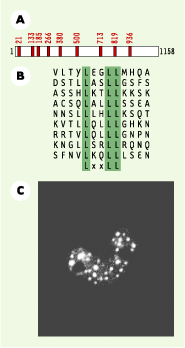
 | Figure 1.
Structure et localization de la proteine RIP140.A. Structure de RIP140. Les motifs LxxLL permettant l’interaction hormonodépendante avec les RN apparaissent sous forme de boîtes rouges. B. Alignement des séquences primaires des neuf motifs LxxLL présents dans la protéine RIP140. C. Localisation subcellulaire de RIP140. L’image a été obtenue par microscopie confocale sur des cellules COS-1 transfectées avec un vecteur d’expression codant pour une protéine chimère correspondant à RIP140 fusionné à la YFP ( yellow fluorescent protein) (d’après [
23]). |
|
Contrôle de l’expression de RIP140 Comme pour beaucoup de corégulateurs transcriptionnels des RN, l’expression de l’ARNm de RIP140 est ubiquitaire. L’analyse de l’expression du transcrit dans différents tissus chez la souris a montré une expression élevée dans des organes cibles des hormones tels que la glande mammaire, la prostate ou les ovaires [
9]. Dès l’identification de RIP140, il avait été noté que les œstrogènes augmentaient l’accumulation de la protéine dans les lignées cancéreuses mammaires [
10]. Cette régulation, également observée au niveau de l’ARNm, semble résulter d’une stimulation directe de la transcription du gène RIP140 par les récepteurs des œstrogènes [
11]. De manière intéressante, les rétinoïdes augmentent aussi l’expression de l’ARNm de RIP140 dans différentes lignées cancéreuses humaines [
12]. Par ailleurs, l’accumulation de l’ARNm est stimulée par les gonadotrophines au niveau de la granulosa ovarienne [
13]. Chez l’homme, le gène RIP140 est localisé sur le chromosome 21 (21q11.2), dans une région pauvre en gènes associée au syndrome de Down et à une forme d’apparition précoce de la maladie d’Alzheimer [
14]. La présence d’un chromosome surnuméraire à l’origine du syndrome de Down semble être associée à une modification de l’expression de RIP140 [
15], mais l’implication dans la pathologie de la trisomie 21 reste à ce jour hypothétique. Concernant l’expression de la protéine RIP140, sa détection par immunofluorescence montre une localisation nucléaire sous forme ponctuée [2]. Le même type de marquage est obtenu en surexprimant la protéine étiquetée par un marqueur fluorescent (Figure 1C) [
16]. L’utilisation de mutants de délétion [
17] a révélé que cette localisation sous forme de petits foyers nucléaires nécessite une séquence de 40 acides aminés (coordonnées 431 à 472 de la protéine). Par ailleurs, un changement de localisation apparaît lors de la coexpression de la protéine 14-3-3, qui délocalise RIP140 dans le cytoplasme [16]. |
Rôle au niveau transcriptionnel Le rôle de RIP140 en tant que répresseur transcriptionnel de la transactivation hormonodépendante de nombreux RN est maintenant clairement établi [2, 3, 8]. La mise en évidence de cette activité répressive renforce le côté atypique de ce cofacteur, puisque les paramètres de sa liaison aux récepteurs (interaction, en présence d’agoniste, avec un récepteur actif sur le plan transcriptionnel) laissaient supposer que RIP140 agisse comme un coactivateur transcriptionnel. Dans le cas du REα, l’effet répresseur de RIP140 a également été observé lorsque le récepteur est recruté de manière indirecte par des interactions protéine-protéine impliquant le complexe AP-1 [8]. De manière intéressante, RIP140 affecte également les réponses hormonales négatives, puisqu’il inhibe la répression par les glucocorticoïdes [6] ou les œstrogènes [
18] exercée respectivement sur les promoteurs des gènes de la collagénase ou du TNFα. Il faut cependant noter que, dans certaines situations, RIP140 règle positivement la transactivation des RN. Plusieurs études ont en effet rapporté que la surexpression de RIP140 augmente l’activité transcriptionnelle de différents RN (récepteurs des œstrogènes, de l’acide rétinoïque ou des glucocorticoïdes) chez S. cerevisiae [
19]. Dans les cellules de mammifères, un effet activateur de RIP140 a également été décrit sur l’activité d’une forme mutée du récepteur des œstrogènes [
20]. |
Mécanisme de la répression transcriptionnelle Le contrôle négatif exercé par RIP140 au niveau transcriptionnel implique des mécanismes complexes faisant intervenir différents médiateurs. Plusieurs travaux ont initialement proposé que l’activité inhibitrice de RIP140 découlait de sa capacité à entrer en compétition avec les coactivateurs transcriptionnels pour une même interface d’interaction sur le domaine de liaison de l’hormone des RN. À titre d’exemple, il a été montré que la liaison hormonodépendante de SRC-1 (steroid receptor coactivator) sur la partie carboxyterminale du récepteur PPARγ (peroxisome proliferator-activated receptor) est inhibée par un excès de RIP140, entraînant le blocage de l’effet positif de SRC-1 sur la transactivation du récepteur [
21]. Cependant, la mise en évidence d’une activité répressive intrinsèque de RIP140 a conduit à l’identification de plusieurs domaines transrépresseurs capables de recruter différents médiateurs pouvant participer à la régulation négative de la transcription (Figure 2). Les premiers partenaires identifiés ont été les histones désacétylases (HDAC). La région aminoterminale de RIP140 (acides aminés 27 à 199) est capable de recruter in vitro les HDAC de classe I (HDAC1, 2 et 3) ou de classe II (HDAC5) [
22, 23]. Cependant, bien qu’une activité enzymatique HDAC ait été retrouvée associée à RIP140, l’utilisation d’inhibiteurs de cette activité, comme la trichostatine A, donne des résultats variables quant à l’effet répressif exercé par RIP140 qui est, selon les cas, aboli [22] ou conservé [23,
24].
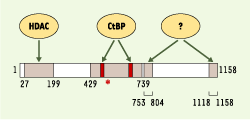
 | Figure 2.
Domaines répresseurs et partenaires de RIP140. Les quatre domaines répresseurs identifiés sont indiqués en gris, avec leurs coordonnées en acides aminés. Les régions permettant le recrutement des histones désacétylases (HDAC) et des C-terminal binding proteins (CtBP) sont notées. Le domaine central possède deux motifs PIxLS d’interaction avec les CtBP (boîtes rouges). Les corépresseurs participant à la répression exercée par les deux domaines carboxyterminaux restent à ce jour à caractériser. L’astérisque représente la lysine 446, dont l’acétylation contrôle le recrutement des CtBP. |
RIP140 possède par ailleurs deux motifs (PIDLS et PINLS, dans la région centrale de la molécule) lui permettant de recruter les protéines CtBP1 [
25] et CtBP2 [23, 24], également décrites comme des médiateurs de régulations transcriptionnelles négatives. Tout un ensemble d’observations suggère cependant que le recrutement des CtBP ne peut expliquer qu’une partie de la transrépression du cofacteur RIP140 qui est, par exemple, toujours mesurable dans des fibroblastes embryonnaires issus de souris dont les gènes CtBP1 et CtBP2 ont pourtant été invalidés [23, 24]. Il est néanmoins possible que l’implication des CtBP varie selon les situations (contexte cellulaire, promoteur considéré…) et selon les modifications post-traductionnelles des différents partenaires. En effet, RIP140 est une protéine acétylée, et il a été montré que l’acétylation de la lysine 446 diminuait fortement son interaction avec CtBP1 [25]. Enfin, deux autres domaines situés dans la partie carboxyterminale de RIP140 (Figure 2) ont été récemment identifiés [23, 24]. Ces deux régions possèdent une activité répressive intrinsèque très marquée, indépendante des HDAC et des CtBP. Différentes approches sont en cours afin d’identifier les médiateurs impliqués dans cette transrépression. |
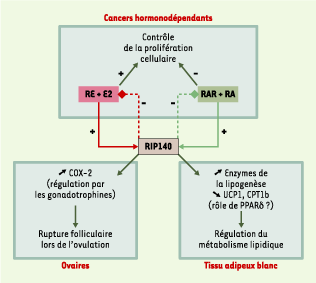
Rôle biologique de RIP140 Le rôle biologique exercé par RIP140 a été abordé par invalidation de son gène chez la souris, la quasi-totalité de la séquence codante ayant été remplacée par le gène lacZ [13]. Cette approche laisse entrevoir des implications de RIP140 dans le contrôle hormonal de différents processus fondamentaux (Figure 3).
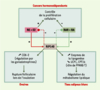 | Figure 3.Effets biologiques de RIP140. Rôles biologiques de RIP140, avec les boucles de régulation au niveau des cellules cancéreuses en réponse aux œstrogènes (œstradiol, ou E2) ou à l’acide rétinoïque (RA) (RE : récepteur des œstrogènes, RAR : récepteur de l’acide rétinoïque). Au niveau des ovaires, l’absence d’ovulation pourrait révéler une implication de RIP140 dans la régulation, par les gonadotrophines, de l’expression du gène codant pour la cyclo-oxygénase COX-2, dont l’invalidation conduit également à un phénotype anovulatoire [
27]. Le rôle de RIP140 au niveau du tissu adipeux blanc est également mentionné, avec la régulation de certaines enzymes de la lipogenèse comme l’acide gras synthétase, ainsi que des gènes CPT1b ( carnitine palmitoyltransferase 1b) et UCP-1 ( uncoupling protein-1, ou thermogénine), qui pourrait découler d’une perte de la répression de l’activité des récepteurs PPARdelta ( peroxisome proliferator-activated receptor) chez les souris dont le gène RIP140 a été invalidé (souris RIPKO). |
Ovulation et fertilité Les animaux n’exprimant pas RIP140 (souris RIPKO) sont viables, mais les femelles ne sont pas fertiles. En réponse à une stimulation par les gonadotrophines, le développement et la lutéinisation du follicule ovarien chez ces animaux sont normaux, mais les ovocytes ne sont pas libérés. Le phénotype observé chez les souris RIPKO est ainsi très proche du luteinised unruptured follicle syndrome, cause d’hypofécondité. Plusieurs arguments indiquent que le défaut se situe au niveau ovarien plutôt qu’au niveau de l’axe hypothalamo-hypophysaire. La démonstration en a été apportée par des expériences de transplantation ovarienne, révélant que l’absence d’expression de RIP140 au niveau de la gonade suffit à rendre compte de la stérilité observée [
26]. Par ailleurs, le transfert d’embryons sauvages chez des souris RIPKO pseudo-gestantes indique que l’efficacité de l’implantation est comparable à celle observée chez les souris sauvages. Enfin, le développement et la maturation des ovocytes chez les souris RIPKO sont également normaux, puisqu’ils peuvent être fécondés in vitro. Au niveau moléculaire, l’une des pistes envisagées concerne l’implication éventuelle de RIP140 dans la régulation par les gonadotrophines de l’expression du gène codant pour la cyclo-oxygénase COX-2, dont l’invalidation conduit également à un phénotype anovulatoire [ 27]. Catabolisme des graisses Les souris RIPKO mâles et femelles présentent une réduction d’environ 20 % de la masse corporelle, liée à une diminution de près de 70 % de la masse graisseuse totale qui affecte plus précisément le tissu adipeux blanc [
28]. Ces animaux présentent des concentrations de leptine circulante réduites de près de 4 fois et, contrairement aux souris sauvages, n’augmentent pas leur masse adipeuse lorsqu’elles sont soumises à un régime riche en graisses. Le phénotype observé ne résulte pas d’un défaut dans la différenciation adipocytaire. En revanche, dans le tissu adipeux blanc, il est associé, d’une part, à une diminution de l’expression de facteurs impliqués dans la lipogenèse (comme l’acide gras synthétase) et, d’autre part, à une forte augmentation de l’expresssion des facteurs de la thermogenèse que sont CPT1b ( carnitine palmitoyltransferase 1b) et UCP-1 ( uncoupling protein-1, ou thermogénine). Dans le tissu adipeux brun, ces facteurs sont impliqués respectivement dans le transport des acides gras au travers de la membrane externe des mitochondries et dans le découplage de l’énergie de la phosphorylation oxydative mitochondriale. Il est cependant nécessaire de mentionner que l’expression des gènes CPT1b et UCP-1 n’est pas significativement modifiée dans les adipocytes bruns des souris RIPKO. RIP140 semble donc jouer un rôle spécifique dans les adipocytes blancs en réprimant l’expression de gènes majeurs de la thermogenèse. L’impact de l’absence de ces différentes régulations sur la diminution de la masse adipeuse blanche chez les animaux RIPKO reste cependant inconnu. D’un point de vue mécanistique, la régulation de l’expression des gènes CPT1b et UCP-1 par RIP140 pourrait impliquer le récepteur PPARδ : en effet, dans des expériences de surexpression ciblée par transgenèse, PPARδ est également capable d’induire l’expression du gène UCP-1 dans le tissu adipeux blanc [
29]. Finalement, RIP140 apparaît, au même titre que les cofacteurs PGC-1α (PPARγ co-activator 1α), SRC-1 et TIF-2 (transcriptional intermediary factor 2), comme un régulateur important de l’homéostasie des lipides. En effet, dans les conditions physiologiques et au niveau du tissu adipeux blanc, RIP140 contrôle l’expression de gènes de la lipogenèse (effet positif) et contrôle négativement des gènes impliqués dans la dissipation de l’énergie et le découplage au niveau mitochondrial. Cancers hormonodépendants Dans les cancers du sein et de l’ovaire, plusieurs gènes codant pour des coactivateurs de RN ( AIB1, RAP250…) sont amplifiés, et la surexpression des protéines correspondantes est vraisemblablement impliquée dans le processus de cancérogenèse (pour revue, voir [
30]). En s’opposant à l’action de ces coactivateurs, RIP140 pourraît jouer un rôle clé dans le contrôle hormonal de la croissance des cancers hormonodépendants. Une étude récente suggère que l’ARNm de RIP140 est exprimé en quantité limitante dans une lignée cancéreuse humaine [
31]. Des variations d’expression de RIP140 de faible amplitude peuvent donc avoir des conséquences importantes sur la réponse hormonale au niveau cellulaire. |
Conclusions et perspectives De nombreux processus physiologiques fondamentaux sont contrôlés par des stimulus hormonaux et relayés par les RN. En participant à la régulation fine de l’activité transcriptionnelle de ces récepteurs hormonaux, le cofacteur RIP140 joue vraisemblablement un rôle important en physiopathologie humaine. L’invalidation du gène RIP140 chez la souris conduit à des phénotypes proches de ceux obtenus après invalidation des gènes codant pour les récepteurs REβ ou PR (phénotype ovarien) ou ERRα (tissu adipeux), suggérant des répercussions physiopathologiques majeures des différentes interactions transcriptionnelles observées entre RIP140 et les RN. L’implication de RIP140 lors de l’ovulation ou dans la régulation du stockage des graisses laisse entrevoir un intérêt évident, comme marqueur biologique, dans certaines formes de stérilités inexpliquées ou d’obésité. Par ailleurs, la localisation chromosomique du gène n’exclut pas un rôle dans le syndrome associé à la trisomie 21. Enfin, des approches visant à surexprimer RIP140 par transgenèse chez la souris déboucheront peut-être sur de nouvelles fonctions biologiques encore inconnues. Il est maintenant nécessaire de mettre en œuvre des études cliniques afin de préciser les dérèglements éventuels de l’expression de ce cofacteur dans ces différentes situations pathologiques. Plusieurs études d’analyse globale du transcriptome humain ont déjà donné des informations et des pistes intéressantes concernant le diabète au cours de la grossesse [
32] ou certains types de cancers [
33]. Ces résultats devraient pouvoir être confirmés au niveau moléculaire par des expériences réalisées en lignées cellulaires, permettant de définir de nouveaux partenaires de RIP140 et/ou de nouvelles voies de régulation au niveau transcriptionnel ou post-transcriptionnel. |
1. Smith CL, O’Malley BW. Coregulator function : a key to understanding tissue specificity of selective receptor modulators. Endocrinol Rev 2004; 25 : 45–71. 2. Cavailles V, Dauvois S, L’Horset F, et al. Nuclear factor RIP140 modulates transcriptional activation by the estrogen receptor. EMBO J 1995; 14 : 3741–51. 3. Lee CH, Chinpaisal C, Wei LN. Cloning and characterization of mouse RIP140, a corepressor for nuclear orphan receptor TR2. Mol Cell Biol 1998; 18 : 6745–55. 4. L’Horset F, Dauvois S, Heery DM, et al. RIP-140 interacts with multiple nuclear receptors by means of two distinct sites. Mol Cell Biol 1996; 16 : 6029–36. 5. Heery DM, Hoare S, Hussain S, et al. Core LXXLL motif sequences in CREB-binding protein, SRC1, and RIP140 define affinity and selectivity for steroid and retinoid receptors. J Biol Chem 2001; 276 : 6695–702. 6. Subramaniam N, Treuter E, Okret S. Receptor interacting protein RIP140 inhibits both positive and negative gene regulation by glucocorticoids. J Biol Chem 1999; 274 : 18121–7. 7. Kumar MB, Tarpey RW, Perdew GH. Differential recruitment of coactivator RIP140 by Ah and estrogen receptors. Absence of a role for LXXLL motifs. J Biol Chem 1999; 274 : 22155–64. 8. Teyssier C, Belguise K, Galtier F, et al. Receptorinteracting protein 140 binds c-Jun and inhibits estradiol-induced activator protein-1 activity by reversing glucocorticoid receptor-interacting protein 1 effect. Mol Endocrinol 2003; 17 : 287–99. 9. Panda S, Antoch MP, Miller BH, et al. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell 2002; 109 : 307–20. 10. Cavailles V, Dauvois S, Danielian PS, Parker MG. Interaction of proteins with transcriptionally active estrogen receptors. Proc Natl Acad Sci USA 1994; 91 : 10009–13. 11. Thenot S, Charpin M, Bonnet S, Cavailles V. Estrogen receptor cofactors expression in breast and endometrial human cancer cells. Mol Cell Endocrinol 1999; 156 : 85–93. 12. Kerley JS, Olsen SL, Freemantle SJ, Spinella MJ. Transcriptional activation of the nuclear receptor corepressor RIP140 by retinoic acid : a potential negative-feedback regulatory mechanism. Biochem Biophys Res Commun 2001; 285 : 969–75. 13. White R, Leonardsson G, Rosewell I, et al. The nuclear receptor co-repressor nrip1 (RIP140) is essential for female fertility. Nat Med 2000; 6 : 1368–74. 14. Katsanis N, Ives JH, Groet J, et al. Localisation of receptor interacting protein 140 (RIP140) within 100 kb of D21S13 on 21q11, a gene-poor region of the human genome. Hum Genet 1998; 102 : 221–3. 15. FitzPatrick DR, Ramsay J, McGill NI, et al. Transcriptome analysis of human autosomal trisomy. Hum Mol Genet 2002; 11 : 3249–56. 16. Zilliacus J, Holter E, Wakui H,et al. Regulation of glucocorticoid receptor activity by 14—3-3-dependent intracellular relocalization of the corepressor RIP140. Mol Endocrinol 2001; 15 : 501–11. 17. Tazawa H, Osman W, Shoji Y, et al. Regulation of subnuclear localization is associated with a mechanism for nuclear receptor corepression by RIP140. Mol Cell Biol 2003; 23 : 4187–98. 18. An J, Ribeiro RC, Webb P, et al. Estradiol repression of tumor necrosis factor-α transcription requires estrogen receptor activation function-2 and is enhanced by coactivators. Proc Natl Acad Sci USA 1999; 96 : 15161–6. 19. Joyeux A, Cavailles V, Balaguer P, Nicolas JC. RIP 140 enhances nuclear receptor-dependent transcription in vivo in yeast. Mol Endocrinol 1997; 11 : 193–202. 20. Henttu PM, Kalkhoven E, Parker MG. AF-2 activity and recruitment of steroid receptor coactivator 1 to the estrogen receptor depend on a lysine residue conserved in nuclear receptors. Mol Cell Biol 1997; 17 : 1832–9. 21. Treuter E, Albrektsen T, Johansson L, et al. A regulatory role for RIP140 in nuclear receptor activation. Mol Endocrinol 1998; 12 : 864–81. 22. Wei LN, Hu X, Chandra D, et al. Receptor-interacting protein 140 directly recruits histone deacetylases for gene silencing. J Biol Chem 2000; 275 : 40782–7. 23. Castet A, Boulahtouf A, Versini G, et al. Multiple domains of the receptor-interacting protein 140 contribute to transcription inhibition. Nucleic Acids Res 2004; 32 : 1957–66. 24. Christian M, Tullet JM, Parker MG. Characterisation of four autonomous repression domains in the corepressor RIP140. J Biol Chem 2004; 279 : 15645–51. 25. Vo N, Fjeld C, Goodman RH. Acetylation of nuclear hormone receptor-interacting protein RIP140 regulates binding of the transcriptional corepressor CtBP. Mol Cell Biol 2001; 21 : 6181–8. 26. Leonardsson G, Jacobs MA, White R, et al. Embryo transfer experiments and ovarian transplantation identify the ovary as the only site in which nuclear receptor interacting protein 1/RIP140 action is crucial for female fertility. Endocrinology 2002; 143 : 700–7. 27. Lim H, Paria BC, Das SK, et al. Multiple female reproductive failures in cyclooxygenase 2-deficient mice. Cell 1997; 91 : 197–208. 28. Leonardsson G, Steel JH, Christian M, et al. Nuclear receptor corepressor RIP140 regulates fat accumulation. Proc Natl Acad Sci USA 2004; 101 : 8437–42. 29. Wang YX, Lee CH, Tiep S,
Peroxisome-proliferator-activated receptor delta activates fat metabolism to prevent obesity. Cell 2003; 113 : 159–70. 30. Rouayrenc JF, Castet A, Rey JM, et al. Genetic alterations of transcription cofactors in solid tumors. Bull Cancer 2002; 89 : 357–64. 31. White KA, Yore MM, Warburton SL, et al. Negative feedback at the level of nuclear receptor coregulation. Self-limitation of retinoid signaling by RIP140. J Biol Chem 2003; 278 : 43889–92. 32. Radaelli T, Varastehpour A, Catalano P, Hauguel-de Mouzon S. Gestational diabetes induces placental genes for chronic stress and inflammatory pathways. Diabetes 2003; 52 : 2951–8. 33. Baldus CD, Liyanarachchi S, Mrozek K, et al. Acute myeloid leukemia with complex karyotypes and abnormal chromosome 21 : amplification discloses overexpression of APP, ETS2 and ERG genes. Proc Natl Acad Sci USA 2004; 101 : 3915–20. |