| |
| Med Sci (Paris). 2005 April; 21(4): 422–427. Published online 2005 April 15. doi: 10.1051/medsci/2005214422.Contrôles épigénétiques, développement et variation génétique naturelle chez les plantes Manoel Prouteau and Vincent Colot* Unité de Recherche en Génomique végétale, UMR INRA 1165-CNRS 8114-UEVE, 2, rue Gaston-Crémieux, 91057 Évry Cedex, France |
Régulations épigénétiques au cours du développement La plupart des données concernant les mécanismes épigénétiques mis en jeu dans le développement des plantes proviennent d’études génétiques et moléculaires menées sur l’espèce modèle Arabidopsis thaliana (Figure 1). À ce jour, de telles régulations ont été identifiées dans un nombre limité de cas, les deux plus frappants touchant un aspect particulier de la floraison et la formation de la graine [
1].  | Figure 1.
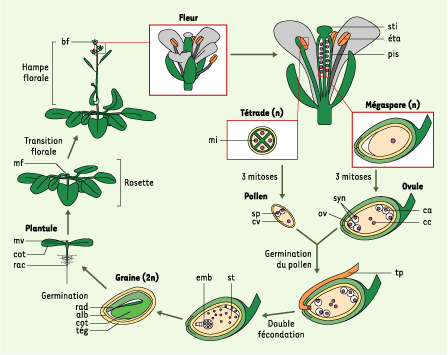
Cycle de vie chez Arabidopsis thaliana. Les fleurs de cette plante sont hermaphrodites et portent à la fois des organes mâles, les étamines (éta), et un organe femelle, le pistil (pis). Au sein des étamines, la méiose produit une tétrade composée de 4 microspores haploïdes (mi) ; chacune subit deux divisions mitotiques successives afin de produire un gamète mâle, le grain de pollen : la première division mitotique produit un noyau végétatif (nv) et une cellule génératrice qui subit à son tour une seconde mitose donnant naissance à deux cellules spermatiques (sp). Le pistil, quant à lui, produit à la suite de la méiose des mégaspores qui subissent trois mitoses successives avant de produire l’ovule mature. Ces trois divisions produisent 7 cellules haploïdes : trois cellules apicales (ca), un ovocyte (ov) encadré par deux synergides (syn) et une cellule centrale (cc) binucléée. Le grain de pollen germe une fois déposé au sommet du pistil, sur le stigmate (sti). Le noyau végétatif produit un tube pollinique (tp) qui pénètre le pistil jusque dans l’ovule. Il y dépose alors les deux cellules spermatiques et l’ovule subit une double fécondation. La première cellule spermatique féconde les deux noyaux de la cellule centrale et forme ainsi un noyau triploïde. La seconde féconde l’oosphère et produit un zygote (2n). Le noyau triploïde se divise initialement sans division cellulaire pour former un syncytium (st) qui donnera l’albumen (alb) après cellularisation partielle. Le zygote se divise également et engendre l’embryon. Lors de la germination, le radicule (rad) perce le tégument de la graine puis se développe en racine (rac). Les deux cotylédons (cot) assurent temporairement la photosynthèse de la plantule qui produit ensuite des feuilles organisées en rosette. C’est au moment de la transition florale que le centre végétatif de la plante acquiert une nouvelle fonction : il produit une hampe florale (hf) au sommet de laquelle se développent les boutons floraux (bf). |
La vernalisation : une mémoire du froid au service du développement floral Chez les plantes à fleurs, la transition entre les phases végétative et reproductive dépend à la fois de signaux endogènes et de conditions environnementales favorables, comme par exemple des jours longs et une température suffisamment clémente. Cependant, de nombreuses plantes nécessitent également pour fleurir une étape préalable de vernalisation, qui consiste en une exposition prolongée au froid à un stade précoce de leur développement. C’est ainsi que si les blés de printemps sont insensibles à la vernalisation, les blés d’hiver doivent impérativement être semés à l’automne afin d’assurer leur floraison au printemps suivant. Chez Arabidopsis, où l’on retrouve le même phénomène, la floraison repose sur un réseau complexe de gènes, parmi lesquels un répresseur central de la transition florale, le FLOWERING LOCUS C (FLC) [
2]. Les études moléculaires ont montré que, dans les souches sensibles à la vernalisation, l’exposition aux basses températures des plantules ou des graines en cours de germination conduit à la répression stable du gène FLC jusqu’à la fin du cycle de reproduction. De plus, l’intensité de la répression augmente progressivement avec le temps passé au froid, et cette mémoire des conditions hivernales détermine la précocité de la floraison au retour des beaux jours [
3]. La recherche de mutants affectés dans la vernalisation a conduit à l’identification de plusieurs facteurs spécifiquement impliqués dans la répression de FLC par le froid [2]. Le gène VERNALIZATION INSENSITIVE 3 (VIN 3) code une protéine à homéodomaine retrouvée dans plusieurs complexes de remodelage de la chromatine [
4]. Son expression est induite par la vernalisation et coïncide avec l’inhibition du gène FLC, alors qu’en contexte mutant vin3, l’expression de FLC demeure inchangée après traitement par le froid. Des expériences d’immunoprécipitation de la chromatine ont par ailleurs montré que la répression de FLC par VIN3 implique des modifications profondes de la chromatine au niveau du locus cible, qui passe d’un état « euchromatinien », caractérisé par une hyperacétylation des histones H3 et H4 ainsi qu’une triméthylation de la lysine 4 de l’histone H3 (H3K4me3), à un état « hétérochromatinien », marqué par une désacétylation des histones H3 et H4 et une méthylation des lysines 9 et 27 de l’histone H3 (H3K9me2 et H3K27me3) [
5]. Il faut cependant noter que la méthylation de l’ADN, marque épigénétique par excellence et qui est étroitement associée à l’hétérochromatine chez Arabidopsis [
6], ne semble intervenir à aucun moment dans la répression de FLC [4,
7]. Si VIN3 est nécessaire à l’initiation de la répression du gène FLC par le froid, il n’est en revanche nullement impliqué dans la perpétuation de l’état réprimé au-delà de la période de vernalisation [4]. L’analyse génétique a montré que cette fonction était dépendante de deux autres gènes, VRN1 et VRN2 (VERNALISATION 1 et 2) (Figure 2) [2, 5]. Le gène VRN1 code une protéine spécifique des plantes capable de se fixer in vitro à l’ADN, tandis que VRN2 est apparenté au gène SUPPRESSOR OF ZESTE 12 (Su[z]12) de la drosophile [
8]. Or, ce dernier est impliqué, avec les autres gènes codant les protéines du groupe Polycomb, dans le maintien de l’état réprimé de nombreux gènes clés du développement de la mouche, et ce au travers de multiples divisions cellulaires [
9]. De plus, la protéine Su(z)12 agit au sein d’un complexe possédant une activité méthyltransférase dirigée vers les lysines 9 et 27 de l’histone H3 [
10]. Enfin, la méthylation de l’ADN est quasiment inexistante chez la drosophile et ne semble y jouer aucun rôle dans la répression transcriptionnelle. La fonction et le mode d’action de la protéine VRN2 seraient donc en tous points comparables à ceux de son homologue animal [2].  | Figure 2.
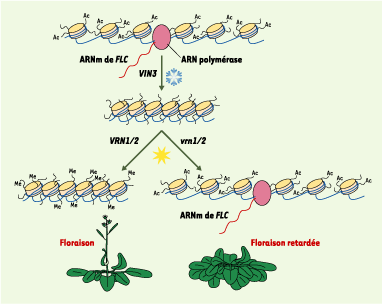
Modèle de répression du gène FLC par la vernalisation. En l’absence de vernalisation, le répresseur floral FLC (flowering locus c) est dans une configuration chromatinienne « ouverte » caractérisée par une hyperacétylation des histones H3 et H4 et une forte expression. Lors d’un traitement par le froid, l’expression du gène VIN3 (VERNALIZATION INSENSITIVE 3) est induite et conduit à la désacétylation des histones H3 et H4 au locus FLC et à son inactivation. La répression transcriptionnelle de FLC, qui n’est que transitoire à ce stade, est maintenue par le complexe VRN1/2 (VERNALISATION 1 et 2) impliqué dans l’ajout de groupements méthyles sur les lysines 9 et 27 de l’histone H3. C’est le maintien de cette inactivation qui permettra une floraison précoce lors de l’allongement des jours et de l’augmentation de la température. En contexte mutant vrn1 ou vrn2, la répression de FLC n’est pas maintenue après vernalisation, ce qui conduit à un retard de la floraison. |
L’empreinte parentale : un mécanisme restreint, chez les plantes, au développement de l’albumen L’empreinte parentale désigne tout processus qui conduit à l’expression différentielle de certains gènes selon qu’ils sont transmis par les gamètes mâles ou femelles. Bien qu’initialement décrite chez le maïs [
11], son analyse est beaucoup plus avancée chez les mammifères (→). Chez la souris, plus de 70 gènes sont régulés ainsi 1. Bien que ce nombre soit limité au regard des 20 000 à 30 000 gènes identifiés dans le génome murin, l’empreinte parentale n’en demeure pas moins essentielle au développement normal de l’embryon comme à celui du placenta (→) et persiste pour certains gènes jusque dans la vie adulte [
12]. Si, pour des raisons évidentes, peu de choses sont encore connues quant à son rôle au cours de l’embryogenèse humaine, nous savons cependant que les défauts d’empreinte sont à l’origine de nombreuses maladies chez l’homme [
13] (→). (→) m/s 2005, n° 4, p. 396
(→) m/s 2005, n° 4, p. 390
(→) m/s 2005, n° 4, p. 405
Chez les plantes, à l’inverse, ce processus n’agit que de façon très restreinte au cours du cycle de vie, et semble ne toucher qu’un très petit nombre de gènes. Chez Arabidopsis, qui compte environ 28 000 gènes et où les travaux sont les plus complets, seuls deux cas d’expression mono-allélique ont été identifiés à ce jour : ils concernent les gènes MEDEA (MEA) et FWA, qui codent respectivement une protéine du groupe Polycomb et une protéine à homéodomaine [
14]. Les analyses génétiques ont montré que la contribution maternelle du gène MEA est nécessaire au développement normal de la graine, où elle jouerait un rôle modérateur de la croissance [
15], en accord avec la théorie du conflit parental postulée chez les mammifères. Des expériences de RT-PCR et d’hybridation in situ ont confirmé que si les deux copies d’origine maternelle sont exprimées dans l’albumen, la copie paternelle est silencieuse [14]. Bien que les données concernant l’embryon demeurent controversées, notamment en raison de l’absence apparente d’expression du génome d’origine paternelle lors des deux premiers jours suivant la formation du zygote [
16], nous savons néanmoins que les copies maternelle et paternelle du gène MEA sont transcrites après germination, et donc non soumises à l’empreinte [14]. À l’inverse, le gène FWA est normalement réprimé dans l’embryon ainsi que durant la phase végétative, où il montre une forte méthylation de sa partie 5’. Cependant, comme MEA, FWA est exprimé dans l’albumen spécifiquement à partir du génome d’origine maternelle, et cette expression est dans les deux cas dépendante de l’activité du gène demeter (DME) [
17]. Ce gène, qui code une ADN glycosylase, présente lui-même un profil d’expression tout à fait spécifique, puisqu’il n’est actif que dans la cellule centrale, et seulement avant fécondation de celle-ci par l’un des deux noyaux spermatiques [
18] (Figure 3). Même si la fonction de la protéine DME demeure inconnue, les données génétiques indiquent qu’elle pourrait agir comme déméthylase de l’ADN. En effet, son action sur les gènes MEA et FWA est contrecarrée par celle de la protéine MET1 [17, 18], principale méthyltransférase de maintenance présente chez Arabidopsis. De plus, une mutation dans le gène met1 induit une expression du gène FWA dans la plante entière [
19], et notamment de la copie paternelle dans l’albumen [17]. Sur la base de ces observations, il a été proposé que les mécanismes épigénétiques de régulation associés à la méthylation de l’ADN ne puissent agir qu’à « sens unique » chez les plantes, et donc exclusivement dans des tissus ne contribuant pas à la lignée germinale, tels que l’albumen [17]. Cette hypothèse, qu’il reste à étayer, expliquerait a contrario l’absence apparente d’implication de la méthylation de l’ADN dans le processus de vernalisation, puisque la répression stable du gène FLC induite par le froid doit être effacée à chaque génération. 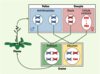 | Figure 3.
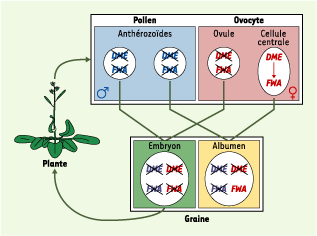
Empreinte parentale sur FWA. Chez la plante adulte, le promoteur du gène FWA est fortement méthylé et transcriptionnellement inactif. Les cellules spermatiques et l’ovocyte héritent donc d’un allèle méthylé et inactif. Cependant, le profil de méthylation de FWA est modifié dans la cellule centrale, sous l’action du gène DME, qui ne s’exprime que dans cette cellule et code une déméthylase présomptive. L’albumen, issu de la fécondation entre la cellule centrale et une cellule spermatique, hérite donc (en deux copies) d’un allèle maternel hypométhylé et actif de FWA, et d’un allèle paternel inactif. L’embryon hérite quant à lui d’allèles maternel et paternel inactifs. |
|
Méthylation de l’ADN et variabilité génétique naturelle La méthylation des cytosines de l’ADN est une modification postréplicative que l’on retrouve chez la plupart des eucaryotes. Elle affecte principalement les séquences répétées du génome et participe à sa stabilisation, d’une part en empêchant la transcription et la mobilisation des éléments transposables, d’autre part en rendant les répétitions inaptes à la recombinaison homologue [
20]. Chez les mammifères, la méthylation touche également les exons et est localisée aussi bien dans les gènes qu’à l’extérieur de ceux-ci, en raison de la colonisation des introns par les éléments répétés [
21,
22]. Chez les plantes, à l’inverse, la distribution de ces éléments - et donc des méthylcytosines - est essentiellement intergénique [22]. Enfin, alors que les profils de méthylation chez la souris varient de façon importante au cours du développement et sont effacés puis rétablis à chaque génération [21,
23], tout indique qu’il n’en est rien chez les plantes, si bien que tout changement « accidentel » de méthylation se produisant à une génération est transmis tel quel au travers de la méiose [
24]. De fait, le premier exemple d’« épimutant » naturel jamais répertorié est le variant génétique peloric de la linaire commune, découvert par le botaniste Carl von Linné en 1749. Ce variant se distingue du type sauvage par sa symétrie florale et avait été décrit alors comme une nouvelle espèce. L’analyse moléculaire a montré que l’altération à l’origine de peloric ne correspond pas à une mutation de la séquence du gène affecté (le gène cycloidea, qui contrôle la symétrie dorsoventrale des fleurs), mais à une extinction de son expression associée à une hyperméthylation du locus [
25]. En accord avec ces données, et bien qu’il soit transmis selon les lois de Mendel, l’allèle peloric est d’une stabilité relative, comme l’indique l’apparition occasionnelle de fleurs à phénotype sauvage ou intermédiaire. De nombreuses épimutations spontanées ou induites expérimentalement sont connues chez le maïs et Arabidopsis [24]. Par exemple, et de façon inattendue puisqu’il s’agit d’un phénomène observé à la suite d’une expérience de mutagenèse à l’EMS (éthylméthane sulfonate), de nombreux « épiallèles » du gène SUPERMAN (SUP) ont été isolés chez Arabidopsis, tous présentant une hyperméthylation du promoteur et une inactivation transcriptionnelle du gène [
26]. Par ailleurs, nous avons signalé plus haut que la mutation met1 conduit à une perte de méthylation au locus FWA et à l’activation transcriptionnelle du gène dans l’ensemble de la plante, et non plus exclusivement dans l’albumen. Or, cette réactivation/déméthylation de FWA est héritée indépendamment de met1, et représente donc un autre cas d’épimutation [19]. Enfin, il a été montré, au moyen d’une approche génomique, que l’induction d’une hypométhylation globale du génome d’Arabidopsis par une mutation du gène DECREASE IN DNA methylation 1 (DDM1), qui code un facteur de remodelage de la chromatine, conduit à un « réveil » transcriptionnel massif des éléments transposables. Cette réactivation est transmise à la descendance même après élimination de la mutation ddm1 par croisement, et a, dans certaines situations, un impact sur l’expression des gènes situés à proximité immédiate. C’est ainsi que la formation d’épiallèles FWA à expression constitutive, observée de façon semblable dans les mutants ddm1 et met1, résulte directement de l’activation de séquences promotrices dérivant d’un rétroélément SINE (short interspersed nuclear element) [6]. À l’échelle du génome, ce sont probablement plusieurs dizaines de gènes qui ont acquis de la sorte une possibilité de varier par épimutation, possibilité qui leur serait autrement interdite (A.V. Gendrel et V. Colot, résultats non publiés). |
Les données génétiques et moléculaires ne laissent guère de doute quant à l’implication relativement modeste des processus épigénétiques dans la régulation du développement chez les plantes. Néanmoins, le génome d’Arabidopsis contient une abondance de gènes2 dont les homologues animaux sont impliqués dans la perpétuation d’états chromatiniens au travers des divisions cellulaires. Il reste donc à comprendre comment ces gènes fonctionnent chez les plantes, puisque les parallèles observés dans le cas de la vernalisation et, dans une moindre mesure, de l’empreinte parentale semblent constituer l’exception. À l’opposé, les plantes se caractérisent par une capacité importante à produire des épimutations transmissibles à la descendance, contrairement aux mammifères. En effet, si chez ces derniers les altérations épigénétiques sont souvent associées au cancer [
27], rares sont celles transmises lors de la méiose [
28]. Cette absence de transmission s’explique par l’existence de mécanismes efficaces de remise à plat à chaque génération des régulations épigénétiques essentielles au développement des mammifères. Quoi qu’il en soit, il semble difficile d’ignorer plus longtemps la possibilité d’une contribution non négligeable des épimutations à la variabilité génétique naturelle, aussi bien chez les plantes que chez d’autres organismes. Compte tenu de leur caractère souvent métastable, ces altérations pourraient en effet être à l’origine de nombreuses situations où une hérédité mendélienne stricte n’est pas observée. |
Footnotes |
1. Steimer A, Schob H, Grossniklaus U. Epigenetic control of plant development: new layers of complexity. Curr Opin Plant Biol 2004; 7 : 11–9. 2. Henderson IR, Dean C. Control of Arabidopsis flowering : the chill before the bloom. Development 2004; 131 : 3829–38. 3. Sheldon CC, Rouse DT, Finnegan EJ, et al. The molecular basis of vernalization : the central role of FLOWERING LOCUS C (FLC). Proc Natl Acad Sci USA 2000; 97 : 3753–8. 4. Sung S, Amasino RM. Vernalization in Arabidopsis thaliana is mediated by the PHD finger protein VIN3. Nature 2004; 427 : 159–64. 5. He Y, Amasino RM. Role of chromatin modification in flowering-time control. Trends Plant Sci 2005; 10 : 30–5. 6. Lippman Z, Gendrel AV, Black M, et al. Role of transposable elements in heterochromatin and epigenetic control. Nature 2004; 430 : 471–6. 7. Bastow R, Mylne JS, Lister C, et al. Vernalization requires epigenetic silencing of FLC by histone methylation. Nature 2004; 427 : 164–7. 8. Gendall AR, Levy YY, Wilson A, Dean C. The VERNALIZATION 2 gene mediates the epigenetic regulation of vernalization in Arabidopsis. Cell 2001; 107 : 525–35. 9. Ringrose L, Paro R. Epigenetic regulation of cellular memory by the Polycomb and Trithorax group proteins. Annu Rev Genet 2004; 38 : 413–43. 10. Muller J, Hart CM, Francis NJ, et al. Histone methyltransferase activity of a Drosophila Polycomb group repressor complex. Cell 2002; 111 : 197–208. 11. Kermicle JL. Dependence of the R-mottled aleurone phenotype in maize on mode of sexual transmission. Genetics 1970; 66 : 69–85. 12. Ferguson-Smith A, Lin SP, Tsai CE, et al. Genomic imprinting-insights from studies in mice. Semin Cell Dev Biol 2003; 14 : 43–9. 13. Walter J, Paulsen M. Imprinting and disease. Semin Cell Dev Biol 2003; 14 : 101–10. 14. Gehring M, Choi Y, Fischer RL. Imprinting and seed development. Plant Cell 2004; 16 (suppl) : S203–13. 15. Grossniklaus U, Vielle-Calzada JP, Hoeppner MA, Gagliano WB. Maternal control of embryogenesis by MEDEA, a polycomb group gene in Arabidopsis. Science 1998; 280 : 446–50. 16. Vielle-Calzada JP, Baskar R, Grossniklaus U. Delayed activation of the paternal genome during seed development. Nature 2000; 404 : 91–4. 17. Kinoshita T, Miura A, Choi Y, et al. One-way control of FWA imprinting in Arabidopsis endosperm by DNA methylation. Science 2004; 303 : 521–3. 18. Choi Y, Gehring M, Johnson L, et al. DEMETER, a DNA glycosylase domain protein, is required for endosperm gene imprinting and seed viability in Arabidopsis. Cell 2002; 110 : 33–42. 19. Soppe WJ, Jacobsen SE, Alonso-Blanco C, et al. The late flowering phenotype of FWA mutants is caused by gain-of- function epigenetic alleles of a homeodomain gene. Mol Cell 2000; 6 : 791–802. 20. Colot V, Rossignol JL. Eukaryotic DNA methylation as an evolutionary device. Bioessays 1999; 21 : 402–11. 21. Yoder JA, Walsh CP, Bestor TH. Cytosine methylation and the ecology of intragenomic parasites. Trends Genet 1997; 13 : 335–40. 22. Rabinowicz PD, Palmer LE, May BP, et al. Genes and transposons are differentially methylated in plants, but not in mammals. Genome Res 2003; 13 : 2658–64. 23. Jaenisch R, Bird A. Epigenetic regulation of gene expression : how the genome integrates intrinsic and environmental signals. Nat Genet 2003; 33 (suppl) : 245–54. 24. Martienssen RA, Colot V. DNA methylation and epigenetic inheritance in plants and filamentous fungi. Science 2001; 293 : 1070–4. 25. Cubas P, Vincent C, Coen E. An epigenetic mutation responsible for natural variation in floral symmetry. Nature 1999; 401 : 157–61. 26. Jacobsen SE, Meyerowitz EM. Hypermethylated SUPERMAN epigenetic alleles in Arabidopsis. Science 1997; 277 : 1100–3. 27. Lund AH, van Lohuizen M. Epigenetics and cancer. Genes Dev 2004; 18 : 2315–35. 28. Chong S, Whitelaw E. Epigenetic germline inheritance. Curr Opin Genet Dev 2004; 14 : 692–6. |