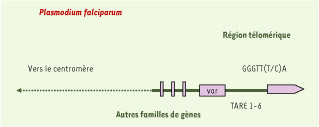
Le paludisme à Plasmodium falciparum reste une cause majeure de morbidité et de mortalité : 300 millions de malades, 2 à 3 millions de décès chaque année. On sait que la virulence est liée à l’adhérence de globules rouges parasités à l’endothélium et entre eux, pour former des rosettes. Cette adhérence est le fait de la protéine de surface PfEMP1 (Plasmodium falciparum erythrocyte membrane protein-1), codée par les gènes de la famille var, qui est aussi un facteur de virulence. Environ 60 gènes var répartis sur les 14 chromosomes du Plasmodium n’expriment jamais qu’une seule protéine par un système de commutation mutuellement exclusive [ 1– 3]. Cette commutation peut atteindre une fréquence de 2 % par génération. Elle a lieu in situ au stade précoce d’anneaux, elle est apparemment contrôlée au niveau de l’initiation de transcription et un mécanisme épigénétique a été évoqué dans cette régulation. Ce phénomène permet une « évasion immunitaire » permanente qui est le problème majeur de toute stratégie vaccinale [ 4]. Les gènes var ont, à plus de 80 %, une localisation subtélomérique. Ils sont séparés du télomère par des séquences répétitives (rep20 ou TARE) en nombre variable (1 à 6) et il y a à leur proximité immédiate d’autres familles multigéniques (rif, stevor, Pf60) (Figures 1 et 2). Quelques gènes var, cependant, ont des localisations internes.
| Figure 1.
Organisation des chromosomes de P. falciparum étudiée par FISH. Il a été établi que les télomères se localisent à la périphérie du noyau et on a constaté une certaine condensation à proximité des télomères. Deux modèles d’organisation de la chromatine sont proposés qui expliqueraient cette condensation : hétérochromatine ou formation de boucles. Les télomères, fixés à la périphérie du noyau, sont représentés en rouge (d’après [
9]). |
Une recherche intense est menée dans de nombreux laboratoires, dont une partie importante à l’Institut Pasteur (Paris, France) par l’équipe que dirige Arthur Scherf. On a montré que les télomères, à proximité desquels sont la majorité des gènes var, s’agglomèrent en 4 à 7 groupes (clusters) à la périphérie du noyau, ce qui pourrait faciliter des recombinaisons ectopiques. Une régulation au niveau de la chromatine a été envisagée qui expliquerait l’extinction d’un gène avec l’activation de l’expression d’un autre gène var [ 5]. Par ailleurs, une recherche menée au Danemark, opérant par des analyses de séquences, a mis en évidence l’existence de sous-groupes de gènes var [ 6]. Les auteurs décrivent trois sous-groupes (A, B, et C) qui se distinguent par leur localisation chromosomique, la direction de transcription, et la structure en domaines de la protéine codée. Le groupe C, en particulier, comporte les gènes var centromériques. Des différences de structure entre les groupes de gènes var se retrouvent au niveau des domaines DBL (duffy binding like) et CIDR (cysteine-rich inter-domain). Les auteurs montrent que les recombinaisons ont lieu préférentiellement à l’intérieur d’un de ces sous-groupes. Ils font l’hypothèse selon laquelle des différences de structure pourraient refléter une diversification fonctionnelle. Les mêmes auteurs ont ensuite mis en évidence l’utilisation préférentielle de gènes télomériques du groupe A dans les formes graves de paludisme chez l’enfant [ 7].
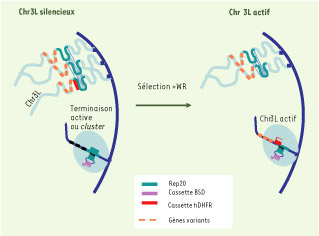
C’est dans ce contexte que vient de paraître une série d’articles qui sont une avancée importante dans la compréhension du mécanisme de régulation des gènes var subtélomériques et de la transcription mutuellement exclusive qui s’effectue in situ. Un travail coopératif, coordonné à Melbourne (Australie), a procédé par comparaison avec des observations faites sur la levure [ 8]. Chez cet eucaryote, on a montré que, dans l’extinction de deux locus HMR et HML, de localisation télomérique, la protéine SIR2 (silent information regulator 2) joue un rôle majeur par désacétylation des histones. Or une protéine homologue, PfSir2, existe chez le Plasmodium. Les auteurs on procédé en insérant entre le télomère et var, dans la région TARE6 du chromosome 3, un transgène hDHFR (dihydrofolate réductase) (Figure 3). Dans cette région subtélomérique, le transgène n’est pas exprimé et se présente donc comme soumis à un contrôle hétérochromatique de même que les gènes var endogènes et le gène rifin. Le rôle de PfSir2 est mis en évidence par le fait que les gènes éteints sont réactivés par interruption de PfSir2 (parasites ∆sir2) en même temps qu’il y a modification de la chromatine et repositionnement dans un site permissif. Le phénomène d’extinction est donc réversible par un mécanisme épigénétique et s’accompagne d’une localisation nucléaire différente, les gènes transcrits n’occupant pas la même position périnucléaire que les gènes éteints.
| Figure 3.
Modèle proposé pour expliquer l’extinction ou l’activation de gènes subtélomériques et leur repositionnement dans le noyau. À l’extrémité du chromosome 3, un transgène hDHFR est inséré dans une région hétérochromatique, entre le gène var et le télomère, et n’est donc pas exprimé. Par ailleurs, un plasmide (BSD) est fixé à la région Rep20 d’un autre chromosome. Son activité transcriptionnelle montre qu’il est situé dans une zone différente de celle du gène silencieux. Une sélection par un antifolate, WR, active la transcription de hDHFR, dont le promoteur se trouve alors dans une conformation ouverte. L’extrémité du chromosome 3 activée se serait délocalisée de la région hétérochromatique dont il faisait partie pour se colocaliser avec le plasmide épisomique dans un compartiment du noyau transcriptionnellement compétent (d’après [8]). |
Une recherche complémentaire, dont les résultats paraissent dans le même numéro de Cell, a été menée par les chercheurs de l’Institut Pasteur et a précisé comment agissent les facteurs épigénétiques qui contrôlent l’expression des gènes var qui sont situés respectivement entre 20 et 50 kb du télomère [9]. Les auteurs individualisent deux facteurs dans la régulation de l’activation ou de l’extinction des gènes : d’une part, une structure d’hétérochromatine, liée à la présence de PfSir2, se propage à partir du télomère ; d’autre part, la réversibilité de l’extinction des gènes est bien liée à l’acétylation des histones. Quand un gène var spécifique est activé, PfSir2 est détaché de la région promotrice du gène, et il y a acétylation des histones. On a observé aussi que des gènes var éteints peuvent être réactivés et transcrits quand on les soustrait de leur contexte chromosomique et qu’on les place sur des épisomes transfectés. En même temps que l’extinction d’un gène sur un chromosome, on constate la diffusion d’une structure hétérochromatique condensée qui part du télomère, s’étend plus ou moins loin sur les régions codantes et s’accompagne d’une désacétylation des promoteurs. Par comparaison avec la levure, on note cependant une différence. Chez la levure, l’effet d’extinction du télomère ne s’étend que sur environ 3 kb alors que, chez le Plasmodium, elle est de l’ordre de 55 kb, atteignant les régions codantes de var et du gène voisin rifin. On constate, sur toute cette longueur du chromosome, un gradient de structure d’hétérochromatine et d’hypoacétylation des histones. La transcription mutuellement exclusive des gènes est donc liée à un remodelage dynamique de la chromatine.
Un dernier article, enfin, a montré que la variation antigénique du Plasmodium s’accompagne d’un déplacement dans la localisation nucléaire [ 10]. Les gènes, à l’état réprimé, sont positionnés à la périphérie du noyau. L’examen de cette zone périphérique du noyau du Plasmodium montre une chromatine condensée dans laquelle on observe quelques brèches de chromatine non condensée. Lors de son activation, la région télomérique où se trouve le gène var se détache du cluster aggloméré dont il faisait partie, il reste à la périphérie, mais est déplacé vers des sites distincts, confirmant ainsi qu’il existe dans les zones périnucléaires des régions permissives pour la transcription (Figure 3).
Comme toujours, une telle série de résultats très informatifs laisse des questions ouvertes [ 11]. Quel est l’élément initiateur de tous ces processus ? Comment sont contrôlés les gènes var qui ne sont pas localisés à proximité des télomères ? Pourquoi, comment l’activation est-elle limitée à un seul gène ? Quels seraient les facteurs d’un contrôle additionnel ? Il reste donc encore à résoudre de multiples problèmes.