| |
| Med Sci (Paris). 2005 August; 21(8-9): 759–764. Published online 2005 August 15. doi: 10.1051/medsci/2005218-9759.Protéomique et auto-anticorps Nadine Machour, Danièle Gilbert, Olivier Vittecoq, Odile Costa, François Tron,* and Roland Charlionet Inserm U.519, Faculté de médecine-pharmacie, 22, boulevard Gambetta, 76183 Rouen Cedex, France |
Le rôle du système immunitaire est de maintenir la cohérence et l’intégrité des organismes multicellulaires vis-à-vis des attaques exogènes (bactérie, virus, parasite) ou endogènes (cancer). Ces événements modifient les structures moléculaires des constituants de l’organisme ou introduisent de nouvelles molécules reconnues par les cellules du système immunitaire. Les lymphocytes T et B reconnaissent spécifiquement ces structures moléculaires, les antigènes, grâce à leurs récepteurs de surface. Chaque cellule lymphocytaire exprime des récepteurs pour l’antigène identiques et spécifiques d’un motif structurel (épitope). Chez un individu, le nombre total de récepteurs exprimés par l’ensemble des lymphocytes constitue le répertoire lymphocytaire. Il est extrêmement divers et produit de manière stochastique par des processus génétiques d’origine somatique. Les lymphocytes T sont responsables de l’immunité cellulaire : ils induisent la destruction intracellulaire des bactéries et lysent les cellules infectées par les virus. Les lymphocytes B sont, quant à eux, responsables de l’immunité humorale : stimulés, ils se différencient, et synthétisent et sécrètent les anticorps qui se lient spécifiquement à leurs cibles antigéniques. La diversité du répertoire est théoriquement illimitée, rendant compte de la capacité des lymphocytes T et B à reconnaître les propres constituants de l’organisme. On conçoit alors qu’une fonction essentielle du système immunitaire est de distinguer les constituants normaux de l’organisme (le soi) des substances étrangères (le non-soi) [
1], et de ne pas élaborer une réponse contre des antigènes qui le constituent : cette passivité immunologique est appelée tolérance. Il s’agit d’un processus, spécifique de l’antigène, acquis par les lymphocytes durant les diverses phases de leur développement ou de leur maturité. La tolérance est induite et maintenue par des mécanismes impliquant des communications bidirectionnelles multiples entre les cellules présentatrices d’antigène, les lymphocytes et les molécules du système immunitaire. Les défauts d’induction ou de maintien de la tolérance entraînent des réponses immunitaires dirigées contre des antigènes du soi (auto-antigènes), et peuvent conduire à des états pathologiques appelés maladies auto-immunes [1,
2]. Il est cependant démontré que les lymphocytes autoréactifs constituent une part substantielle du répertoire normal des lymphocytes : par exemple, ces lymphocytes B sécrètent des auto-anticorps, dits naturels, qui sont détectés dans le sérum des individus normaux et dont la synthèse peut être augmentée dans de nombreuses conditions pathologiques. La plupart des maladies auto-immunes sont donc caractérisées par la production d’auto-anticorps spécifiques de la situation pathologique et par la modification du répertoire des auto-anticorps naturels. Certains de ces auto-anticorps sont directement responsables des lésions tissulaires. Par conséquent, l’analyse du répertoire des auto-anticorps constitue une étape essentielle de l’étude des processus physiopathologiques à l’œuvre dans les maladies auto-immunes : elle permet d’identifier les auto-antigènes et les modifications qu’ils subissent au cours de ces maladies, de caractériser de nouvelles populations d’auto-anticorps et de découvrir de nouveaux marqueurs biologiques des maladies auto-immunes. Ces analyses sont classiquement réalisées à partir de tests d’immunofluorescence indirecte [
3] et ELISA [
4]. L’identification des antigènes cibles a connu un large essor dans les années 1990, grâce à l’immunocriblage des banques d’expression d’ADNc dans les cellules bactériennes [
5]. Aujourd’hui, des stratégies méthodologiques, faisant appel aux technologies du génie génétique, de l’analyse protéomique ciblée, de la robotique et de l’informatique, ont été développées pour proposer des approches diagnostiques non plus fondées sur un petit nombre de marqueurs, mais sur l’analyse des modifications globales du répertoire des auto-anticorps. Nous décrivons trois d’entre elles et comparons leurs mérites respectifs. |
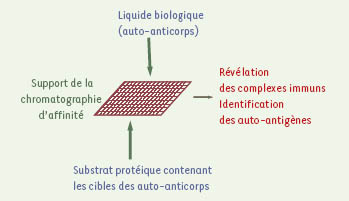
Les trois méthodes d’exploration du répertoire des auto-anticorps présentées ici reposent toutes sur l’isolement des cibles des auto-anticorps, les auto-antigènes, à partir d’une séparation par chromatographie d’affinité fondée sur la réaction antigène/anticorps. Cette chromatographie d’affinité se déroule en deux temps : il y a d’abord fixation des antigènes (ou des anticorps) sur un support approprié, puis filtration de la solution contenant les auto-anticorps (ou les auto-antigènes) sur ce support affin. La Figure 1 illustre les quatre éléments techniques principaux à examiner. Le premier concerne la solution d’auto-anticorps, dont la source est constituée par les liquides physiologiques, sérum, liquide céphalorachidien, liquide synovial. La concentration en anticorps est un point crucial, car les trois approches analytiques proposées n’ont pas toutes les mêmes besoins quantitatifs. La deuxième considération technique concerne le substrat, généralement de nature protéique : il doit théoriquement contenir toutes les cibles potentielles des auto-anticorps, car l’exploration du répertoire sera d’autant plus exhaustive que le nombre de cibles potentielles présentes dans le substrat sera grand. Le troisième élément concerne la réalisation de la chromatographie d’affinité. Celle-ci a pour support une surface sur laquelle se déroulent les réactions antigène/anticorps : il peut s’agir de membranes de plusieurs dizaines de cm2, ou de puces microscopiques en verre ou polymère ; la taille de cette surface détermine la quantité nécessaire de réactifs. Les réactions immunochimiques ont lieu sur la surface affine et permettent de retenir, par simple filtration des solutions d’auto-anticorps (ou de substrat protéique), les auto-anticorps (ou les auto-antigènes) spécifiques. Un même principe est donc commun à chacune des approches et les rend bien adaptées à l’analyse du répertoire des auto-anticorps. Cependant, les trois méthodologies diffèrent par la nature de ce qui est fixé en premier sur la surface (les anticorps ou les cibles protéiques potentielles), par la manière de réaliser cet accrochage, en vrac ou dans l’ordre, et par l’utilisation de conditions expérimentales éventuellement dénaturantes pour les antigènes, ce qui a des conséquences sur les liaisons qui doivent s’établir. Enfin, le quatrième et dernier élément technique concerne la révélation des complexes immuns et l’identification des auto-antigènes. Sa mise en œuvre diffère considérablement d’une technologie à l’autre : s’il s’agit d’une étape relativement simple pour les technologies comportant une présentation ordonnée des cibles protéiques préalablement à la chromatographie d’affinité, c’est une étape délicate pour les autres, impliquant des séparations et identifications liées à la spectrométrie de masse. 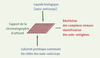 | Figure 1.
Analyse du répertoire des auto-anticorps. Les auto-anticorps à étudier sont mis au contact du substrat protéique contenant les cibles auto-antigéniques. Les interactions antigène/anticorps ont lieu sur un support de chromatographie d’affinité de taille macro- ou microscopique (filtre membranaire ou puce) qui fixe les complexes immuns. Plusieurs stratégies sont utilisées pour repérer les complexes immuns et pour identifier la cible des auto-anticorps. |
|
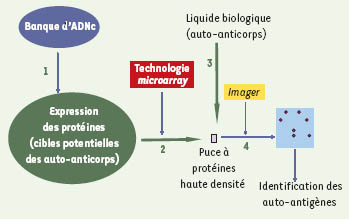
Puce à protéines recombinantes Cette approche est illustrée dans la Figure 2. La surface affine, qui permet la liaison des auto-anticorps, est constituée par une puce à protéines recombinantes [
6]. Des centaines, voire des milliers de protéines purifiées non redondantes, supposées être des cibles potentielles des auto-anticorps, sont disposées de manière ordonnée à la surface d’une puce. L’élaboration de cette surface affine nécessite l’emploi d’outils de clonage directionnel de gènes conduisant à l’expression et la purification d’un très grand nombre de protéines recombinantes, ainsi que l’utilisation de robots adaptés à la distribution de nanoquantités de solutions de ces protéines [
7–
9]. La maîtrise de ces techniques représente la principale difficulté de cette approche et explique que sa mise en œuvre est réservée à quelques laboratoires spécialisés. Lueking et al. [
10] décrivent deux applications principales des puces à protéines recombinantes : l’étude de la sélectivité et, à l’inverse, des réactivités croisées et de la polyspécificité des anticorps, d’une part, et la caractérisation du profil du répertoire des auto-anticorps dans les liquides physiologiques chez des patients atteints de différentes maladies auto-immunes (alopécies et polyarthrite rhumatoïde), d’autre part. 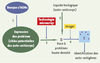 | Figure 2.
Puce à protéines recombinantes. Une banque d’ADNc préparée à partir de cerveau humain fœtal est directionnellement clonée dans un vecteur bactérien qui permet l’expression des protéines (étape 1). Les centaines ou milliers de protéines recombinantes non redondantes constituent ainsi le substrat protéique susceptible de contenir les cibles des auto-anticorps. Elles sont disposées isolément à l’aide d’un robot et fixées sur la surface chimiquement active d’une puce (étape 2). Celle-ci se comporte alors comme une surface affine vis-à-vis des auto-anticorps contenus dans le liquide physiologique à analyser (étape 3), qui est ainsi filtré par la puce. Après incubation avec des anticorps secondaires couplés à un traceur, l’observation de la puce à l’aide d’un Imager permet de repérer la localisation des spots immunoréactifs et donc d’identifier directement les auto-antigènes (étape 4). |
Les avantages de cette technologie sont la facilité d’observation et d’identification des auto-antigènes, ainsi que la faible quantité de liquide biologique nécessaire à l’analyse, qui en font un outil idéal pour étudier de façon exhaustive le répertoire des auto-anticorps et comparer le répertoire d’un grand nombre d’échantillons biologiques [6]. Cependant, c’est au niveau du substrat protéique contenant les cibles antigéniques que réside la limite. Outre la complexité de sa mise en œuvre [
11], l’obtention d’un panel suffisamment large d’auto-antigènes recombinants reste problématique : en effet, la plupart des vecteurs d’expression protéique utilisés (bactéries, levures) ne permettent pas la synthèse de protéines ayant des modifications post-traductionnelles (notamment phosphorylation ou glycosylation) identiques à celles des mammifères, ce qui peut entraîner une altération de l’antigénicité. En outre, les protéines de grande taille sont difficilement exprimées par les vecteurs et l’expression de protéines tronquées, voire la non-expression de protéines, due au décalage du cadre de lecture, sont à l’origine d’une perte de cibles antigéniques [
12]. |
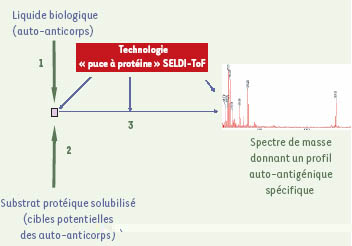
Technologie SELDI (surface-enhanced laser desorption/ionisation) Cette technologie, développée par la société Ciphergen, est illustrée dans la Figure 3. La surface affine est ici constituée par une puce à auto-anticorps. Par simple filtration du substrat protéique solubilisé, les auto-antigènes cibles se lient à la puce [
13]. Fixés de façon non ordonnée, ils sont ensuite dissociés, rangés dans l’ordre de leur masse moléculaire et détectés par une technique de spectrométrie de masse SELDI-ToF (time of flight) mise au point par Ciphergen [
14]. Les spectres obtenus constituent alors des profils auto-antigéniques caractéristiques de la solution d’auto-anticorps étudiée, c’est-à-dire spécifiques de certains états pathologiques auto-immuns. Cependant, l’identification des auto-antigènes nécessite une étape méthodologique supplémentaire comportant une digestion enzymatique des protéines, directement sur la puce, suivie du séquençage peptidique par spectrométrie de masse en tandem.  | Figure 3.
Technologie SELDI (surface-enhanced laser desorption/ionisation). La solution d’auto-anticorps à analyser est incubée avec une puce à protéine A, fournie par Ciphergen, qui a la propriété de fixer spécifiquement les immunoglobulines de classe G. Cette puce à anticorps est ensuite incubée avec une solution contenant les protéines susceptibles d’être les cibles des auto-anticorps (étape 2). Les auto-antigènes, fixés sur la puce de manière non ordonnée, peuvent cependant être rangés dans l’ordre de leur masse moléculaire en appliquant directement sur la puce la technologie de spectrométrie de masse SELDI-ToF (time of flight) (étape 3). |
La technologie SELDI est un outil de recherche clinique déjà très utilisé et particulièrement bien adapté à la mise en évidence de biomarqueurs spécifiques de divers états pathologies [
15]. Cette approche a été récemment proposée pour l’exploration comparative des répertoires d’auto-anticorps [13] et elle est, sans doute, promise à un grand avenir dans cette application. En effet, elle utilise des substrats, dans des conditions non dénaturantes, dont la diversité des cibles auto-antigéniques n’est pas limitée par la technologie elle-même. Sa principale limite réside dans les difficultés d’identification et de caractérisation structurale des auto-antigènes reconnus [
16] : les auto-antigènes ne sont en effet pas ordonnés avant l’étape de chromatographie d’affinité, et leur identification nécessite, dans l’état actuel de la technologie, de mettre en œuvre un travail d’analyse supplémentaire complexe. Par ailleurs, il est important de signaler que des systèmes de couplage de la chromatographie d’affinité à la spectrométrie de masse Maldi-Tof (matrix assisted laser desorption ionization) commencent à apparaître. La société Bruker Daltonics, notamment, a créé un tel appareil intégré, dont la partie chromatographique est constituée par des perles magnétiques spécialement traitées pour mettre en œuvre des interactions hydrophobes [
17]. Lorsque cette technologie sera complètement développée, elle pourra être utilisée, de manière tout à fait semblable à la technologie SELDI, pour l’analyse du répertoire des auto-anticorps. |
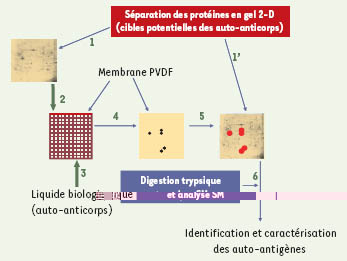
Immunocriblage de gel 2-D couplé à la spectrométrie de masse Cette approche est illustrée dans la Figure 4. La surface affine permet la fixation des auto-anticorps que l’on veut caractériser. Elle est constituée par les protéines du substrat contenant les cibles potentielles des auto-anticorps, séparées par électrophorèse bidimensionnelle et transférées sur une membrane immobilisante (par exemple, membrane de PVDF, polyvinylidene fluoride) de quelques dizaines de cm2. Après filtration de la solution d’auto-anticorps à étudier par la membrane affine, les anticorps secondaires couplés à un traceur permettent de repérer les spots immunoréactifs. Les protéines correspondantes sont localisées sur une réplique du gel bidimensionnel. L’identification et la caractérisation des auto-antigènes sont alors réalisées par spectrométrie de masse après digestion enzymatique de la protéine in gel.  | Figure 4.
Immunocriblage de gel 2-D couplé à la spectrométrie de masse. Le substrat protéique susceptible de contenir des cibles auto-antigéniques est soumis à une séparation par électrophorèse bidimensionnelle (étape 1), qui permet de séparer de manière ordonnée plusieurs centaines, voire plusieurs milliers de polypeptides, visualisés par des colorants classiques. Comme cette technique séparative est très reproductible, des duplicata de gel 2-D peuvent être obtenus (étape 1’). Les polypeptides séparés sont transférés sur une membrane immobilisante (étape 2) qui se comporte alors comme un filtre affin vis-à-vis des auto-anticorps (étape 3). Les spots immunoréactifs sont révélés à l’aide d’anticorps secondaires marqués (étape 4) et les polypeptides auto-antigéniques correspondants sont repérés sur le gel 2-D dupliqué (étape 5). L’identification et la caractérisation des spots auto-antigéniques sont réalisées, à partir du gel dupliqué, après digestion enzymatique in gel et analyse par spectrométrie de masse (étape 6). PVDF : polyvinylidene fluoride ; SM : spectrométrie de masse.
|
Les auto-anticorps produits au cours des maladies auto-immunes peuvent être dirigés contre des épitopes dont l’antigénicité est apportée par des modifications post-traductionnelles. Or, la reconnaissance des modifications post-traductionnelles est pratiquement réalisée de manière optimale et sans a priori par l’association classique de l’électrophorèse bidimensionnelle et la spectrométrie de masse : les gels 2-D révélant les modifications qui altèrent le point isoélectrique et la masse moléculaire, tandis que l’analyse en spectrométrie de masse permet de préciser la nature structurelle et la position de la modification sur la séquence protéique [
18]. Cela constitue le principal avantage et l’originalité de cette technologie, largement utilisée pour l’exploration du répertoire des auto-anticorps dans diverses pathologies et pour l’analyse des processus physiopathologiques à l’œuvre dans les maladies auto-immunes [
19–
23]. Son principal inconvénient réside dans la lourdeur de sa mise œuvre et de sa standardisation. On peut alors utiliser une variante de cette technologie en substituant la séparation du substrat protéique utilisant l’électrophorèse bidimensionnelle par la séparation du substrat à l’aide d’une électrophorèse en gel de polyacrylamide en présence de sodium dodécyle sulfate (SDS-PAGE). Les protéines sont alors séparées selon le seul critère de masse moléculaire, et les protéines trop hydrophobes ou de pI extrême échappant à l’analyse par électrophorèse bidimensionnelle peuvent être récupérées. Il est ainsi possible de traiter en même temps un grand nombre de substrats protéiques et de les immunocribler par un grand nombre de solutions d’auto-anticorps. Les profils auto-antigéniques produits sont analysés par des outils statistiques à variables multiples qui, de manière tout à fait similaire à l’approche SELDI, rendent cette technologie particulièrement adaptée à la recherche clinique [
24–
27]. |
Les trois méthodes d’exploration du répertoire des auto-anticorps actuellement utilisables ont en commun de disposer d’une surface affine permettant de retenir, par simple filtration du liquide biologique à étudier, les auto-anticorps (ou les auto-antigènes) spécifiques. Pour une pratique courante, on peut écarter l’approche par puce à protéines recombinantes, qui semble être réservée aux laboratoires disposant de la maîtrise de techniques haut-débit de clonage directionnel de gènes, permettant l’expression et la purification d’un très grand nombre de protéines recombinantes. La technologie SELDI et celle de l’immunocriblage de gels 2-D couplé à la spectrométrie de masse, ainsi que sa variante SDS-PAGE, sont en revanche utilisables dans la plupart des laboratoires de recherches biologiques actuels. Leurs avantages et inconvénients ont été décrits. Il est important d’insister sur le rôle de la composition quantitative et qualitative du substrat dans l’exploration du répertoire des auto-anticorps. Celui-ci doit théoriquement contenir toutes les cibles potentielles de la réponse auto-immune. En outre, les auto-anticorps peuvent être dirigés contre des épitopes dont l’antigénicité est apportée par des modifications post-traductionnelles, par exemple des protéines citrullinées dans le cas de la polyarthrite rhumatoïde. Leur mise en évidence nécessite de disposer de substrats contenant ces protéines modifiées. L’analyse du répertoire des auto-anticorps passe donc par une réflexion approfondie préalable quant au choix des substrats protéiques, dont la sélection se fera selon la maladie auto-immune étudiée. |
1. Schwartz RS. Autoimmunity and autoimmune diseases. In : Paul WE, ed. Fundamental immunology. New York : Raven Press, 1993 : 1033–97. 2. Oppezzo P, Dighiero G. Autoantibodies, tolerance and autoimmunity. Pathol Biol 2003; 51 : 297–304. 3. Holman HR, Kunkel HG. Affinity between the lupus erythematosus serum factor and cell nuclei and nucleoprotein. Science 1957; 126 : 162–3. 4. Ulvestad E, Kanestrom A, Madland TM, et al. Evaluation of diagnostic tests for antinuclear antibodies in rheumatological practice. Scand J Immunol 2000; 52 : 309–15. 5. Sahin U, Tureci O, Pfreundschuh M. Serological identification of human tumor antigens. Curr Opin Immunol 1997; 9 : 709–16. 6. Robinson WH, Steinman L, Utz PJ. Protein arrays for autoantibody profiling and fine-specifcity mapping. Proteomics 2003; 3 : 2077–84. 7. MacBeath G, Schreiber SL. Printing proteins as microarrays for high-trhoughput function determination. Science 2000; 289 : 1760–3. 8. Lueking A, Horn M, Eickhoff H, et al. Protein microarrays for gene expression and antibody screening. Anal Biochem 1999; 270 : 103–11. 9. Angenendt P, Glökler J, Sobek J, et al. Next generation of protein microarray support materials: evaluation for protein and antibody microarray applications. J Chromatogr A 2003; 1009 : 97–104. 10. Lueking A, Possling A, Huber O, et al. A non-redundant human protein chip for antibody screening and serum profiling. Mol Cell Proteomics 2003; 12 : 1342–9. 11. Schmitt J, Papisch W. Recombinant autoantigens. Autoimmun Rev 2002; 1 : 79–88. 12. Makrides SC. Strategies for achieving high-level expression of genes in E. coli.
Microbiol Rev 1996; 60 : 512–38. 13. Grus FH, Joachim SC, Pfeiffer N. Analysis of complex autoantibody repertoires by surface-enhanced desorption/ionisation-time of flight mass spectrometry. Proteomics 2003; 3 : 957–61. 14. Merchant M, Weinberger SR. Recent advancements in surface-enhanced laser desorption/ionisation-time of flight-mass spectrometry. Electrophoresis 2000; 21 : 1164–77. 15. Gravett MG, Novy MJ, Rosenfeld RG, et al. Diagnosis of intra-amniotic infection by proteomic profiling and identification of novel biomarkers. JAMA 2004; 292 : 462–9. 16. Caputo E, Moharram R, Martin BM. Methods for on-chip protein analysis. Anal Biochem 2003; 321 : 116–24. 17. Zhang X, Leung S-M, Morris CR, Shigenaga MK. Evaluation of a novel, integrated approach using functionnalized magnetic beads, bench-top Maldi-Tof MS with prestructured sample supports, and pattern recognition software for profiling potential biomarkers in human plasma. J Biomol Techn 2004; 15 : 167–75. 18. Rabilloud T. Two-dimensional gel electrophoresis in proteomics: old, old fashioned, but it still climbs up the mountains. Proteomics 2002; 2 : 3–10. 19. Thebault S, Gilbert D, Machour N, et al. Two-dimensional electrophoresis and mass spectrometry identification of prtoteins bound by a murine monoclonal anti-cardiolipin antibody: a powerful technique to characterize the cross-reactivity of a single autoantibody. Electrophoresis 2000; 21 : 2531–9. 20. LeNaour F, Brichory F, Misek DE, et al. A distinct repertoire of autoantibodies in hepatocellular carcinoma identified by proteomic analysis. Mol Cell Proteomics 2002; 1 : 197–203. 21. Thebault S, Gilbert D, Hubert M, et al. Orderly pattern of development of the autoantibody response in (NZWxBXSB)F1 lupus mice: characterization of target antigens and antigen spreading by two-dimensional gel electrophoresis and mass spectrometry. J Immunol 2002; 169 : 4046–53. 22. Saulot V, Vittecoq O, Charlionet R, et al. Presence of autoantibodies to the glycolytic enzyme alpha-enolase in sera from patients with early RA. Arthr Rheum 2002; 46 : 1196–1201. 23. Unwin RD, Harnden P, Pappin D, et al. Serological and proteomic evaluation of antibody responses in the identification of tumor antigens in renal cell carcinoma. Proteomics 2003; 3 : 45–55. 24. Latif N, Baker CS, Dunn MJ, et al.Frequency and specificity of antiheart antibodies in patients with dilated cardiomyopathy detected using SDS-PAGE and western blotting. J Am Coll Cardiol 1993; 22 : 1378–84. 25. Pohlner K, Portig I, Pankuweit S, et al. Identification of mitochondrial antigens recognized by antibodies in sera of patients with idiopathic dilated cardiomyopathy by two-dimensional gel electrophoresis and protein sequencing. Am J Cardiol 1997; 80 : 1040–5. 26. Almeras L, Lefranc D, Drobecq H, et al. New antigenic candidates in multiple sclerosis: a serological proteomic analysis. Proteomics 2004; 7 : 2184–94. 27. Lefranc D, Almeras L, Dubucquoi S, et al. Distortion of the self-reactive IgG antibody repertoire in multiple sclerosis as a new diagnostic tool. J Immunol 2004; 172 : 669–78. |