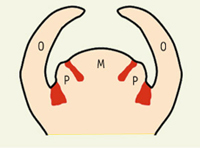
Chez les plantes, tous les organes latéraux de la partie aérienne, qu’il s’agisse de feuilles ou de pièces florales comme les pétales ou les étamines, sont issus des méristèmes. Ces structures en prolifération contiennent les cellules souches de la plante et sont le siège d’une organogenèse continue. Au sein des méristèmes, suivant un minutage et à une position finement contrôlés, un groupe de cellules va s’individualiser, bourgeonner en un primordium et donner naissance à un organe (Figure 1). Les primordia d’organes sont séparés entre eux et du méristème par un domaine frontière formé par un ruban de cellules. Les cellules appartenant à ces domaines sont reconnaissables par les gènes qu’elles expriment. Ainsi, chez la plante modèle Arabidospis thaliana, les cellules des domaines frontières expriment les gènes CUC1, 2 et 3 [ 1– 3]. Ces marqueurs moléculaires des domaines frontières en sont aussi des déterminants majeurs, puisque l’inactivation d’un ou de plusieurs d’entre eux conduit à des défauts de spécification du domaine frontière entraînant des fusions entre organes. Par exemple, le double mutant cuc1 cuc2 se caractérise par une fusion des cotylédons, leur donnant une apparence de coupe (d’où le nom des gènes CUP-SHAPED COTYLEDON, cotylédon en forme de coupe), et une fusion entre sépales1, et entre étamines2. Les gènes CUC1, 2 et 3 codent pour des protéines qui font partie de la famille des facteurs de transcription de type NAC, comprenant chez Arabidopsis plus d’une centaine de membres. Bien que très proches, ces trois gènes se distinguent néanmoins par l’existence d’un site de fixation d’un microARN (miARN), miR164, sur les transcrits des gènes CUC1 et CUC2, ce site étant absent du transcrit de CUC3.
Les miARN, identifiés à la fois chez les animaux et les végétaux, sont de petits ARN simple brin d’environ 21 nucléotides qui règlent de façon post-transcriptionnelle l’expression de gènes cibles avec lesquels ils présentent une complémentarité de séquence [ 4]. Chez les plantes, les miARN ont été identifiés physiquement par séquençage de banques de petits ARN ou prédits par différents algorithmes bio-informatiques. Parallèlement, les cibles potentielles de ces miARN ont été prédites grâce à la complémentarité de séquence entre le miARN et ses cibles. Ainsi, miR164 pourrait cibler 6 facteurs de transcription de la famille NAC, dont CUC1 et CUC2, et serait produit à partir de 3 gènes MIR164A, B et C [ 5] (Figure 2).
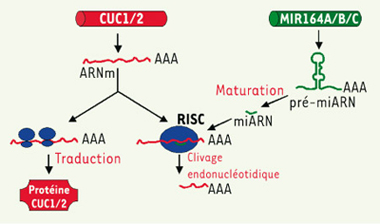
| Figure 2.
Régulation des gènes CUC1 et 2 par miR164. Le microARN, miR164, est un inhibiteur de l’expression des gènes CUC1 et 2. En l’absence de miR164, les gènes CUC1 et 2 sont transcrits en ARNm qui eux-mêmes sont traduits en protéines. La transcription des gènes MIR164A, B et C produit des pré-miARN qui, après plusieurs étapes de maturation, donnent naissance au miARN, miR164. Ce dernier, au sein d’un complexe protéique appelé RISC (RNA-induced silencing complex), reconnaît les transcrits des gènes CUC1 et 2 et provoque leur clivage endonucléotidique. Les deux produits ainsi engendrés sont dégradés. |
Ces prédictions ont été validées expérimentalement de plusieurs façons. La plupart des miARN végétaux contrôlent l’expression de leurs cibles en induisant le clivage de leurs transcrits entre les nucléotides appariés aux 10e et 11e nucléotides du miARN. La détection des produits de clivage peut alors servir de trace de l’action du miARN. De tels produits de clivage ont été effectivement observés pour CUC1 et CUC2 [ 6]. Le séquençage de l’extrémité du fragment 3’ a révélé que le clivage avait eu lieu au niveau du site de fixation de miR164, impliquant ainsi directement ce miARN dans ce processus. De plus, miR164 est effectivement produit par les 3 gènes MIR164A, B et C, puisque leur surexpression provoque une augmentation du contenu en miR164, ce qui entraîne une diminution du niveau d’accumulation des transcrits des gènes CUC1 et 2. Comme attendu, la réduction de l’expression des gènes CUC conduit à des anomalies développementales embryonnaires ou florales similaires à celles des doubles mutants cuc1 cuc2 [ 7– 10] (Figure 3).
Se pose ensuite la question du rôle de la régulation des gènes CUC1 et CUC2 par miR164 au cours du développement. Pour y répondre, une démarche classique de génétique a été suivie, visant à perturber ou à bloquer cette régulation et à en analyser les conséquences. La première approche utilisée fut d’exprimer des versions de CUC1 ou CUC2 résistantes à l’action du miARN. Ces versions résistantes ont été créées en introduisant des mutations dans le gène cible au niveau du site de fixation du miARN, empêchant ainsi la formation de duplex miARN/cible. Pour ne pas modifier en même temps la fonctionnalité de la protéine, on a bien pris garde de n’introduire que des mutations silencieuses sans effet sur la séquence protéique. L’expression des formes résistantes à miR164 de CUC1 ou 2 perturbe le développement de la plante, alors que l’expression de formes non modifiées de ces gènes reste sans effet, soulignant l’importance de la régulation de ces deux gènes par miR164. Plus précisément, les plantes exprimant une forme modifiée de CUC1 produisent un nombre anormal d’organes floraux [7, 9]. De telles modifications du nombre d’organes ne sont pas observées chez des plantes exprimant une version miR164-résistante de CUC2, qui montrent quant à elles un élargissement du domaine frontière [ 8]. Cet élargissement est visible morphologiquement, en particulier au niveau des sépales. Il est aussi directement visible au niveau cytologique au cours du développement de la fleur grâce à l’expression d’une protéine fluorescente, la GFP, dans les cellules faisant partie du domaine frontière. Un tel élargissement des frontières est également observé chez des mutants affectés dans la biogenèse des miARN et qui présentent une accumulation réduite de miR164 [8]. L’analyse de ces mutants, bien que confirmant le rôle de miR164 dans la stabilisation des frontières, reste délicate car un grand nombre, sinon la totalité, des miARN y sont modifiés, conduisant à une large gamme de perturbations. L’alternative est d’analyser des mutants affectés spécifiquement dans les gènes MIR164. La première mutation décrite inactive le gène MIR164B mais, bien que réduisant fortement la teneur en miR164, elle ne conduit à aucun défaut décelable dans le fonctionnement du méristème, suggérant que les deux autres gènes MIR164A et C toujours fonctionnels sont suffisants [7]. Au contraire, l’inactivation partielle du gène MIR164C conduit à une augmentation du nombre de pétales comme cela avait déjà été observé chez des plantes exprimant une forme miR164-résistante de CUC1 [9]. Ces modifications sont dues à une expression plus forte des gènes CUC1 et CUC2 et à une fragmentation de leur domaine d’expression, ce qui serait la cause de la formation des organes surnuméraires. Néanmoins, dans ce mutant, le domaine d’expression des gènes CUC n’est pas élargi, ce qui suggère que MIR164C ne limiterait pas l’expansion du domaine frontière mais contrôlerait le niveau d’accumulation des transcrits CUC au sein de ce domaine. Cette hypothèse est renforcée par l’observation que MIR164C est exprimé dans un domaine chevauchant celui des gènes CUC.
L’ensemble de ces observations suggère deux mécanismes par lesquels miR164 contribue à stabiliser les frontières. Le premier, révélé par le phénotype des plantes exprimant une version miR164-résistante de CUC2, est de limiter l’expansion des frontières. Le second, mis en lumière par le mutant mir164c, est de tamponner l’expression des gènes CUC1 et 2 au sein même des domaines frontières. Des questions subsistent néanmoins : quel est le rôle des gènes MIR164A et B ? Et quelles seraient les conséquences sur la plante d’une absence totale de miR164 ?
L’analyse du rôle de miR164 a mis en évidence un nouveau niveau de régulation du développement des domaines frontières dans le méristème et illustre le rôle des miARN dans la régulation de gènes-clés du développement. Dans le cas de miR164, la régulation est probablement très ancienne, puisque des homologues des gènes CUC1 et 2 possédant un site de fixation de miR164 et miR164 lui-même sont présents chez d’autres dicotylédones, des monocotylédones et même des gymnospermes3, [7, 11]. Cette conservation et le degré de redondance génique laissent penser que les domaines frontières pourraient avoir un rôle plus important que la seule séparation des organes. Un tel rôle reste toutefois à découvrir.