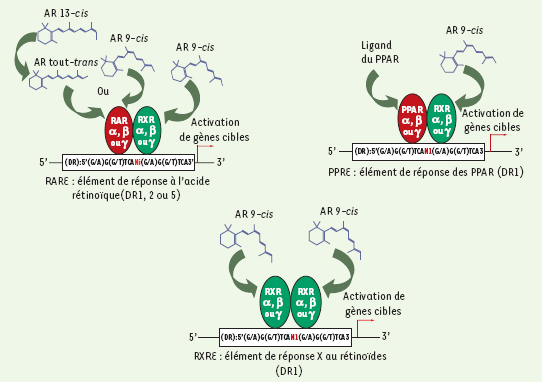
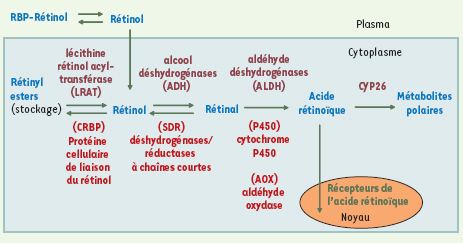
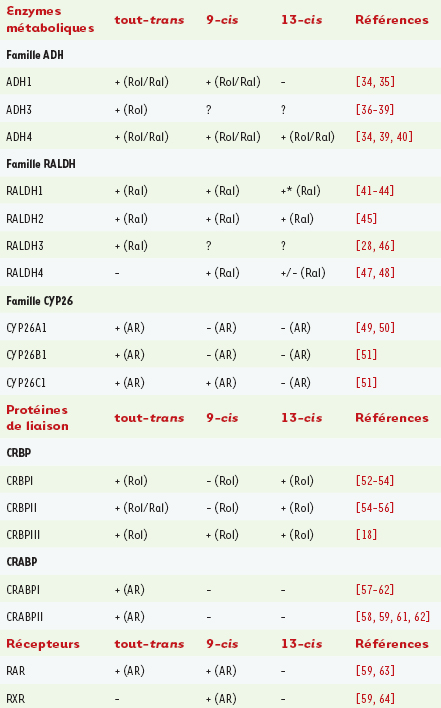
1.
Napoli JL. Interactions of retinoid binding proteins and enzymes in retinoid metabolism. Biochim Biophys Acta 1999; 1440 : 139–62.
2.
Armstrong JL, Redfern CP, Veal GJ. 13-cis retinoic acid and isomerisation in paediatric oncology--is changing shape the key to success ? Biochem Pharmacol 2005; 69 : 1299–306.
3.
Giguere V. Retinoic acid receptors and cellular retinoid binding proteins: complex interplay in retinoid signaling. Endocrinol Rev 1994; 15 : 61–79.
4.
Chambon P. A decade of molecular biology of retinoic acid receptors. FASEB J 1996; 10 : 940–54.
5.
Shulman AI, Larson C, Mangelsdorf DJ, et al. Structural determinants of allosteric ligand activation in RXR heterodimers. Cell 2004; 116 : 417–29.
6.
Miller WH Jr. The emerging role of retinoids and retinoic acid metabolism blocking agents in the treatment of cancer. Cancer 1998; 83 : 1471–82.
7.
Petkovich PM. Retinoic acid metabolism. J Am Acad Dermatol 2001; 45 : S136–42.
8.
Sun SY, Lotan R. Retinoids and their receptors in cancer development and chemoprevention. Crit Rev Oncol Hematol 2002; 41 : 41–55.
9.
Freemantle SJ, Spinella MJ, Dmitrovsky E. Retinoids in cancer therapy and chemoprevention: promise meets resistance. Oncogene 2003; 22 : 7305–15.
10.
Berard J, Laboune F, Mukuna M, et al. Lung tumors in mice expressing an antisense RARbeta2 transgene. FASEB J 1996; 10 : 1091–7.
11.
Pitha-Rowe I, Petty WJ, Kitareewan S, et al. Retinoid target genes in acute promyelocytic leukemia. Leukemia 2003; 17 : 1723–30.
12.
Rigas JR, Dragnev KH. Emerging role of rexinoids in non-small cell lung cancer: focus on bexarotene. Oncologist 2005; 10 : 22–33.
13.
Duester G, Mic FA, Molotkov A. Cytosolic retinoid dehydrogenases govern ubiquitous metabolism of retinol to retinaldehyde followed by tissue-specific metabolism to retinoic acid. Chem Biol Interact 2003; 143–144 : 201–10.
14.
Abu-Abed S, Dolle P, Metzger D, et al. The retinoic acid-metabolizing enzyme, CYP26A1, is essential for normal hindbrain patterning, vertebral identity, and development of posterior structures. Genes Dev 2001; 15 : 226–40.
15.
Yashiro K, Zhao X, Uehara M, et al. Regulation of retinoic acid distribution is required for proximodistal patterning and outgrowth of the developing mouse limb. Dev Cell 2004; 6 : 411–22.
16.
Niederreither K, Abu-Abed S, Schuhbaur B, et al. Genetic evidence that oxidative derivatives of retinoic acid are not involved in retinoid signaling during mouse development. Nat Genet 2002; 31 : 84–8.
17.
Abu-Abed S, Dolle P, Metzger D, et al. Developing with lethal RA levels: genetic ablation of Rarg can restore the viability of mice lacking Cyp26a1. Development 2003; 130 : 1449–59.
18.
Vogel S, Mendelsohn CL, Mertz JR, et al. Characterization of a new member of the fatty acid-binding protein family that binds all-trans-retinol. J Biol Chem 2001; 276 : 1353–60.
19.
Ghyselinck NB, Bavik C, Sapin V, et al. Cellular retinol-binding protein I is essential for vitamin A homeostasis. EMBO J 1999; 18 : 4903–14.
20.
Lampron C, Rochette-Egly C, Gorry P, et al. Mice deficient in cellular retinoic acid binding protein II (CRABPII) or in both CRABPI and CRABPII are essentially normal. Development 1995; 121 : 539–48.
21.
Stoppie P, Borgers M, Borghgraef P, et al. R115866 inhibits all-trans-retinoic acid metabolism and exerts retinoidal effects in rodents. J Pharmacol Exp Ther 2000; 293 : 304–12.
22.
Armstrong JL, Ruiz M, Boddy AV, et al. Increasing the intracellular availability of all-trans retinoic acid in neuroblastoma cells. Br J Cancer 2005; 92 : 696–704.
23.
Bhat PV, Lacroix A. Metabolism of retinol and retinoic acid in N-methyl-N-nitrosourea-induced mammary carcinomas in rats. Cancer Res 1989; 49 : 139–44.
24.
Hayden LJ, Satre MA. Alterations in cellular retinol metabolism contribute to differential retinoid responsiveness in normal human mammary epithelial cells versus breast cancer cells. Breast Cancer Res Treat 2002; 72 : 95–105.
25.
Farias EF, Ong DE, Ghyselinck NB, et al. Cellular retinol-binding protein I, a regulator of breast epithelial retinoic acid receptor activity, cell differentiation, and tumorigenicity. J Natl Cancer Inst 2005; 97 : 21–9.
26.
Mira YLR, Zheng WL, Kuppumbatti YS, et al. Retinol conversion to retinoic acid is impaired in breast cancer cell lines relative to normal cells. J Cell Physiol 2000; 185 : 302–9.
27.
Paik J, Blaner WS, Swisshelm K. Cis-retinol dehydrogenase: 9-cis-retinol metabolism and its effect on proliferation of human MCF7 breast cancer cells. Exp Cell Res 2005; 303 : 183–96.
28.
Rexer BN, Zheng WL, Ong DE. Retinoic acid biosynthesis by normal human breast epithelium is via aldehyde dehydrogenase 6, absent in MCF-7 cells. Cancer Res 2001; 61 : 7065–70.
29.
Kim H, Lapointe J, Kaygusuz G, et al. The retinoic acid synthesis gene ALDH1a2 is a candidate tumor suppressor in prostate cancer. Cancer Res 2005; 65 : 8118–24.
30.
Dragani TA, Falvella FS, Manenti G, et al. Downexpression of aldehyde dehydrogenase 1 in murine lung tumors. Mol Carcinog 1996; 16 : 123–5.
31.
Jette C, Peterson PW, Sandoval IT, et al. The tumor suppressor adenomatous polyposis coli and caudal related homeodomain protein regulate expression of retinol dehydrogenase L. J Biol Chem 2004; 279 : 34397–405.
32.
Cain JM, Zaino R, Shearer D, et al. Expression of a retinol dehydrogenase (hRoDH-4), a member of the retinol/steroid dehydrogenase family implicated in retinoic acid biosynthesis, in normal and neoplastic endometria. Am J Obstet Gynecol 2002; 186 : 675–83.
33.
Cerignoli F, Guo X, Cardinali B, et al. retSDR1, a short-chain retinol dehydrogenase/reductase, is retinoic acid-inducible and frequently deleted in human neuroblastoma cell lines. Cancer Res 2002; 62 : 1196–204.
34.
Martras S, Alvarez R, Martinez SE, et al. The specificity of alcohol dehydrogenase with cis-retinoids. Activity with 11-cis-retinol and localization in retina. Eur J Biochem 2004; 271 : 1660–70.
35.
Deltour L, Foglio MH, Duester G. Metabolic deficiencies in alcohol dehydrogenase Adh1, Adh3, and Adh4 null mutant mice. Overlapping roles of Adh1 and Adh4 in ethanol clearance and metabolism of retinol to retinoic acid. J Biol Chem 1999; 274 : 16796–801.
36.
Molotkov A, Fan X, Deltour L, et al. Stimulation of retinoic acid production and growth by ubiquitously expressed alcohol dehydrogenase Adh3. Proc Natl Acad Sci USA 2002; 99 : 5337–42.
37.
Lee SL, Wang MF, Lee AI, Yin SJ. The metabolic role of human ADH3 functioning as ethanol dehydrogenase. FEBS Lett 2003; 544 : 143–7.
38.
Molotkov A, Fan X, Duester G. Excessive vitamin A toxicity in mice genetically deficient in either alcohol dehydrogenase Adh1 or Adh3. Eur J Biochem 2002; 269 : 2607–12.
39.
Boleda MD, Saubi N, Farres J, Pares X. Physiological substrates for rat alcohol dehydrogenase classes: aldehydes of lipid peroxidation, omega-hydroxyfatty acids, and retinoids. Arch Biochem Biophys 1993; 307 : 85–90.
40.
Allali-Hassani A, Peralba JM, Martras S, et al. Retinoids, omega-hydroxyfatty acids and cytotoxic aldehydes as physiological substrates, and H2-receptor antagonists as pharmacological inhibitors, of human class IV alcohol dehydrogenase. FEBS Lett 1998; 426 : 362–6.
41.
Labrecque J, Bhat PV, Lacroix A. Purification and partial characterization of a rat kidney aldehyde dehydrogenase that oxidizes retinal to retinoic acid. Biochem Cell Biol 1993; 71 : 85–9.
42.
Posch KC, Burns RD, Napoli JL. Biosynthesis of all-trans-retinoic acid from retinal. Recognition of retinal bound to cellular retinol binding protein (type I) as substrate by a purified cytosolic dehydrogenase. J Biol Chem 1992; 267 : 19676–82.
43.
El Akawi Z, Napoli JL. Rat liver cytosolic retinal dehydrogenase: comparison of 13-cis-, 9-cis-, and all-trans-retinal as substrates and effects of cellular retinoid-binding proteins and retinoic acid on activity. Biochemistry 1994; 33 : 1938–43.
44.
Gagnon I, Duester G, Bhat PV. Enzymatic characterization of recombinant mouse retinal dehydrogenase type 1. Biochem Pharmacol 2003; 65 : 1685–90.
45.
Gagnon I, Duester G, Bhat PV. Kinetic analysis of mouse retinal dehydrogenase type-2 (RALDH2) for retinal substrates. Biochim Biophys Acta 2002; 1596 : 156–62.
46.
Grun F, Hirose Y, Kawauchi S, et al. Aldehyde dehydrogenase 6, a cytosolic retinaldehyde dehydrogenase prominently expressed in sensory neuroepithelia during development. J Biol Chem 2000; 275 : 41210–8.
47.
Lin M, Zhang M, Abraham M, et al. Mouse retinal dehydrogenase 4 (RALDH4), molecular cloning, cellular expression, and activity in 9-cis-retinoic acid biosynthesis in intact cells. J Biol Chem 2003; 278 : 9856–61.
48.
Lin M, Napoli JL. cDNA cloning and expression of a human aldehyde dehydrogenase (ALDH) active with 9-cis-retinal and identification of a rat ortholog, ALDH12. J Biol Chem 2000; 275 : 40106–12.
49.
Sonneveld E, van den Brink CE, van der Leede BM, et al. Human retinoic acid (RA) 4-hydroxylase (CYP26) is highly specific for all-trans-RA and can be induced through RA receptors in human breast and colon carcinoma cells. Cell Growth Differ 1998; 9 : 629–37.
50.
Marikar Y, Wang Z, Duell EA, et al. Retinoic acid receptors regulate expression of retinoic acid 4-hydroxylase that specifically inactivates all-trans retinoic acid in human keratinocyte HaCaT cells. J Invest Dermatol 1998; 111 : 434–9.
51.
Taimi M, Helvig C, Wisniewski J, et al. A novel human cytochrome P450, CYP26C1, involved in metabolism of 9-cis and all-trans isomers of retinoic acid. J Biol Chem 2004; 279 : 77–85.
52.
Ong DE. A novel retinol-binding protein from rat. Purification and partial characterization. J Biol Chem 1984; 259 : 1476–82.
53.
MacDonald PN, Ong DE. Binding specificities of cellular retinol-binding protein and cellular retinol-binding protein, type II. J Biol Chem 1987; 262 : 10550–6.
54.
Newcomer ME. Retinoid-binding proteins: structural determinants important for function. FASEB J 1995; 9 : 229–39.
55.
Inagami S, Ong DE. Purification and partial characterization of cellular retinol-binding protein, type two, from human small intestine. J Nutr 1992; 122 : 450–6.
56.
MacDonald PN, Ong DE. Binding specificities of cellular retinol-binding protein and cellular retinol-binding protein, type II. J Biol Chem 1987; 262 : 10550–6.
57.
Fiorella PD, Napoli JL. Expression of cellular retinoic acid binding protein (CRABP) in Escherichia coli. Characterization and evidence that holo-CRABP is a substrate in retinoic acid metabolism. J Biol Chem 1991; 266 : 16572–9.
58.
Fogh K, Voorhees JJ, Astrom A. Expression, purification, and binding properties of human cellular retinoic acid-binding protein type I and type II. Arch Biochem Biophys 1993; 300 : 751–5.
59.
Allenby G, Bocquel MT, Saunders M, et al. Retinoic acid receptors and retinoid X receptors: interactions with endogenous retinoic acids. Proc Natl Acad Sci USA 1993; 90 : 30–4.
60.
Kleywegt GJ, Bergfors T, Senn H, et al. Crystal structures of cellular retinoic acid binding proteins I and II in complex with all-trans-retinoic acid and a synthetic retinoid. Structure 1994; 2 : 1241–58.
61.
Fiorella PD, Giguere V, Napoli JL. Expression of cellular retinoic acid-binding protein (type II) in Escherichia coli. Characterization and comparison to cellular retinoic acid-binding protein (type I). J Biol Chem 1993; 268 : 21545–52.
62.
Norris AW, Cheng L, Giguere V, et al. Measurement of subnanomolar retinoic acid binding affinities for cellular retinoic acid binding proteins by fluorometric titration. Biochim Biophys Acta 1994; 1209 : 10–8.
63.
Petkovich M, Brand NJ, Krust A, Chambon P. A human retinoic acid receptor which belongs to the family of nuclear receptors. Nature 1987; 330 : 444–50.
64.
Levin AA, Sturzenbecker LJ, Kazmer S, et al. 9-cis retinoic acid stereoisomer binds and activates the nuclear receptor RXR alpha. Nature 1992; 355 : 359–61.