| |
| Med Sci (Paris). 2007 October; 23(10): 834–839. Published online 2007 October 15. doi: 10.1051/medsci/20072310834.Les domaines à doigts de zinc Vers la modification de la structure et de l’activité des génomes Sarah Dion,* Marie-Véronique Demattéi, and Sylvaine Renault* Laboratoire d’Étude des Parasites Génétiques, FRE CNRS 2969, Université François Rabelais, Parc Grandmont, 37270 Tours, France |
Les protéines possédant des motifs de liaison à l’ADN de type doigts de zinc (zinc finger, ZF) sont connues depuis environ 25 ans. Néanmoins, ce n’est que récemment que leur système de reconnaissance de l’ADN est utilisé non seulement pour cibler spécifiquement l’action activatrice ou inhibitrice d’un facteur de transcription, mais également pour réaliser le remplacement spécifique de gènes. L’utilisation de ces protéines zinc finger (ZFP) pour réaliser la correction de l’expression défectueuse de certains gènes et pour modifier un gène impliqué dans une maladie génétique est donc très prometteuse. |
Les ZF Cys 2His 2 : structure et interaction avec l’ADN Les protéines possédant des domaines ZF sont impliquées dans la régulation de la transcription de très nombreux gènes. Il existe différents types de domaines ZF dont celui de type Cys2His2, qui est présent dans environ 900 facteurs de transcription sur les 2 000 recensés dans le génome humain [
1]. Les domaines ZF sont également les plus abondants parmi les familles de protéines se liant à l’ADN autres que les facteurs de transcription chez les eucaryotes [1]. Le motif protéique ZF Cys2His2 est constitué d’environ 30 acides aminés avec une séquence consensus YF-X-C-X2-5-C-X3-YF-X5-L-X2-H-X3-5-H [
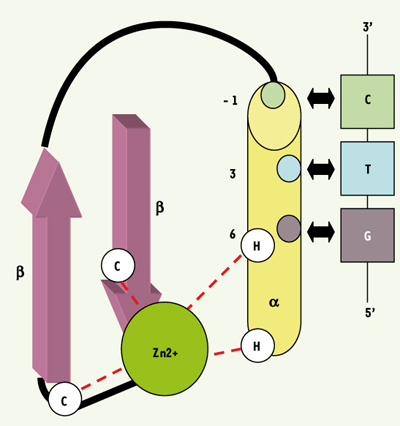
2]. Ces acides aminés sont organisés en une structure compacte ββα stabilisée par un ion zinc. L’atome de zinc est coordonné par les cystéines (des feuillets β) et les histidines (de l’hélice α) conservées (Figure 1). 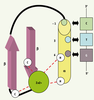 | Figure 1.
Représentation schématique d’un motif en doigts de zinc de type Cys2His2. Le motif est représenté avec les acides aminés jouant un rôle clé dans l’interaction avec le triplet de nucléotides. Le résidu en position +6 dans l’hélice α du motif ZF interagit avec la base en 5’ du triplet d’ADN reconnu, le résidu en +3 est en contact avec la base centrale du triplet, et le résidu en position -1 de l’hélice interagit avec la base en 3’ (d’après [
35]). |
L’étude cristallographique du complexe formé entre le facteur de transcription murin Zif268 (composé de 3 domaines ZF) et la séquence ADN de 9 paires de bases (pb) qu’il reconnaît, a montré que le mode de reconnaissance est relativement simple : un motif ZF reconnaît spécifiquement 3pb contiguës [
3]. Les acides aminés en positions -1, +3 et +6 de l’hélice α du motif ZF interagissent avec un brin de l’ADN (Figure 1). Des études complémentaires ont mis en évidence l’importance de l’acide aminé en position +2 de l’hélice α dans la spécificité de reconnaissance de l’ADN par le motif ZF, en permettant l’interaction avec le brin d’ADN opposé [
4]. Cette dernière caractéristique est à prendre en considération lorsque plusieurs motifs ZF sont associés les uns aux autres. En effet, l’assemblage de plusieurs motifs ZF, possédant des spécificités différentes de reconnaissance de l’ADN, permet de reconnaître une séquence nucléotidique plus longue. Par exemple, l’association de 6 motifs ZF permet de cibler une séquence de 18 pb, soit une séquence unique dans le génome humain. |
Les ZFP Cys 2His 2 : assemblage et sélection de nouveaux motifs ZF La nature modulaire des motifs ZF permet leur utilisation comme des blocs individuels permettant ainsi de créer des domaines à plusieurs motifs ZF capables de reconnaître une séquence ADN cible plus spécifique. Contrairement aux autres motifs de liaison à l’ADN, les motifs ZF ne requièrent pas de séquences cibles palindromiques et peuvent ainsi fonctionner sous forme de monomères, ce qui augmente le nombre de cibles potentielles qu’ils peuvent reconnaître. Cependant, toutes les combinaisons de motifs ZF permettant de reconnaître une séquence d’ADN déterminée n’existent pas naturellement. En se fondant sur les nombreuses études effectuées sur Zif268, plusieurs méthodes de sélection ont été développées pour obtenir des ZFP capables de reconnaître toute séquence de 9pb. Ces méthodes, qui utilisent la technique du phage display, ont permis d’obtenir des ZFP d’affinité plus élevée que celle des ZF des facteurs de trancription naturels [
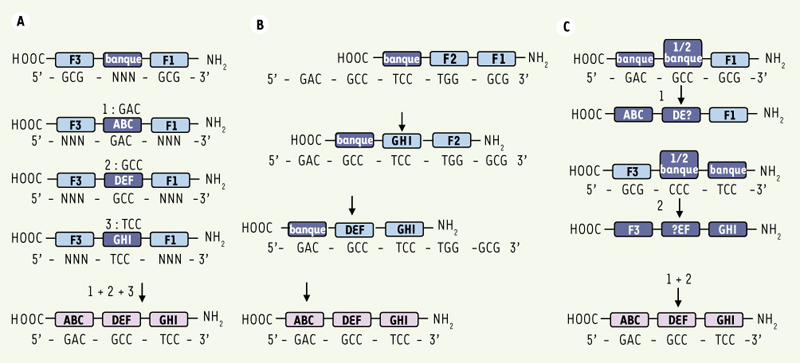
5]. Pour construire des ZFP composées de 3 motifs ZF, trois stratégies de sélection sont décrites dans la littérature (Figure 2).  | Figure 2.
Les stratégies pour produire des ZF avec une spécificité de reconnaissance de l’ADN cible. A. La sélection parallèle est basée sur un fonctionnement indépendant des motifs ZF individuels dans les ZFP, chaque doigt de zinc individuel dans une ZFP interagit avec son site de reconnaissance indépendamment du doigt de zinc voisin. Chacun des doigts de zinc est donc sélectionné indépendamment des autres. B. Dans la stratégie de sélection séquentielle, les doigts de zinc individuels sont sélectionnés dans le contexte de ses voisins, et par conséquent, les contraintes dues à la nature de la séquence ADN cible sont prises en compte. Chacun des doigts de zinc est donc sélectionné successivement en fonction des doigts de zinc adjacents. C. La stratégie bipartite utilise deux banques ZFP pré-créées avec dans chacune la moitié des trois motifs ZF partiellement randomisée au niveau des acides aminés clés qui font le contact avec l’ADN. La partie amino-terminale de la ZFP est inter-changée à partir d’une banque pendant que la partie carboxy-terminale est inter-changée à partir d’une autre banque. La sélection est faite en parallèle en utilisant les moitiés 5’ et 3’, respectivement, de la séquence cible. Les sélections faites à partir des banques individuelles sont associées et sélectionnées une nouvelle fois en utilisant la séquence cible entière dans le but d’obtenir une ZFP avec la spécificité désirée et une bonne affinité de reconnaissance (d’après [
25]). |
La sélection parallèle [
6,
7] consiste à sélectionner individuellement chacun des motifs ZF pour chaque triplet de la séquence d’ADN choisie, puis de lier les 3 motifs ZF obtenus indépendamment par un linker canonique (Figure 2A). Actuellement, des motifs ZF spécifiques capables de reconnaître les deux tiers du code génétique ont été décrits. Ces motifs reconnaissent tous les triplets ADN de type GNN ou ANN. Cependant, même si cette première méthode permet de cibler une séquence d’ADN choisie, même si elle est rapide et accessible à tous les laboratoires, elle ne prend pas en compte les interactions possibles entre les motifs ZF adjacents, obligeant à mettre au point une autre méthode, la sélection séquentielle [
8]. Cette deuxième méthode a été développée car il a été montré que l’asparagine en position 2 du doigt de zinc 2 de Zif268 interagit avec le site de liaison du doigt de zinc 1 conduisant à ce que ce site soit de type GNN, TNN ou CNN [
9]. Cette contrainte doit donc être prise en compte pour une affinité optimale. La sélection séquentielle consiste dans un premier temps à assembler un motif ZF variable à 2 motifs ZF fixés du domaine Zif268. Le ZF variable est modifié en testant d’autres motifs ZF d’une banque jusqu’à trouver celui qui a la meilleure affinité. Dans un deuxième temps, sur le même principe, le second motif ZF, puis le troisième motif ZF, sont ainsi sélectionnés. Les 3 motifs ZF ainsi obtenus ont une affinité très élevée pour la séquence d’ADN choisie, mais cette méthode est longue et difficile d’accès pour de nombreux laboratoires car elle nécessite l’utilisation de plusieurs banques de motifs ZF (Figure 2B). Enfin, il existe une troisième méthode de sélection combinant les avantages des deux premières, la sélection bipartite [4–
10]. Cette stratégie utilise deux banques, dont chacune contient un doigt de zinc et demi de Zif268. Cette méthode consiste, pour une séquence de 9pb, en une première sélection en parallèle sur 5pb du côté 5’ et sur 5pb du côté 3’. La deuxième sélection consiste en l’addition des deux motifs et demi ZF ainsi obtenus qui sont alors optimisés sur la séquence finale (Figure 2C). Les stratégies utilisant la technique de phage display nécessitent beaucoup de cycles d’enrichissement et d’amplification, ce qui a suscité le développement de systèmes alternatifs plus rapides de sélection en levure [
11], en bactéries [
12] et, depuis peu, en cellules de mammifères, permettant ainsi de prendre en compte l’influence du contexte cellulaire [
13]. En augmentant le nombre de motifs ZF dans les ZFP, le nombre de bases ciblées peut être plus important. En théorie, il est possible de cibler des sites potentiellement uniques dans le génome humain en utilisant six motifs ZF ou plus [
14]. Des ZFP à six motifs ZF (6ZFP) ont été produites en utilisant soit un linker canonique de 5 acides aminés, TGEKP, entre chaque ZF [14–
16], soit en associant deux fois trois motifs ZF avec un plus long linker [
17]. L’augmentation de l’affinité et de la spécificité que confère l’utilisation d’un plus long linker a été attribuée à l’augmentation de la flexibilité dans le complexe 6ZFP/ADN. En effet, de plus petits linkers pourraient générer une perte d’entropie entraînant une perte de l’affinité [
18]. La construction d’une 6ZFP hautement spécifique a été obtenue par l’ajout de 3 groupes de deux ZF associés par un linker canonique modifié [
19]. Pour que les ZFP conçues in vitro puissent agir de manière efficace, il faut pour les concevoir cibler une zone ouverte du génome. |
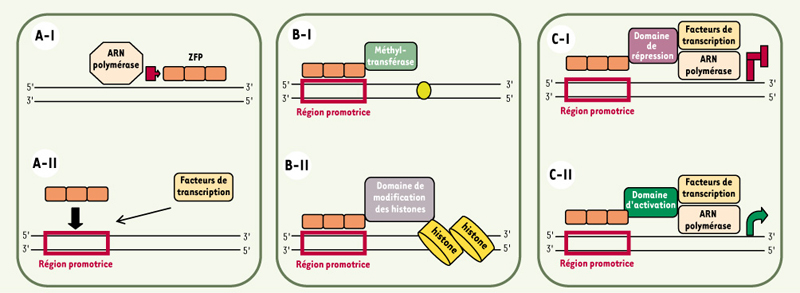
Les ZFP Cys 2His 2 : leurs différentes applications Les domaines de reconnaissance à l’ADN de type ZF peuvent être fusionnés ou non à un domaine effecteur afin de modifier l’expression de gènes ou le génome lui-même. Modification de l’expression génique Les domaines de type ZF non fusionnés à un domaine effecteur peuvent agir de deux manières. Premièrement, la liaison des ZF sur leur cible peut agir en tant que barrière. Ainsi l’ARN polymérase (Figure 3A-I) a été bloquée lors de l’utilisation d’une ZFP de trois motifs ZF bloquant l’expression de la protéine oncogène p190 BCR-ABL [
20]. Deuxièmement, les domaines de type ZF seuls peuvent également agir comme compétiteurs vis-à-vis des protéines de liaison à l’ADN comme les facteurs de transcription. Une protéine contenant 6 motifs ZF, 6F6KOX, a permis l’inhibition de la réplication de HSV1 (herpes simplex virus de type 1) en bloquant le site de liaison de l’activateur viral VP16 [
21] (Figure 3A-II). Cependant l’action de ZF non fusionnés à un domaine effecteur est souvent transitoire du fait du temps de demi-vie très court de ces protéines. 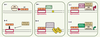 | Figure 3.
Les différentes applications des ZF. Le mécanisme d’action des facteurs de transcription artificiels dépend du site ADN ciblé et du domaine effecteur (s’il y en a un) fusionné aux ZF (un rectangle orange représente un ZF). En l’absence de domaine effecteur (A), les ZF peuvent être utilisés comme des barrières naturelles vis-à-vis du complexe transcriptionnel (A-I) ou comme des compétiteurs vis-à-vis d’autres protéines (les activateurs ou répresseurs naturels), qui se fixent au niveau des régions promotrices (rectangle rouge) (A-II). Les ZF peuvent également être utilisés pour créer des enzymes chimériques (B) capables de modifier directement l’ADN, par exemple par méthylation de l’ADN lui-même (B-I) ou des histones qui sont des facteurs de remodelage de la chromatine pouvant agir soit comme activateurs, soit comme répresseurs (B-II). Les ZF conçus in vitro peuvent être utilisés comme des facteurs de transcription ciblant les régions promotrices des gènes d’intérêt (C). Dans ces cas, les nouveaux facteurs de transcription interagissant avec d’autres éléments de la machinerie transcriptionnelle et selon le domaine effecteur fusionné auront une activité d’inhibiteur (C-I) ou d’activateur (C-II) sur la transcription (d’après [ 21]). |
La fusion de motifs ZF à des domaines effecteurs interagissant directement avec l’ADN permet potentiellement de cibler l’action d’un grand nombre de domaines de modification de l’ADN à un locus désiré. Les domaines ADN méthyltransférases, des domaines répresseurs et activateurs de transcription, ont été utilisés avec succès pour modifier l’expression des gènes adjacents au site de fixation des ZFP. Les ADN méthyltransférases permettent la méthylation de l’ADN au niveau du carbone 5 des cytosines de l’ADN. Cette méthylation joue un rôle important dans l’établissement des profils d’expression génique corrects chez les vertébrés, souvent par répression de la transcription. Dans de nombreux cancers et autres maladies, des profils aberrants de méthylation de l’ADN ont été observés. La capacité à cibler la méthylation des cytosines, en particulier dans les régions promotrices, serait donc cruciale pour rétablir la régulation du gène impliqué (Figure 3B-I). Dans cet objectif, la capacité de méthylation spécifique des dinucléotides GC ou CG par les ADN méthyl-transférase M.CviPI et M.SssI, lorsqu’elles sont fusionnées à Zif268, a été augmentée d’un facteur 20 chez la levure [
22]. Les méthylations des histones peuvent engendrer des changements structuraux de la chromatine et par conséquent des profils d’expression génique (Figure 3B-II). La répression efficace de l’expression génique en fusionnant des ZF synthétiques ciblant le promoteur VEGF-A à un domaine minimal d’une histone méthyltransférase a été démontrée [
23]. Dans ces études, la méthylation de la lysine 9 de l’histone H3 a engendré une diminution de l’expression du VEGF-A. Dans de nombreuses applications, l’ajout d’un domaine d’activation ou d’inhibition de la transcription peut ainsi créer de nouveaux facteurs de transcription pouvant réguler l’expression d’un gène d’intérêt (Figure 3C). Les domaines répresseurs comme kruppel associated box (KRAB) [
24], ERF repressor domain (ERD) [25] et, parmi les domaines activateurs, le domaine VP16 de HSV1 [21], ont permis, lorsqu’ils sont fusionnés avec un ZF, de cibler des régions promotrices et de modifier l’expression des gènes adjacents [25–
27]. La capacité à pouvoir cibler de manière spécifique des séquences ADN fait des motifs ZF d’excellents outils pour l’étude fonctionnelle des gènes [27]. Le domaine VP16 fusionné à un ZF ciblant le gène AP3 d’Arabidopsis thaliana a permis l’expression stable de ce gène pendant plusieurs générations [27]. L’expression cellulaire d’une ZFP appropriée permet de cibler les 2 allèles du gène en même temps contrairement aux autres systèmes existants, anti-sens, ribozymes ou ARN interférence, qui doivent cibler toutes les copies d’ARNm ou tous les produits protéiques. Sûrement le champ d’application le plus excitant est l’utilisation des ZFP en thérapie génique. Selon le type de ZFP utilisé (répresseur, activateur), le traitement par un transgène ZFP offre de nombreux avantages par rapport aux approches thérapeutiques conventionnelles. Par exemple, les répresseurs ZFP (Figure 3C-I) sont plus sélectifs dans la répression génique et ont un risque moindre de provoquer une réponse cellulaire non spécifique, à l’inverse des approches de ciblages d’ARN ou de produits protéiques. Réciproquement, les activateurs ZFP (Figure 3C-II) peuvent être utilisés pour augmenter l’expression endogène d’un gène ayant un bénéfice thérapeutique. Cette dernière approche peut être le meilleur moyen pour exprimer une protéine thérapeutique dans les cas où les ADNc correspondants ont une taille trop importante pour utiliser les vecteurs de transgenèse classiques. De plus, cette approche permet l’expression de tous les variants d’épissage du gène thérapeutique nécessaires pour la fonction de nombreux gènes. Un activateur ZFP du VEGF-A (vascular endothelial growth factor A) est actuellement en cours d’étude pour le développement d’un traitement contre les maladies vasculaires [
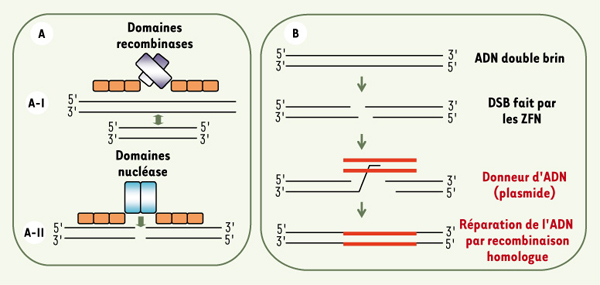
28]. Ces études ont démontré que cette ZFP active l’expression de toutes les isoformes majeurs du VEGF, induisant la croissance des vaisseaux sanguins et apportant ainsi un bénéfice thérapeutique dans les modèles animaux de maladie artérielle périphérique. Modification du génome Deux stratégies de modifications du génome par ZFP sont actuellement en cours de développement : l’une consiste à intégrer un gène d’intérêt à un site cible défini afin d’éviter les mutations insertionnelles qui peuvent être délétères ; l’autre consiste à induire une coupure dans le gène à modifier et à utiliser les capacités de la cellule à réparer les coupures double- brin (DSB) en utilisant comme matrice une version corrigée du gène d’intérêt. La première stratégie utilise des domaines catalytiques de type intégrases et résolvases (Figure 4A-I). Dans le but de créer des vecteurs de transfert de gène à partir de rétrovirus ayant des intégrations contrôlées au niveau de sites chromosomiques définis, l’équipe de Chow [
29] a fusionné à des intégrases tronquées du VIH (virus de l’immunodéficience humaine) la protéine synthétique E2C comportant six doigts de zinc. Les auteurs ont ainsi pu montrer un point chaud d’intégration situé à 20pb du site reconnu par E2C.  | Figure 4.
Modification du génome par des ZFP. A. Des ZF fusionnées avec la nucléase de FokI sont capables de faire une coupure double brin et permettent le remplacement de gènes dans 10 % des cellules. A-I. Des ZF-recombinases sont capables de médier des événements de recombinaison/intégration. A-II. Dans ce mode d’action, le site de liaison des ZF peut être choisi dans ou en dehors de la région promotrice. B. Principe de la recombinaison homologue favorisée par une cassure double brin (DSB) crée par les ZF-nucléases (ZFN) (d’après [
33]). |
Des résolvases ont également été utilisées (Figure 4A-I). Notamment dans le travail sur la recombinaison site-spécifique par la recombinase chimère composée du domaine catalytique d’un mutant hyperactif de la résolvase Tn3 fusionné aux domaines de reconnaissance de l’ADN de Zif268. Par ces travaux, les auteurs ont pu créer des chimères actives en bactéries catalysant des recombinaisons spécifiques sur des séquences cibles [
30]. Cependant, pour de nombreuses maladies génétiques, une deuxième stratégie a été mise en place afin d’apporter une version corrigée du gène muté (Figure 4A-II). Cette stratégie d’édition du génome fondée sur l’activité de nucléases semble très prometteuse notamment celle qui utilise la nucléase Fok1 [
31] (Figure 4A-II). À l’inverse des approches fondées sur l’intégration d’un ADNc, l’édition du génome offre la possibilité de réellement réparer le gène, les cellules traitées avec succès ne sont pas modifiées, excepté pour la correction du gène muté. L’avantage de cette approche est qu’elle assure une régulation appropriée du gène corrigé. L’activité des nucléases-ZFP a été démontrée dans les cellules de métazoaires : celles-ci produisent des cassures double-brin au site cible chromosomique stimulant le système de réparation par recombinaison homologue (Figure 4B). Ce système est fondé sur des études précédentes sur l’endonucléase I-SceI [
32]. Donc, en traitant les cellules avec des vecteurs exprimant une nucléase-ZFP et en apportant un ADN donneur correspondant au segment du gène muté, il sera possible d’obtenir une édition très efficace du génome dans le but d’introduire des changements génétiques précis (Figure 4). Cette approche a été récemment validée dans des cellules d’insecte [31], humaines [33] et de plantes [
34]. |
Le concept de la technologie ZF, permettant de cibler une séquence précise d’un génome en étant associée à des domaines protéiques effecteurs, en fait un outil puissant de régulation de l’expression et de modification des génomes procaryotes et eucaryotes. De nombreuses applications biotechnologiques et thérapeutiques sont décrites depuis une dizaine d’années, principalement relayées par l’entreprise Sangamo Biosciences. Les axes d’application de cette technologie concernent essentiellement le développement de facteurs de transcription synthétiques et la correction de gènes. En effet, la production de facteurs de transcription synthétiques permet aussi bien de faire produire en grande quantité une protéine mal exprimée chez l’homme qu’une protéine d’intérêt agronomique chez une plante. Ainsi un essai clinique de phase 2 est actuellement en cours pour l’utilisation d’une ZFP capable d’inhiber l’expression du VEGF-A impliquée dans les diabètes neuropathiques. L’autre domaine d’application, la correction thérapeutique de gènes, a été validée sur le gène IL-2Rγ impliqué un syndrome sévère d’immunodéficience associé à l’X. De nombreuses autres gènes, comme le gène de la β globine, muté dans la β-thalassémie, sont également en cours de modification. Il n’y a guère de doutes que nous verrons se multiplier dans les mois et années à venir les essais de thérapie sur de nombreuses maladies, monogéniques dans un premier temps, pour ensuite se développer certainement sur des maladies multigéniques. |
1. Tupler R, Perini G, Green MR. Expressing the human genome. Nature 2001; 409 : 832–3. 2. Miller J, McLachlan AD, Klug A. Repetitive zinc-binding domains in the protein transcription factor IIIA from Xenopus oocytes. EMBO J 1985; 4 : 1609–14. 3. Pavletich NP, Pabo CO. Zinc finger-DNA recognition: crystal structure of a Zif268-DNA complex at 2,1 Å. Science 1991; 252 : 809–17. 4. Isalan M, Klug A, Choo Y. Comprehensive DNA recognition through concerted interactions from adjacent zinc fingers. Biochemistry 1998; 37 : 12026–33. 5. Mc Namara RA, Ford KG. A novel four zinc-finger protein targeted against p190BcrAbl fusion oncogene cDNA: utilisation of zinc-finger recognition codes. Nucleic Acids Res 2000; 24 : 4865–72. 6. Desjarlais JR, Berg JM. Use of a zinc-finger consensus sequence framework and specificity rules to design specific DNA binding proteins. Proc Natl Acad Sci USA 1993; 90 : 2256–60. 7. Choo Y, Klug A. Toward a code for interactions of zinc fingers with DNA: selection of randomized fingers displayed on phage. Proc Natl Acad Sci USA 1994; 91 : 11163–7. 8. Greisman HA, Pabo CO. A general strategy for selecting high-affinity zinc finger proteins for diverse DNA target sites. Science 1997; 275 : 657–61. 9. Dreier B, Fuller RP, Segal DJ, et al. Development of zinc finger domains for recognition of the 5’-CNN-3’ family of DNA sequences and their use in the construction of artificial transcription factors. J Biol Chem 2005; 280 : 35588–97. 10. Isalan M, Klug A, Choo Y. A rapid, generally applicable method to engineer zinc fingers illustrated by targeting the HIV-1 promoter. Nat Biotechnol 2001; 19 : 656–60. 11. Bartsevich VV, Juliano RL. Regulation of the MDR1 gene by transcriptional repressors selected using peptide combinatorial libraries. Mol Pharmacol 2000; 58 : 1–10. 12. Hurt JA, Thibodeau SA, Hirsh AS, et al. Highly specific zinc finger proteins obtained by directed domain shuffling and cell based selection. Proc Natl Acad Sci USA 2003; 100 : 12271–6. 13. Bae KH, Do Kwon Y, Shin HC, et al. Human zinc fingers as building blocks in the construction of artificial transcription factors. Nat Biotechnol 2003; 21 : 275–80. 14. Liu PQ, Segal DJ, Ghiara JB, Barbas CF 3rd. Design of polydactyl zinc-finger proteins for unique addressing within complex genomes. Proc Natl Acad Sci USA 1997; 94 : 5525–30. 15. Segal DJ, Barbas CF 3rd. Custom DNA-binding proteins come of age: polydactyl zinc-finger proteins. Curr Opin Biotechnol 2001; 12 : 632–7 16. Beerli RR, Barbas CF 3rd. Engineering polydactyl zinc-finger transcription factors. Nat Biotechnol 2002; 20 : 135–41. 17. Kim JS, Pabo CO. Getting a handhold on DNA: design of poly-zinc finger proteins with femtomolar dissociation constant. Proc Natl Acad Sci USA 1998; 95 : 2812–7. 18. Peisach E, Pabo CO. Constraints for zinc finger linker design as inferred from X-ray crystal structure of tandem Zif268-DNA complexes. J Mol Biol 2003; 330 : 1–7. 19. Moore M, Klug A, Choo Y. Improved DNA binding specificity from polyzinc finger peptides by using strings of two finger units. Proc Natl Acad Sci USA 2001; 98 : 1437–41. 20. Choo Y, Sanchez-Garcia I, Klug A. In vivo repression by a site-specific DNA-binding protein designed against an oncogenic sequence. Nature 1994; 372 : 642–5. 21. Papworth M, Moore M, Isalan M, et al. Inhibition of herpes simplex virus 1 gene expression by designed zinc-finger transcription factors. Proc Natl Acad Sci USA 2003; 100 : 1621–6. 22. Carvin CD, Parr RD, Kladde MP. Site-selective in vivo targeting of cytosine-5 DNA methylation by zinc-finger proteins. Nucleic Acids Res 2003; 31 : 6493–501. 23. Snowden AW, Gregory PD, Case CC, Pabo CO. Gene-specific targeting of H3K9 methylation is sufficient for initiating repression in vivo. Curr Biol 2002; 12 : 2159–66. 24. Thiesen HJ, Bellefroid E, Revelant O, Martial JA. Conserved KRAB protein domain identified upstream from the zinc finger region of Kox 8. Nucleic Acids Res 1991; 19 : 3996. 25. Beerli RR, Segal DJ, Dreier B, Barbas III CF. Toward controlling gene expression at will : specific regulation of the erbB-2/HER-2 promoter by using polydactyl zinc finger proteins constructed from modular building blocks. Proc Natl Acad Sci USA 1998; 95 : 14628–33. 26. Bartsevich VV, Miller JC, Case CC, Pabo CO. Engineered zinc finger proteins for controlling stem cell fate. Stem Cells 2003; 21 : 632–7. 27. Guan X, Stege J, Kim M, et al. Heritable endogenous gene regulation in plants with designed polydactyl zinc finger transcription factors. Proc Natl Acad Sci USA 2002; 99 : 13296–301. 28. Rebar EJ, Huang Y, Hickey R, et al. Induction of angiogenesis in a mouse model using engineered transcription factors. Nat Med 2002; 8 : 1427–32. 29. Tan W, Zhu K, Segal DJ, et al. Fusion proteins consisting of human immunodeficiency virus type 1 integrase and the designed polydactyl zinc finger protein EC2 direct integration of viral DNA into specific sites. J Virol 2004; 78 : 1301–13. 30. Akopian A, He J, Boocock MR, Stark WM. Chimeric recombinases with designed DNA sequence recognition. Proc Natl Acad Sci USA 2003; 100 : 8688–91. 31. Bibikova M, Golic M, Golic KG, Caroll D. Targeted chromosomal cleavage and mutagenesis in drosophila using Zinc Finger nucleases. Genetics 2002; 161 : 1169–75. 32. Puchta H, Dujon B, Hohn B. Homologous recombination in plant cells is enhanced by in vivo induction of double-strand breaks into DNA by a site-specific endonuclease. Nucleic Acids Res 1993; 21 : 5034–40. 33. Urnov FD, Miller JC, Lee YL, et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature 2005; 435 : 646–51. 34. Wright DA, Townsend JA, Winfrey H, et al. High-frequency homologous recombination in plants mediated by zinc-finger nucleases. Plant J 2005; 44 : 693–705. 35. Uil TG, Haisma HJ, Rots MG. Therapeutic modulation of endogenous gene function by agents with designed DNA-sequence specificities. Nucleic Acids Res 2003; 31 : 6064–78. |