| |
| Med Sci (Paris). 2007 April; 23(4): 349–350. Published online 2007 April 15. doi: 10.1051/medsci/2007234349.L’horloge circadienne centrale et les horloges périphériques Décentralisation et contrôle hiérarchique Benoît Kornmann* Département de Biologie Moléculaire, Université de Genève, 30, quai Ernest-Ansermet, 1211 Genève 4, Suisse MeSH keywords: Facteurs de transcription ARNTL, Animaux, Facteurs de transcription à motif basique hélice-boucle-hélice, Horloges biologiques, Thermorégulation du corps, Protéines du cycle cellulaire, Rythme circadien, Protéines de liaison à l'ADN, Doxycycline, Analyse de profil d'expression de gènes, Humains, Hypothalamus, Lumière, Foie, Souris, Souris transgéniques, Modèles biologiques, Protéines nucléaires, Membre-1 du groupe D de la sous-famille-1 de récepteurs nucléaires, Séquençage par oligonucléotides en batterie, Protéines circadiennes Period, Protéomique, Récepteurs cytoplasmiques et nucléaires, Protéines de fusion recombinantes, Noyau suprachiasmatique, Facteurs de transcription, Transcription génétique |
La gestion d’une structure d’une certaine taille s’accompagne souvent de lourdeurs administratives. Maintenir fonctionnalité et réactivité nécessite alors de déléguer des pouvoirs à des sous-structures plus petites. C’est ce phénomène qui pousse les grandes organisations étatiques à la décentralisation des compétences vers les collectivités locales. En biologie, où l’évolution tend vers la complexification des organismes, la gestion de la physiologie cellulaire relève bien souvent de la compétence autonome de la cellule elle-même, l’organisme gardant toutefois un contrôle systémique (hormones ou influx nerveux jouant le rôle de directives, courriels et téléfax…) pour assurer la cohérence de l’ensemble. Il en est de même pour l’organisation hiérarchique de l’horloge circadienne. Cette horloge, qui nous permet d’anticiper les variations du cycle jour-nuit et d’adapter en conséquence de nombreux aspects de la physiologie (cycles veille-sommeil, prise alimentaire, température corporelle, etc.) relève ainsi de la compétence de la cellule tout en étant soumise à un contrôle systémique [
1]. En effet, chaque cellule de l’organisme possède un mécanisme d’horlogerie moléculaire capable d’engendrer une oscillation d’environ 24 heures. Mais, afin de synchroniser les changements physiologiques circadiens aux changements environnementaux dus au cycle jour-nuit, un mécanisme de mise à jour opère quotidiennement et adapte la phase des horloges cellulaires à l’heure du jour. Comme on pouvait s’y attendre, c’est la lumière qui est utilisée comme référence de temps. Le signal lumineux, capté par la rétine, est intégré au niveau du noyau suprachiasmatique (NSC), à la base de l’hypothalamus. L’horloge du NSC, mise à jour par le signal rétinien, envoie à son tour des signaux systémiques qui relaient l’information lumineuse auprès des horloges périphériques. Elle joue en cela un rôle d’« horloge centrale » [1]. |
La hiérarchie des mécanismes Cela étant établi, il devient nécessaire de distinguer, dans la cellule périphérique, deux types de mécanismes circadiens : ceux qui sont dus aux signaux systémiques obéissant au contrôle direct de l’administration centrale, et ceux dont la gestion est décentralisée vers les horloges locales. C’est ce distinguo que nous avons voulu faire [
2]. La stratégie est simple : si nous détruisons le mécanisme moléculaire d’horlogerie dans la périphérie uniquement, nous devrions observer l’arrêt des processus circadiens décentralisés, alors que ceux qui sont contrôlés par les signaux systémiques devraient continuer leur cycle. Nous nous sommes spécifiquement intéressés aux horloges périphériques du foie. Afin d’en arrêter l’horloge, nous avons créé des souris exprimant un transgène codant pour Rev-erbα, spécifiquement dans les hépatocytes et sous le contrôle de la doxycycline (Dox) [
3]. Rev-erbα est un répresseur naturel de la transcription du gène Bmal1, qui est lui-même une composante essentielle de l’oscillateur moléculaire [
4,
5]. Lorsque notre transgène Rev-erbα est exprimé, le niveau de Bmal1 chute dans les hépatocytes au point que l’oscillateur cesse de fonctionner. En revanche, l’ajout de Dox à la nourriture des souris inactive le transgène et permet aux oscillateurs hépatiques de fonctionner. |
Le transcriptome hépatique : une mine à explorer Afin d’estimer les dégâts occasionnés au système circadien par l’expression de notre transgène, nous avons étudié les variations circadiennes du transcriptome hépatique. En effet, plusieurs études montrent que l’abondance de 1-5 % des transcrits varie avec l’heure du jour [
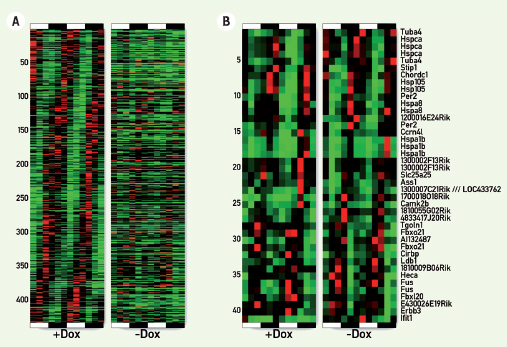
6]. Voilà donc une mine de processus circadiens parmi lesquels nous pouvons chercher ceux dont le contrôle est systémique et ceux dont le contrôle est local. Le résultat est sans appel : une très large majorité des quelque 350 transcrits dont l’accumulation varie au cours de la journée voient leur profil d’accumulation circadien compromis par l’expression du transgène et l’arrêt concomitant de l’oscillateur (Figure 1A), indiquant que la majorité de ces processus sont gérés de manière décentralisée.  | Figure 1.
Analyse transcriptomique des messagers hépatiques circadiens lorsque l’horloge du foie est fonctionnelle (+Dox) ou arrêtée (-Dox). A. Plus de 350 transcrits ont une accumulation circadienne lorsque l’horloge hépatique est fonctionnelle. Une large majorité de ceux-ci est considérablement affectée par l’arrêt de l’oscillateur. Chaque ligne correspond à une sonde sur le microarray (certains transcrits sont représentés par plusieurs sondes) et chaque colonne, un temps sur une période de 48 h. Le rouge correspond à un maximum d’expression et le vert à un minimum. B. Un sous-groupe des transcrits présentés en A n’est pas affecté outre mesure par l’arrêt de l’oscillateur local. Ceux-ci sont donc potentiellement régulés par des signaux systémiques. |
Toutefois, nous avons détecté un petit sous-ensemble d’une trentaine de transcrits dont le profil circadien n’est en rien affecté par l’absence d’horloge locale (Figure 1B). Ce sous-ensemble de transcrits potentiellement contrôlés par des signaux systémiques est d’autant plus intéressant qu’il contient le transcrit de mPer2. Cela est tout à fait inattendu car le gène mPer2 est non seulement un rouage essentiel du mécanisme moléculaire de l’horloge [
7,
8], mais aussi un gène dont la transcription circadienne ne dépend pas strictement de signaux systémiques puisqu’elle peut être reconstituée in vitro dans des cultures de cellules ou des explants d’organes [
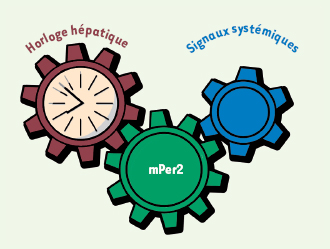
9]. De plus il a été suggéré que l’expression de mPer2 - au moins dans le NSC - nécessitait Bmal1 [4, 9]. Comment concilier ces observations ? Nous suggérons que la régulation de mPer2 est soumise à deux mécanismes distincts (Figure 2). L’horloge locale, par le biais de Bmal1, active périodiquement mPer2, créant un cycle d’accumulation de mPer2 essentiel au mécanisme de l’horloge. En parallèle, des signaux systémiques sont intégrés sur le promoteur de mPer2 et conduisent à son expression circadienne, également en l’absence d’horloge locale. Au final, la phase de l’expression de mPer2, contrôlée par les signaux systémiques, dictera la phase de l’oscillateur local.  | Figure 2.
Le gène mPer2 est tout à la fois un rouage essentiel de l’horloge cellulaire locale et un gène contrôlé par des signaux systémiques. Ce double rôle pourrait permettre à la phase de l’horloge hépatique de rester cohérente avec la phase de l’organisme, et à l’horloge centrale de garder le contrôle sur les horloges périphériques. |
Et voilà comment l’horloge centrale pourrait décentraliser ses compétences vers la périphérie tout en gardant les horloges subordonnées sous contrôle. Quelle pourrait être la nature de ces signaux systémiques ? Le sous-ensemble de transcrits auquel appartient mPer2 nous apporte un début de réponse. En effet, dans ce groupe, outre mPer2, se trouvent cinq transcrits de protéines de choc thermique (HSP, heat-shock proteins) ainsi qu’un transcrit induit par le froid (Cirbp). Le pic d’expression des HSP coïncide avec le maximum du cycle de température corporelle des souris alors que le pic d’expression de Cirbp coïncide avec son minimum. Il est donc tentant de penser que le cycle de température corporelle conduit le cycle d’accumulation de ces transcrits ainsi que celui de mPer2. De fait, nous avons montré, dans des explants de tissus placés en culture, que des chocs thermiques induisent l’expression de mPer2, rendant le modèle d’autant plus attractif. |
1. Schibler U. The daily rhythms of genes, cells and organs. Biological clocks and circadian timing in cells. EMBO Rep 2005; 6 (n° special) : S9–13. 2. Kornmann B, Schaad O, Bujard H, et al. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol 2007; 5 : e34. 3. Kistner A, Gossen M, Zimmermann F, et al. Doxycycline-mediated quantitative and tissue-specific control of gene expression in transgenic mice. Proc Natl Acad Sci USA 1996; 93 : 10933–8. 4. Bunger MK, Wilsbacher LD, Moran SM, et al. Mop3 is an essential component of the master circadian pacemaker in mammals. Cell 2000; 103 : 1009–17. 5. Preitner N, Damiola F, Lopez-Molina L, et al. The orphan nuclear receptor REV-ERBalpha controls circadian transcription within the positive limb of the mammalian circadian oscillator. Cell 2002; 110 : 251–60. 6. Duffield GE. DNA microarray analyses of circadian timing: the genomic basis of biological time. J Neuroendocrinol 2003; 15 : 991–1002. 7. Bae K, Jin X, Maywood ES, et al. Differential functions of mPer1, mPer2, and mPer3 in the SCN circadian clock. Neuron 2001; 30 : 525–36. 8. Zheng B, Larkin DW, Albrecht U, et al. The mPer2 gene encodes a functional component of the mammalian circadian clock. Nature 1999; 400 : 169–73. 9. Yoo SH, Ko CH, Lowrey PL, et al. A noncanonical E-box enhancer drives mouse Period2 circadian oscillations in vivo. Proc Natl Acad Sci USA 2005; 102 : 2608–13. |