La maturation de l’affinité des anticorps au cours de la réponse immunitaire correspond à la sélection, dans la population des lymphocytes B à mémoire, des cellules présentant la meilleure affinité contre l’agent infectieux, ce qui permettra une réponse plus rapide et plus efficace lors d’une nouvelle rencontre avec ce pathogène. Ce processus bien connu fonde la démarche vaccinale.
La modification du répertoire des gènes des immunoglobulines au cours de l’activation des lymphocytes B représente cependant, en termes moléculaires, un mécanisme biologique unique chez les eucaryotes supérieurs. Elle consiste en effet en une mutagenèse localisée, induite par l’agent infectieux, aboutissant à l’évolution accélérée des acteurs précisément mobilisés pour son élimination.
Cette capacité de modification des gènes des immunonoglobulines est restée pendant près de trente ans sans explication moléculaire, jusqu’à la découverte, par le groupe de Tasuku Honjo, de la protéine AID (activation-induced cytidine deaminase). L’inactivation du gène codant pour cette enzyme abolit tous les processus de modification des gènes des immunoglobulines postérieurs à leur assemblage par recombinaison, c’est-à-dire non seulement les mutations somatiques, mais également la commutation isotypique et la conversion génique [ 1, 2]. L’AID, qui est une cytidine désaminase, déclenche ces processus par une désamination des cytosines en uraciles au locus des immunoglobulines, une modification potentiellement mutagène par elle-même.
Mais l’action de l’AID ne suffit pas à expliquer à elle seule la mutagenèse des gènes des immunoglobulines [ 3]. La première raison en est que les mutations observées ne se réduisent pas aux seuls sites de désamination (les bases C), mais affectent en proportion égale les bases A/T et les bases G/C. La deuxième est que l’uracile est une lésion assez banale dans l’ADN, produite soit par désamination (métabolique) des cytosines (quelques centaines par jour dans le génome humain), soit par incorporation incorrecte d’uraciles (à la place de la thymidine) lors de la réplication (quelques milliers par cycle de réplication). La cellule eucaryote possède de nombreux systèmes de réparation dédiés à la reconnaissance et à l’élimination de cette base anormale, en premier lieu desquels les uraciles glycosylases (UNG, plus particulièrement impliquée lors de la réplication et SMUG, plutôt dirigée vers l’élimination des produits de désamination).
Dans l’hypermutation, la problématique est naturellement différente, puisqu’il s’agit bien de créer des mutations dans l’ADN après reconnaissance de l’uracile, et non de le réparer sans laisser de traces. Pour muter les gènes des immunoglobulines, il va falloir en premier lieu détourner ces systèmes de réparation de leur rôle de gardiens de l’information génétique. La mutagenèse induite par l’AID ne va donc pas fonctionner par saturation des capacités de réparation de la cellule (un processus sans doute trop dangereux, car pas assez précis), mais par modification des circuits habituellement mobilisés.
Trois facteurs principaux ont été décrits comme participant au processus d’hypermutation : l’uracile glycosylase (UNG), le mismatch repair (MSH2-MSH6) et l’ADN polymérase eta [ 4– 7]. L’uracile glycosylase enlève habituellement la base anormale, puis la chaîne d’ADN est incisée et la base manquante resynthétisée. Le mismatch repair est lui en charge des erreurs introduites lors de la réplication, et est composé d’un complexe reconnaissant les mésappariements (MSH2-MSH6), et d’un complexe effecteur (PMS2-MLH1) qui va diriger la resynthèse d’une portion d’ADN autour du mésappariement. Enfin, l’ADN polymérase eta appartient à la famille des polymérases translésionnelles (qui inclut au moins quatre autres membres dont pol zeta et Rev1), polymérases qui permettent le franchissement des lésions de l’ADN bloquant la progression de la fourche de réplication. Leur recrutement, lors des accidents rencontrés pendant la réplication, est le plus souvent protecteur pour le génome des eucaryotes supérieurs. In vitro au contraire, ces polymérases sont très mutagènes quand elles copient un ADN normal.
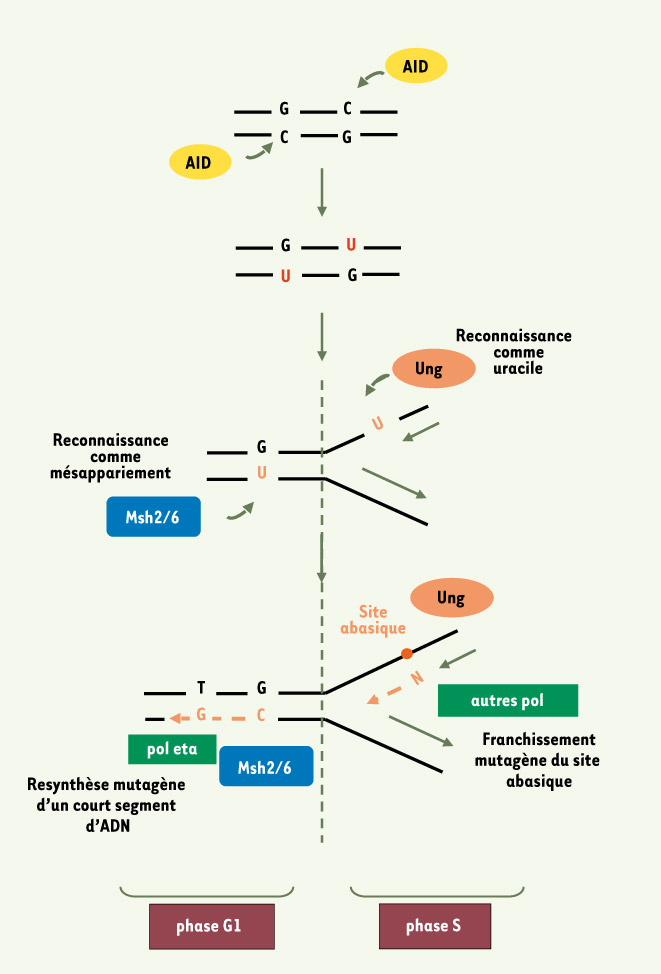
L’obtention de souris déficientes en mismatch repair et en ADN polymérase eta nous a récemment permis de proposer un schéma simplifié du processus d’hypermutation (Figure 1) [ 6, 8] : les bases A/T sont produites par l’action du mismatch repair recrutant l’ADN polymérase eta, et les mutations G/C créées pour l’essentiel par la réplication des sites abasiques produits par l’uracile glycosylase. Dans l’hypermutation, tout fonctionne donc « de travers » et concourt à l’accumulation d’erreurs introduites en cascade dans la gestion d’une lésion banale de l’ADN. L’uracile est reconnu en première intention comme un mésappariement G/U par un des composants du système de réparation normalement en charge des erreurs de la réplication (le complexe MSH2-MSH6). Ce complexe va recruter l’ADN polymérase eta pour « réparer » cette lésion en resynthétisant un court segment d’ADN, introduisant de ce fait de nouvelles mutations, essentiellement sur les bases A/T qui représentent la signature de cette enzyme. L’activité de l’uracile glycosylase se trouve ainsi confinée à la phase S du cycle cellulaire sur l’ADN simple brin, et son intervention, qui va créer des sites abasiques à l’approche de la fourche de réplication, sera tout naturellement mutagène, un site abasique ne portant pas d’information pour les polymérases translésionnelles capables de le franchir.
| Figure 1.
Vue schématisée du rôle des ADN polymérases translésionnelles dans le processus d’hypermutation des gènes des immunoglobulines. L’action de l’AID au locus des immunoglobulines désamine des cytosines en uraciles. Ces uraciles présents dans l’ADN double-brin vont être préférentiellement reconnus comme des mésappariements par le complexe MSH2-MSH6 (normalement en charge de la reconnaissance des mésappariements post-réplicatifs), et empêcher ainsi le fonctionnement de la voie classique de réparation par l’uracile glycosylase UNG. Cette reconnaissance enclenche une réparation à fort taux d’erreur, par le recrutement de l’ADN polymérase eta. La signature moléculaire de cette enzyme est caractérisée par une mutagenèse sur les bases A/T, son taux d’erreurs de synthèse étant le plus fort en face d’une thymidine. Lors de la phase S, UNG pourra accéder aux uraciles exposés sur l’ADN en cours de réplication et produire des sites abasiques. La réplication de ces sites fait appel à d’autres polymérases translésionnelles (Rev1 et probablement d’autres comme pol zeta [
10,
11]), dont l’action mutagène par incorporation d’une base quelconque (« N ») sera ciblée sur les bases G/C. Si le recrutement en phase S des polymérases translésionnelles correspond bien à leur rôle physiologique de franchissement des lésions bloquant la progression de la fourche de réplication, l’intervention du mismatch repair couplé à l’ADN polymérase eta apparaît, elle, comme un processus unique aux lymphocytes B engagés dans la réponse immunitaire (adapté de [8]). |
On retrouve donc les ADN polymérases dites translésionnelles à l’origine de toutes les mutations induites par l’AID, soit dans leur fonction spécifique (franchissement des sites abasiques), soit dans un rôle inhabituel et qui semble restreint aux cellules B (pour l’ADN polymérase eta). On observe ainsi curieusement, au cours du processus d’hypermutation, une résurgence du rôle mutagène des polymérases translésionnelles, tel qu’il a été proposé pour la réponse SOS chez E. coli [ 9], et l’on peut évidemment trouver quelque analogie entre la mutagenèse qu’entraîne une réponse au stress chez la bactérie et la variabilité induite sur le récepteur de la cellule B lors de la réponse à l’agression par un agent pathogène.
Une telle dérive des systèmes de maintenance de l’ADN est impressionnante, et la question se pose bien évidemment de savoir quels sont les signaux spécifiques au lymphocyte B qui permettent, lors de la réponse immunitaire, cette gestion non orthodoxe d’une lésion ubiquitaire de l’ADN. Plus généralement se pose également la question du ciblage de l’AID au locus des immunoglobulines, question difficile puisque l’hypermutation, au contraire de la recombinaison des gènes des immunoglobulines, n’implique pas la reconnaissance de signaux spécifiques. S’il est probable que la cellule tolère un certain degré de non-spécificité, le processus d’hypermutation ciblant un ou deux kilobases en aval d’un promoteur actif et la majorité des gènes ne comptant pas ou peu d’exons codants sur une telle distance, le locus des immunoglobulines constitue clairement la cible préférentielle du processus. Conformation spécifique du locus, dialogue particulier entre promoteur et enhancer, relocalisation dans le noyau à des sites constituant des mutation factories, beaucoup d’hypothèses sont ouvertes et restent à tester.