La production des protéines à partir de l’ARNm est un procédé stochastique qui permet de contrôler finement les fonctions cellulaires. Un des principaux défis de la recherche en biologie est de pouvoir fournir une description quantitative de ces évènements qui permettrait de mieux comprendre et, éventuellement d’agir, sur ces fonctions cellulaires lors de pathologies.
Certains travaux ont analysé le contenu protéique cellulaire en utilisant des techniques de microscopie de fluorescence permettant de compter des protéines à l’échelle de la molécule unique. Cependant ces approches, qui ne permettent de compter qu’un type de protéine à la fois, ont des applications limitées [ 1, 2]. De plus, elles sont restreintes aux cas où l’environnement cellulaire ne modifie pas la fluorescence des sondes utilisées pour détecter les protéines.
Or, une toute nouvelle approche, publiée dans la revue Science, permet de compter au niveau moléculaire plusieurs types de protéines exprimées au sein d’une cellule unique [ 3].
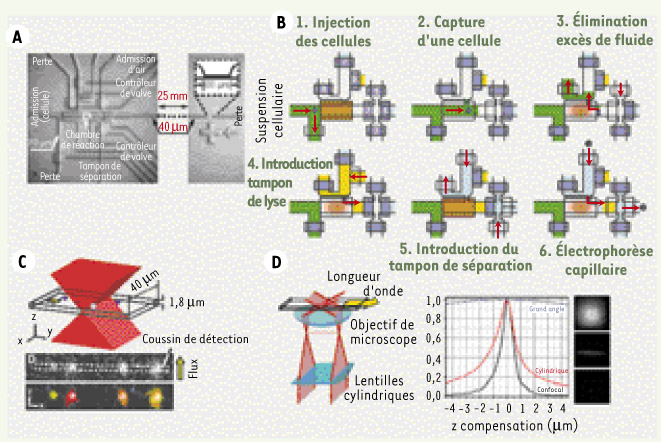
La stratégie expérimentale repose sur l’utilisation d’une cellule microfluidique dans laquelle il est possible de manipuler une seule cellule, d’en extraire le contenu protéique, de marquer ces protéines, les séparer et les quantifier en utilisant des techniques de microscopie de fluorescence adaptées au comptage de molécules uniques. Ce système microfluidique est composé de polydiméthylsiloxane [(C2H6OSi)n], plus connu sous le nom de silicone, ce qui lui confère des propriétés rhéologiques adaptées à l’utilisation des différents tampons pour la manipulation des cellules et de leur contenu. Il possède 3 sections principales (Figure 1A) : la première permet de manipuler les cellules, la deuxième de réaliser une électrophorèse capillaire et la troisième de compter les molécules fluorescentes séparées électrophorétiquement à l’échelle de la molécule unique.
| Figure 1.
Caractéristique et fonctionnement de la cellule microfluidique. A. Organisation générale de la cellule microfluidique avec, à gauche, la partie pour manipuler les cellules, suivie de la section où se réalise l’électrophorèse capillaire et, à droite, la plate-forme de comptage des protéines. B. procédure de l’analyse : (1) Injection des cellules ; la valve 1 s’ouvre et se ferme (rectangle violet si la valve est fermée et blanc si ouverte) jusqu’à ce qu’une cellule s’approche de la valve à 3états (rectangle orange). (2) La valve à 3états s’ouvre alors pour permettre de capturer une cellule dans la chambre de réaction (délimitée par la valve à 3 états et la valve 2). (3) La valve à 3 états est alors mise en position semi-ouverte (dégradé orange), l’ouverture de la valve 2 et 5 permet d’injecter de l’air et d’éliminer l’excès de fluide. (4) La valve 6 est alors ouverte pour permettre l’introduction du tampon nécessaire à la lyse de la cellule dans la chambre de réaction. (5) La valve 6 est ensuite refermée; la fermeture de la valve à 3 états et l’ouverture de la valve 7 et 8 permet d’introduire le tampon utilisé pour la séparation électrophorétique. (6) Finalement, la valve à 3 états est réglée en position semi-ouverte, les valves 2 et 4 sont ouvertes et l’électrophorèse capillaire est alors déclenchée. C. Caractéristiques du coussin de détection permettant de compter des molécules uniques. D. Modification du microscope confocal par des lentilles cylindriques pour l’obtention d’un champ d’excitation par le laser plus large. |
La manipulation des cellules et des différents tampons s’effectue via un système de valve à 2 ou 3 états : ouvert, fermé et semi-ouvert (les valves à 2 états sont représentées par un rectangle violet et celles à 3 états par un rectangle orange sur la Figure 1B). Ces valves permettent de capturer la cellule dans la chambre de réaction et d’introduire les réactifs pour lyser la cellule et marquer le contenu protéique si nécessaire (en jaune sur la Figure 1B). En effet, dans le cas où les protéines d’intérêt ne sont pas naturellement fluorescentes, elles peuvent être marquées par des anticorps couplés à une molécule fluorescente. Après la réaction, l’excès d’anticorps fluorescent peut être séparé de la protéine d’intérêt par électrophorèse capillaire. Finalement, à la fin du canal de séparation, les molécules fluorescentes sont comptées au sein d’un coussin de détection dont les caractéristiques sont présentées dans la Figure 1C. Celui-ci est généré par utilisation d’un microscope confocal modifié par la présence de lentilles cylindriques qui permettent d’élargir le champ d’excitation par le laser. La Figure 1D montre la différence entre le système confocal et celui qui est modifié par les lentilles cylindriques. La fluorescence des molécules est alors enregistrée par une caméra CCD (charge-coupled device) permettant ainsi de calculer le nombre de molécules qui traversent le coussin de détection.
Ce système microfluidique a d’abord été utilisé pour compter le nombre de copies d’une protéine membranaire de la superfamille des récepteurs couplés aux protéines G, le récepteur β2-adrénergique, exprimé dans une cellule d’insecte (SF9) par infection virale. Ce récepteur possède une étiquette moléculaire (FLAG) qui est reconnue par un anticorps monoclonal fluorescent (M1-Cy5). Après capture de la cellule dans la chambre de réaction, les récepteurs sont extraits de la membrane plasmique par l’utilisation d’un détergent, le dodécylmaltoside, et marqués par M1-Cy5. Les complexes récepteur/M1-Cy5 sont alors séparés et comptés. Il a été ainsi possible de compter un très petit nombre de récepteurs (de 2 000 jusqu’à 57 000 copies par cellule).
Cette approche a ensuite permis de quantifier plusieurs protéines présentes dans une cyanobactérie1, (Synechococcus sp. PCC 7942). Les protéines étudiées, qui sont auto-fluorescentes, font partie d’un complexe protéique photosynthétique (le phycobilisome, PBS)2 qui permet d’absorber l’énergie lumineuse et de la transférer au centre réactionnel de la photosynthèse [ 4]. Le comptage de ces protéines a été réalisé pour un très petit nombre de copies (600) et dans différentes conditions de culture. L’analyse des résultats du comptage a permis de mettre en évidence qu’en l’absence d’une source d’azote, la cyanobactérie déclenche de façon coordonnée la dégradation de ces protéines au sein du PBS.
Cette méthode innovante permet donc de compter directement les protéines exprimées dans une cellule unique, et cela, même si le nombre de ces protéines est très faible. Les protéines peu abondantes jouent un rôle majeur dans les fonctions cellulaires. Cette méthode va donc trouver des applications dans une multitude de domaines de la biologie cellulaire, comme dans l’étude des cancers. Ces dernières années, l’utilisation de puces à ADN a révélé l’extraordinaire complexité des modifications génomiques responsables de la formation d’une cellule cancéreuse. La technique décrite ici permettrait d’analyser dans des cellules uniques les niveaux d’expression des protéines identifiées par les études de génomique. En répétant cette analyse à partir des différentes sous-populations de cellules cancéreuses, il serait aussi possible d’analyser l’hétérogénéité cellulaire au sein d’un même tissu cancéreux, et de mieux comprendre les mécanismes cellulaires qui permettent au cancer d’évoluer, de développer des résistances, et d’envahir le reste de l’organisme. Cette méthode ouvre donc des perspectives nouvelles pour le développement de la protéomique et de la génomique fonctionnelle.