| |
| Med Sci (Paris). 2007 June; 23(6-7): 592–594. Published online 2007 June 15. doi: 10.1051/medsci/20072367592.Diagnostic prénatal non invasif de la trisomie 21 Chimère ou réalité ? Patrizia Paterlini-Bréchot* Inserm U807, Faculté de Médecine Necker-Enfants malades, Université Paris V, 156, rue de Vaugirard, 75015 Paris, France MeSH keywords: ADN, Syndrome de Down, Femelle, Maladies foetales, Humains, Placenta, Grossesse, Diagnostic prénatal, Transcription génétique |
Malgré les progrès remarquables de la médecine moderne, un diagnostic prénatal fiable de la trisomie 21 est possible, à l’heure actuelle, uniquement par des méthodes invasives, notamment l’amniocentèse et la biopsie des villosités choriales, qui sont associées à un risque non négligeable de fausse-couche (0,8 % à 3 % des cas). Depuis une quinzaine d’années, les chercheurs ont essayé d’isoler et analyser les cellules fœtales rarissimes (environ une par ml de sang) qui circulent dans le sang maternel (CFC, circulating fetal cells). Après la publication en 2002 [
1] d’une étude multicentrique américaine rapportant l’isolement d’au moins une cellule fœtale chez seulement 41,4 % des mères, la recherche des CFC a été abandonnée par plusieurs groupes de recherche au profit de l’étude des acides nucléiques d’origine fœtale circulant sous forme libre dans le plasma. La proportion de l’ADN fœtal dans le plasma (3,4 %) peut être augmentée en ajoutant de la formaldéhyde au prélèvement sanguin. Les indications actuelles de l’utilisation de l’ADN fœtal concernent la détermination du sexe fœtal et le génotypage RHD (Rhésus D) [
2,
3], qui ciblent des séquences absentes dans l’ADN maternel. Deux travaux récents ont exploré la faisabilité de l’utilisation des acides nucléiques d’origine fœtale circulant sous forme libre dans le plasma pour le diagnostic d’aneuploïdie. |
Quantification des transcrits fœtaux Le travail de Dennis Lo et al. [
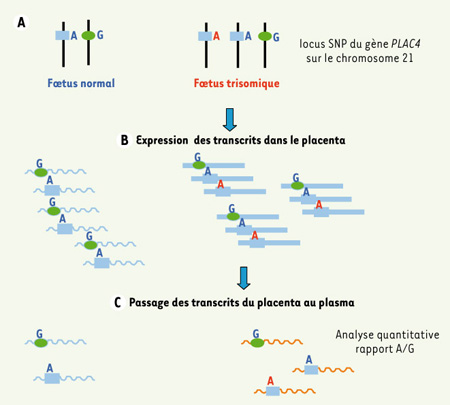
4] (Figure 1) utilise le génotypage par SNP (single nucleotide polymorphism) et la quantification des transcrits du gène PLAC4 (placenta specific protein 4) situé sur le chromosome 21, exprimés de façon prévalente au niveau du placenta et dont une proportion passe dans la circulation [
5]. La méthode implique l’extraction des ARN du plasma, l’amplification par RT-PCR d’un locus SNP (rs8130833) sur le chromosome 21 (locus qui n’est informatif que dans 45 % de la population) et l’analyse quantitative par spectrométrie de masse des produits d’amplification. Les auteurs ont défini les variations du ratio entre les deux allèles quantifiés chez les femmes portant un fœtus euploïde et, par comparaison, ont effectué le diagnostic de trisomie 21 quand ce ratio dépassait ces limites (par excès quantitatif d’un allèle ou de l’autre). La méthode a permis de classer correctement 9 cas sur 10 de trisomie 21 et 55 cas sur 57 d’euploïdie avec une sensibilité de 90 %, une spécificité de 96,5 % et un taux de faux positifs de 2,98 %.  | Figure 1.
Principe de la méthode utilisée par D. Lo et al. [ 4] . A. En cas d’hétérozygotie du locus SNP (rs8130833) sur le gène PLAC4 (taux d’hétérozygotie dans la population caucasienne: 45%), le foetus normal a une base différente sur chacun des deux allèles (ici A et G). Chez le fœtus trisomique, il y a duplication d’un allèle. B. En cas de trisomie 21, les transcrits du gène PLAC4 au niveau du placenta sont caractérisés par une quantité plus importante de transcrits avec A par rapport aux transcrits avec G (si le taux de transcription des trois allèles du gène est le même). C. Il y a une réduction du nombre de transcrits PLAC4 du placenta vers le plasma, mais, si la proportionnalité entre transcrits est maintenue, le nombre des transcrits avec A est supérieur à celui des transcrits avec G. |
Cette méthode, qui a pu être mise au point actuellement avec un seul marqueur (rs8130833), présent sous forme hétérozygote chez 45 % des sujets, cible environ la moitié de la population. Si la combinaison de plusieurs marqueurs permet son extension, elle pourrait être applicable à environ 95 % de la population caucasienne. Techniquement, l’approche est fondée sur l’amplification quantitative des ARN placentaires transférés dans le plasma maternel. Il est donc essentiel que la proportionnalité quantitative des transcrits provenant des deux ou trois allèles du gène PLAC4 dans le plasma maternel soit identique à ce qu’elle est dans le placenta. Si tel est le cas, les transcrits exprimés par les deux allèles identiques seront en quantité supérieure à celle des transcrits issus de l’autre allèle dans le plasma des femmes avec fœtus trisomique. Toutefois, de façon surprenante, la quantité globale des ARN PLAC4 dans le plasma des mères avec fœtus trisomique n’était pas supérieure à celle détectée dans le plasma des femmes dont le fœtus était normal [4]. Il faut donc imaginer que des problèmes relatifs au nombre de copies des transcrits du gène exprimés par allèle et/ou à leur amplification et/ou à leur passage dans le plasma maternel pourraient expliquer ce résultat et constituer un point critique pour la mise au point d’une méthode fiable en prévision de sa diffusion à large échelle. |
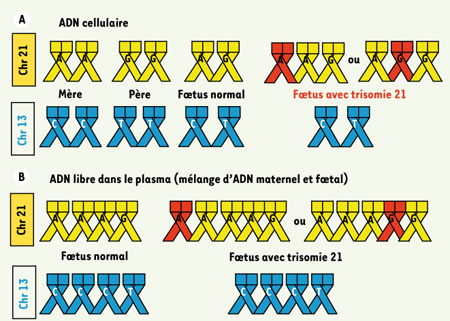
Ou quantification d’allèles des chromosomes 13 et 21 Le travail de R. Dhallan et al. [
6] (Figure 2) cible l’ADN fœtal libre dans le plasma, après un traitement du sang avec du formaldéhyde. Les auteurs détectent ainsi une proportion moyenne d’ADN fœtal de 34 % par rapport à l’ADN libre maternel. L’ADN plasmatique du sang maternel est analysé par amplification avec des amorces spécifiques de SNP informatifs ciblant les chromosomes 13 et 21. Le produit d’amplification, obtenu avec des amorces biotinylées portant un site de restriction, est capturé sur un support solide par une liaison avidine-biotine, coupé, marqué avec des nucléotides fluorescents, et déposé sur gel de séquence. Le signal est « pixelisé » et quantifié. Le test est basé sur le rapport entre l’intensité du signal de l’allèle paternel et celui de l’allèle maternel à la fois pour les SNP du chromosome 13 et ceux du chromosome 21. Un rapport de 0,333 est attendu en cas d’euploïdie (exemple : 1T/3C) et de 0,25 (exemple : 1G/4A) en cas de trisomie 21 (Figure 2). Les auteurs ont étudié 57 femmes porteuses d’un fœtus euploïde et 3 femmes porteuses d’un fœtus ayant une trisomie 21. Cinquante-six cas normaux ont été identifiés correctement ainsi que 2 des 3 trisomiques (sensibilité : 66,7 %, spécificité : 98,2 %, taux de faux positifs : 1,6 %).  | Figure 2.
Principe de la méthode utilisée par R. Dhallan et al. [ 6] . A. Exemple de SNP informatif ; allèles sur les chromosomes 21 et 13 au niveau cellulaire. B. Allèles des chromosomes 21 et 13 au niveau de l’ADN du plasma (mélange d’ADN maternel et fœtal). Si le fœtus est normal, les rapports des signaux (analyse quantitative) des allèles G et A (1/3 = 0,333) sur le chromosome 21 et T et C (1/3 = 0,333) sur le chromosome 13 sont identiques. Si le fœtus est atteint de trisomie 21, les rapports des signaux (analyse quantitative) des allèles G et A (1/4 = 0,25 ou 2/3 = 0.666) sur le chromosome 21 et T et C (1/3 = 0,333) sur le chromosome 13 sont différents. |
La performance de cette méthode, dont la sensibilité est inférieure à celle des tests sérologiques, ne peut pas être évaluée en raison du nombre trop faible des cas avec trisomie 21. L’approche technique est intéressante, mais il est clair que sa fiabilité dépend, comme les auteurs le remarquent, de la proportion relative de l’ADN fœtal par rapport à l’ADN maternel, variable d’un prélèvement à l’autre, et du nombre de SNP informatifs qui peuvent être détectés sur les chromosomes 13 et 21. Il est intéressant de noter qu’une différence significative entre les rapports des deux allèles calculés sur le chromosome 13 et sur le chromosome 21 n’a été trouvée que dans trois cas sur les 60 testés [6]. Deux de ces cas ont permis le diagnostic correct de trisomie 21, le troisième a été interprété comme une trisomie 21, mais il s’agissait d’un faux positif (le pourcentage absolu d’ADN fœtal était de 21,7 % et le nombre de SNP trouvés sur les deux chromosomes était bas). Enfin, un autre cas, pour lequel la proportion d’ADN fœtal était de 31,8 % et le nombre de SNP utilisés plus important, a été interprété comme normal alors qu’il s’agissait d’une trisomie 21. |
Ces travaux soulignent les difficultés auxquelles se heurte l’utilisation des acides nucléiques fœtaux libres dans le plasma maternel pour la mise au point d’un diagnostic fiable de trisomie 21. Le dépistage non invasif, réalisé actuellement au second trimestre (marqueurs sériques et analyse échographique de l’épaisseur de la nuque), a une sensibilité de 82,5 % et un taux de faux positifs de 5 % alors que le dépistage combiné réalisé au premier et au second trimestre a une sensibilité de 89,7 % et un taux de faux positifs de 4,2 % [
7]. Les limites des méthodes moléculaires, réalisées à partir d’échantillons plasmatiques, ne permettent pas, en l’état actuel, de les proposer en complément des méthodes de dépistage actuelles (ce qui conduirait à augmenter les coûts sans véritable avantage diagnostique) ni en remplacement des méthodes diagnostiques invasives. Il apparaît clair que, si les acides nucléiques fœtaux libres dans le plasma sont d’obtention facile, leur mélange avec ceux de la mère impose des développements techniques dont la reproductibilité et la validation ne sont pas aisées. Par ailleurs, l’isolement par ISET (isolation by size of epithelial tumor/trophoblastic cells) des cellules trophoblastiques à partir du sang maternel constitue un espoir tangible car il permet de réaliser les analyses génétiques sur de l’ADN fœtal pur, non mélangé à l’ADN maternel [
8–
10]. La mise au point d’une méthode fiable et applicable à large échelle de diagnostic prénatal non invasif de la trisomie 21 n’est sûrement pas une chimère, mais reste un défi pour la médecine moderne. |
1. Bianchi DW, Simpson JL, Jackson LG, et al. Fetal gender and aneuploidy detection using fetal cells in maternal blood : analysis of NIFTY I data. National Institute of Child Health and development fetal cell isolation study. Prenat Diagn 2002; 22 : 609–15. 2. Bianchi DW, Avent ND, Costa JM, Van der Schoot CE. Noninvasive prenatal diagnosis of fetal Rhesus D: ready for Prime(r) Time. Obstet Gynecol 2005; 106 : 841–4. 3. Costa JM, Benachi A, Gautier E, et al. First-trimester fetal sex determination in maternal serum using real-time PCR. Prenat Diagn 2001; 21 : 1070-4 et 2002; 22 : 1242. 4. Lo YM, Tsui NB, Chiu RW, et al. Plasma placental RNA allelic ratio permits noninvasive prenatal chromosomal aneuploidy detection. Nat Med 2007; 13 : 218–23. 5. Tsui NB, Ng EK, Lo YM. Molecular analysis of circulating RNA in plasma. Methods Mol Biol 2006; 336 : 123–34. 6. Dhallan R, Guo X, Emche S, et al. A non-invasive test for prenatal diagnosis based on fetal DNA present in maternal blood: a preliminary study. Lancet 2007; 369 : 474–81. 7. Rozenberg P, Bussieres L, Chevret S, et al. Screening for Down syndrome using first-trimester combined screening followed by second-trimester ultrasound examination in an unselected population. Am J Obstet Gynecol 2006; 195 : 1379–87. 8. Vona G, Béroud C, Benachi A, et al. Enrichment and genetic analyses of fetal cells circulating in the maternal blood by the ISET technique and single cell microdissection : a non-invasive tool for early prenatal diagnosis. Am J Pathol 2002; 160 : 51–8. 9. Béroud C, Karliova M, Bonnefont JP, et al. Prenatal diagnosis of spinal muscular atrophy (SMA) by genetic analysis of circulating fetal cells. Lancet 2003; 361 : 1013–4. 10. Saker A, Benachi A, Bonnefont JP, et al. Genetic characterization of circulating fetal cells allows non invasive prenatal diagnosis of cystic fibrosis. Prenat Diagn 2006; 26 : 906–16. |