Une étape importante pour la compréhension du rôle des modifications épigénétiques de la chromatine dans la régulation de la pluripotence des cellules de l’embryon de mammifère a été franchie avec une étude publiée récemment dans le journal Nature par l’équipe de M. Zernicka-Goetz [ 1]. Les bases de ce travail avaient été posées en 2005 avec la publication par cette même équipe d’une étude établissant qu’en fonction de l’orientation et de la séquence des clivages qui leur donnent naissance, les blastomères composant l’embryon de souris au stade 4 n’ont pas les mêmes capacités développementales, et que les destins de leur descendance peuvent être drastiquement différents [ 2].
Le premier plan de clivage du zygote, environ vingt-quatre heures après la fécondation, passe par son axe animal-végétal (AV), marqué au pôle animal par la présence du second globule polaire. Ce type de clivage est dit méridional (M). Le clivage de chacun des deux blastomères résultant peut être soit méridional, soit équatorial (E), c’est-à-dire perpendiculaire à l’axe AV (Figure 1A). Ces deux blastomères ne se divisent pas en même temps et l’embryon passe donc par un stade intermédiaire où il ne compte que trois blastomères.
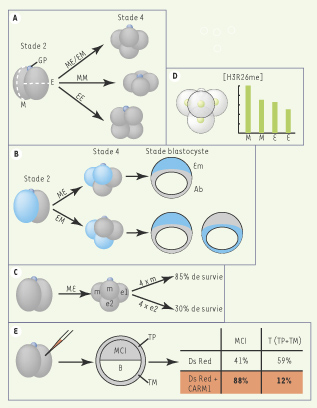
| Figure 1.
Modifications épigénétiques de la chromatine dans la régulation de la pluripotence des cellules de l’embryon de souris. A. L’origine des trois types de morphologie de l’embryon de souris au stade 4, de haut en bas, tétraédrique, en étoile, plane. M, clivage méridional ; E, clivage équatorial ; gp, deuxième globule polaire. B. Relation entre la séquence de clivage des embryons tétraédriques et la contribution des descendants des deux premiers blastomères aux régions embryonnaire (Em) et ab-embryonnaire (Ab) du blastocyste. C. Capacité des chimères d’agrégation des blastomères m ou e2 à se développer. D. Distribution et quantification relative de la marque H3R26me dans un embryon ME. E. L’injection de la méthyl-transférase CARM1 dans un blastomère au stade 2 a pour effet d’augmenter la contribution de ses descendants à la masse cellulaire interne au stade blastocyste. B : blastocèle ; MCI : masse cellulaire interne ; TP : trophectoderme polaire ; TM : trophectoderme mural. |
Au stade 4, trois morphologies embryonnaires sont distinguées, que l’on peut décrire comme tétraédrique, plane et en étoile (Figure 1A). Des études de lignage, après marquage d’une cellule au stade 2 blastomères, ont permis de comprendre leur origine [ 3]. La majorité (81 %) des embryons au stade 4 ont une morphologie tétraédrique qui résulte d’une séquence de clivage ME ou EM. Les embryons dont la morphologie est en étoile (11 %) sont le résultat d’une séquence MM, les embryons dont la morphologie est plane (8 %), d’une séquence EE. Il avait été noté précédemment que pour une proportion importante des embryons, la descendance du premier blastomère à s’être divisé contribue de façon majoritaire à la région embryonnaire du blastocyste, comprenant la masse cellulaire interne (MCI, dont les descendants formeront les tissus fœtaux) et le trophectoderme polaire (tissu extra-embryonnaire). Dans ce cas, la descendance du deuxième blastomère contribue préférentiellement à la région ab-embryonnaire, comprenant la partie de la MCI bordant la cavité du blastocèle et le trophectoderme mural. Cependant, ce type de répartition dépend très étroitement de la façon dont les deux premiers blastomères se sont divisés et ne représente pas le schéma général pour tous les embryons. De façon remarquable, il a été montré que c’est seulement dans le cas d’une séquence de clivage ME qu’il est possible de prédire la future polarité du blastocyste (Figure 1B). Dans le cas d’un clivage EM, la descendance du premier blastomère a des chances égales de contribuer soit à la région embryonnaire, soit à la région ab-embryonnaire du blastocyste. Pour les séquences de clivage MM et EE, l’allocation des cellules descendant des deux premiers blastomères n’est pas restreinte à l’une ou l’autre de ces régions.
Les embryons ME sont donc les seuls permettant de prédire le destin de chacun des quatre blastomères les constituant. Des expériences d’agrégation ont permis de préciser les capacités développementales de chacun de ces blastomères [2]. Le blastomère e2, résultant de la division E et héritant de la majorité du cytoplasme de la région végétative, contribue principalement au trophectoderme mural, et ne permet que rarement d’obtenir un embryon viable lorsqu’il est agrégé à trois autres cellules d’origine identique (Figure 1C). En revanche, l’agrégation de blastomères m, issus de la division M, permet d’obtenir avec un taux comparable aux contrôles le développement à terme des embryons chimères résultants. Il semble donc que ces blastomères diffèrent dans leur capacité à permettre le développement d’un embryon, ce qui suggère le partage inégal entre eux d’une information ou d’une activité préexistante.
Les marques épigénétiques qui décorent la chromatine ont souvent été proposées comme étant susceptibles de véhiculer une information importante pour la régulation de la pluripotence des cellules embryonnaires. Tirant parti de leur connaissance approfondie des séquences de clivage chez l’embryon de souris, Torres-Padilla et al. se sont intéressés à la distribution au stade 4 de trois modifications de l’histone H3, résultant de la méthylation d’arginines aux positions 2, 17 ou 26. L’utilisation d’anticorps spécifiques pour chacune de ces modifications a permis leur quantification relative au sein des noyaux d’un même embryon [1]. Il a ainsi été montré que pour une fraction des embryons au stade 4, le taux de méthylation n’est pas le même dans chaque blastomère. Les contrastes les plus élevés étant obtenus pour la modification H3R26me, c’est en suivant celle-ci que les investigations se sont poursuivies. Torres-Padilla et al. ont démontré que ce sont les blastomères des embryons ME qui présentent les variations les plus importantes, et que le niveau de méthylation détecté dans chaque noyau est étroitement corrélé à l’identité du blastomère considéré. Les blastomères e2 présentent ainsi un taux de méthylation qui peut être inférieur de moitié à celui du blastomère m présentant le taux le plus élevé dans le même embryon (Figure 1D). De façon intéressante, les embryons EM, dont la polarité ne peut être prédite, présentent dans les noyaux de leurs blastomères des niveaux de méthylation beaucoup plus proches les uns des autres. Au vu de ces résultats, un lien causal a été recherché entre l’activité de méthylation de l’histone H3 dans un blastomère et le devenir du blastomère en question.
Les arginines 2, 17 et 26 de l’histone H3 sont les cibles d’une même méthyl-transférase, appelée CARM1. Les auteurs ont montré que chez l’embryon, CARM1 est exprimée à partir d’un messager maternel, et qu’il y a une corrélation entre la répartition de la protéine et l’intensité du marquage H3R26me dans les noyaux d’un embryon de stade 4. Afin de tester son impact sur le destin et la potentialité des cellules, l’ARN de CARM1, ainsi que celui du traceur fluorescent DsRed, ont été co-injectés dans un blastomère unique au stade 2. L’analyse des embryons injectés au stade blastocyste a permis de montrer que la descendance des blastomères surexprimant CARM1 contribue de façon prédominante à la MCI (Figure 1E). L’utilisation d’une forme mutée de CARM1 a confirmé ce résultat en démontrant que c’est bien l’activité méthyl-transférase qui est requise. L’accroissement de l’activité CARM1 s’accompagne bien sûr d’une augmentation du marquage H3R26me, mais également d’une expression plus forte des facteurs Nanog et Sox2 qui sont étroitement associés au maintien et à la pluripotentialité des cellules de la MCI.
Cette étude est la première à établir ainsi un lien direct entre la présence d’une marque épigénétique et la détermination d’un destin cellulaire chez un embryon de mammifère. Elle ne permet pas d’expliquer ce qui se passe chez les embryons qui ne subissent pas une séquence de clivage ME, et dont la plupart se développeront pourtant normalement. Cependant, elle démontre une fois de plus que se focaliser sur un cas particulier permet certaines avancées, et suggère une route possible pour de futures investigations. Rappelons qu’à ce jour près de soixante-dix marques épigénétiques sont connues, et en quête de fonctions. De plus, cette étude met en évidence des différences marquées entre les blastomères d’un embryon qui commence à peine à se développer. Nul doute que ces données contribueront au débat très animé que suscite la possibilité d’une régionalisation précoce de l’embryon de souris [ 4– 7].