| |
| Med Sci (Paris). 2007 August; 23(8-9): 735–740. Published online 2007 August 15. doi: 10.1051/medsci/20072389735.De la régulation du génome à la progression tumorale Acétylation de la lysine 16 de l’histone H4 Benoit Miotto* and Kevin Struhl Department of Biological Chemistry and Molecular Pharmacology, Harvard Medical School, 240 Longwood Avenue, Boston, Massachusetts 02115, États-Unis |
Dans le noyau, la double hélice d’ADN est condensée en une fibre de chromatine dont l’unité élémentaire est le nucléosome. Loin d’être une simple « brique » architecturale, celui-ci est un élément essentiel du processus de régulation du génome. En effet, l’enroulement de l’ADN autour d’un octamère de protéines histones pour former le nucléosome constitue une barrière naturelle pour les machineries nucléaires nécessitant d’avoir accès au matériel génétique. En conséquence, l’altération de la structure nucléosomique ou de l’agencement des nucléosomes le long de la double hélice, en affectant le degré d’accessibilité de l’ADN, régule de nombreux processus biologiques et pathologiques de la cellule [
1,
2]. Parmi les différentes voies de régulation qui ont été caractérisées et agissent souvent de manière coordonnée, la plus étudiée reste le processus de modification post-traductionnelle des queues amino-terminales des histones. Les quatre histones nucléosomiques (H2A, H2B, H3 et H4) exposent à des modifications les résidus cibles de leurs queues amino-terminales qui émergent à la surface du nucléosome. Ces modifications (méthylation, acétylation, phosphorylation) pouvant survenir au sein de résidus de différentes queues d’histones ou au sein d’une même queue, engendreraient différentes combinatoires. Ce « code histone » serait ensuite traduit en un état chromatinien particulier par des protéines régulatrices recrutées spécifiquement dans des régions plus ou moins vastes du génome par certaines combinaisons [
3]. Loin de considérer les multiples données susceptibles de cerner la compréhension actuelle du « code histone », cette synthèse se limite à l’observation d’une modification particulière de H4 : l’acétylation de la lysine 16 (K16). Phénomène majeur chez les eucaryotes supérieurs, des études récentes ont démontré le caractère singulier de cette modification dans la régulation du génome. |
Acétylation : enzymes et régulation Les modifications des histones sont réversibles. Dans le cas de l’acétylation, des études de cinétique, avec pour modèle la levure, ont estimé à quelques minutes seulement le retour vers un état d’acétylation normal ou « de base », à partir d’un état hypo-acétylé ou hyper-acétylé [
4]. Le niveau d’acétylation d’un résidu reflète, en fait, la différence d’activité entre deux familles d’enzymes antagonistes, les histones acétyltransférases (HAT) et les histones désacétylases (HDAC). Ces HAT/HDAC se recrutent au sein de régions cibles spécifiques, et, de plus, modifient le nucléosome à travers le génome entier indépendamment de toute séquence ADN spécifique [
5–
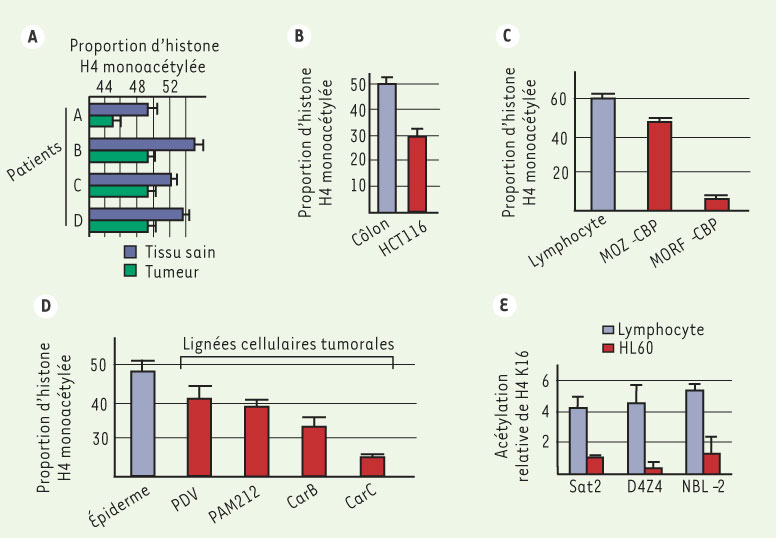
7]. De fait, chaque histone peut être acétylée à différents niveaux en fonction de l’activité sélective et globale des HAT/HDAC. De nombreuses enzymes capables de modifier K16 ont été caractérisées (Tableau I). Mais de façon surprenante, K16 est le seul résidu d’histone pour lequel des HAT/HDAC spécifiques ont été détectées. Il en est ainsi chez l’homme et la drosophile de l’HAT MOF ou bien encore du couple HAT Sas2 et HDAC Sir2 chez la levure [5–
10].  | Tableau I
Liste des HAT et HDAC modificatrices de la lysine 16 de l’histone H4. L’équilibre de l’état d’acétylation de la lysine 16 est maintenu par les activités opposées des HAT et HDAC répertoriées dans cette figure. L’acétyl coenzymeA est le donneur du groupement acétyl transféré par les HAT sur le groupement ε-NH3+ des lysines des domaines amino-terminaux des histones. La réaction inverse est catalysée par les HDAC. * Enzyme spécifique de la Lys-16. ** Données obtenues dans un système hétérologue, la protéine Chameau étant capable de modifier la Lys-16 de H4 dans le contexte de sur-expression dans les cellules humaines. |
|
Lysine 16 et autres sites d’acétylation de H4 De nombreuses études d’immunodétection in vivo menées à l’aide d’anticorps spécifiques de chacun des sites d’acétylation, du microséquençage ou de la spectrométrie de masse ont démontré que le pool de H4 est acétylé majoritairement au niveau de K16 chez les mammifères [
11–
13]. De plus, l’abondance des autres sites acétylés de H4 (soit les lysines 5, 8 et 12) ne serait pas aléatoire mais en concordance avec une décroissance qui se manifeste vers la position la plus amino-terminale, de + 12 à + 5 [
12]. Il a certes été admis durant plusieurs années que l’acétylation de K16 était requise pour que l’acétylation des autres lysines de H4 se produise et/ou qu’elle était la plus résistante à l’activité des HDAC grâce à sa localisation plus éloignée de la position + 1 [12]. Toutefois, l’étude moléculaire de la déplétion de MOF dans des cellules humaines a démontré que la perte d’acétylation de K16 n’affectait pas l’acétylation des autres lysines de H4 [5, 7]. Il est de fait peu probable que l’acétylation de K16 soit un pré-requis à toute autre réaction d’acétylation de H4 quoique son abondance suggère clairement un rôle distinct de celui des autres sites d’acétylation. |
Lysine 16 et régulation de l’expression des gènes Historiquement, l’acétylation a été considérée comme un processus de neutralisation des charges positives des queues d’histone qui entraînerait une décondensation de la chromatine et l’instauration d’un état chromatinien adéquat pour la transcription. De nombreuses études, fondées notamment sur des immunoprécipitations de la chromatine (ChIP) avec pour intermédiaires des anticorps spécifiques de la queue H4 acétylée (parfois d’une position donnée), couplées à des analyses d’expression des gènes, ont été menées pour élucider l’effet de l’acétylation sur le processus de transcription. Certaines études ont non seulement fourni la cartographie et la description des portions acétylées de différents génomes, mais, de plus, elles ont démontré que dans les régions promotrices des gènes transcrits les histones étaient globalement hyper-acétylées [
14,
15]. Toutefois, ces études n’ont pas permis de corréler les niveaux de transcription à l’acétylation d’une lysine donnée de H4, probablement à cause d’une redondance entre lysines, mais plus simplement, dans la plupart des cas, par suite de l’absence d’étude cinétique du processus d’initiation de la transcription. À l’inverse, l’étude de l’effet de la mutation de chacune des lysines acétylables de H4 sur l’expression des gènes chez la levure a mis en évidence un rôle particulier de K16 [15]. En effet, seul le remplacement de K16 par l’arginine (mimant un état non-acétylé) entraîne un profil d’expression des gènes distinct (~100 gènes) indépendamment de la mutation des autres lysines de H4. À l’inverse, les autres mutations présentent des défauts d’expression génique similaires aggravés par l’augmentation du nombre de lysines mutées. L’acétylation de K16 serait donc interprétée par un mécanisme spécifique contrairement aux autres lysines. De plus, elle pourrait dicter un état transcriptionnel actif indépendamment des autres positions acétylées de H4. Il est probable que la densité nucléosomale dans les régions promotrices (souvent affectée au cours du processus de transcription) ou encore la nature des séquences régulatrices, non prises en compte dans ces études, contribue au phénomène. Toutefois, des expériences de ChIP à l’échelle du génome jointes à des analyses d’expression des gènes ont permis d’associer un niveau élevé de méthylation de H3 K4 et d’acétylation de K16 dans les régions promotrices de certains gènes transcriptionnellement actifs. Plus récemment, par fractionnement biochimique, il a été démontré qu’existe une association stable entre MOF et MLL1, la méthyltransférase de H3 K4 [
6]. De plus, les deux activités enzymatiques sont requises in vitro pour l’expression d’un gène rapporteur dans un système de transcription et in vivo pour l’expression du gène HoxA9 [6]. Ainsi le co-recrutement de MOF et MLL1 expliquerait le rôle singulier de l’acétylation de K16 à certains locus et illustre parfaitement la notion de « code histone ». |
Acétylation de la lysine 16 et décryptage du « code histone » Des analyses génétiques, de ChIP ou d’interaction protéique in vitro ont aussi démontré que l’acétylation de K16 régulait l’association de plusieurs facteurs chromatiniens avec leur(s) cible(s) génomique(s) comme le suggère le postulat du « code histone ». Ainsi, la protéine Sir3 de levure et les facteurs humains TBL1/TBLR1 se lient préférentiellement aux queues H4 déacétylées de K16 [
9,
16]. À l’inverse, la sous-unité TAF1 du complexe d’initiation de la transcription des métazoaires, la sous-unité TIP5 du complexe NoRC humain et l’HAT Gcn5 de levure interagissent préférentiellement avec H4 acétylée au niveau de K16 [
17–
19]. De manière intéressante, les trois derniers facteurs arborent un module protéique particulier, le bromodomaine, capable de lier sélectivement des lysines acétylées. L’acétylation de K16 régule aussi l’activité intrinsèque de plusieurs enzymes, elles-mêmes susceptibles de modifier la structure chromatinienne. Ainsi, l’activité lysine 5 et 12 acétyltransférase de HAT1 in vitro et la fonction de remodelage de la chromatine de ISWI chez la drosophile sont inhibées par l’acétylation de K16 [
20–
23]. Pour finir, des données de ChIP à l’échelle du génome indiquent que le niveau d’acétylation de K16 participerait à la sélection des sites de liaison d’un facteur de transcription liant une séquence ADN spécifique [
24]. L’ensemble de ces données illustre le caractère pléiotrope de l’acétylation de K16 dans le processus de régulation de la transcription. Celle-ci ne dicte pas toujours la même séquence d’événements moléculaires et peut aller jusqu’à entraîner des réponses transcriptionnelles (et biologiques) opposées. Ainsi, dans les régions télomériques chez la levure, la déacétylation de K16 permet la propagation de la structure hétérochromatinienne (Figure 1) tandis que l’acétylation de K16 promeut la répression transcriptionnelle des gènes ribosomiques par le complexe NoRC [
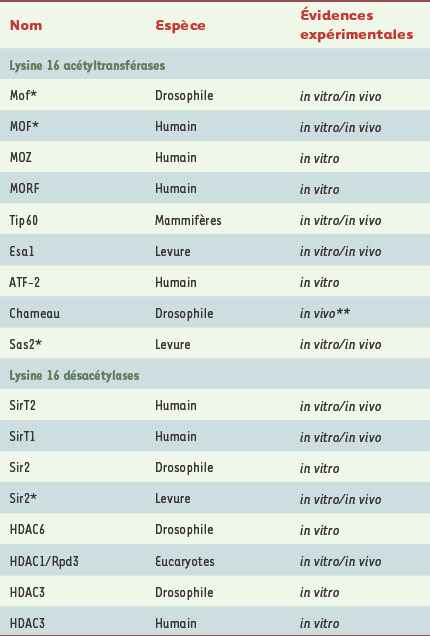
8, 9, 17].  | Figure 1.
Mode d’action proposé de l’acétylation de la lysine 16 de H4 dans le processus de propagation de l’hétérochromatine télomérique chez la levure. Le positionnement de la frontière euchromatine/hétérochromatine constitue un modèle d’étude idéal de l’intégration des activités histones modificatrices, de remodelage de la chromatine et d’échange des histones. A. La distribution complémentaire de l’HAT Sas2 et de la HDAC Sir2, respectivement dans l’euchromatine et dans l’hétérochromatine, induit la formation d’un gradient d’acétylation de la lysine 16 au sein des régions sub-télomériques. L’acétylation de la lysine 16 inhibe l’association du complexe SIR avec la chromatine pour engendrer une structure chromatinienne « ouverte ». À l’inverse, la déacétylation de la lysine 16 promeut le recrutement du complexe SIR et la formation de la structure hétérochromatinienne. Ce processus est très dynamique et a pu être établi par l’analyse génétique et moléculaire de plusieurs mutants affectant le positionnement de la frontière. B. Dans un contexte mutant pour Sas2 (∆Sas2), l’absence d’acétylation de la lysine 16 permet l’interaction de la protéine hétérochromatinienne Sir3 avec la queue de H4 et le recrutement du complexe SIR. Celui-ci en déacétylant les nucléosomes environnants entraîne la propagation de la structure hétérochromatinienne et la répression transcriptionnelle des gènes contenus dans la région euchromatique envahie (on parle de régulation par effet de position) [ 9,
25]. C. À l’inverse, la mutation de Sir2 (∆Sir2), comme celle de Sir3 (∆Sir3), bloque le processus auto-induit de recrutement du complexe SIR et de propagation de la structure hétérochromatinienne [ 9]. Par ailleurs, dans un contexte mutant Sas2 (B) le variant d’histone H2A.Z et Bdf1, une des sous-unités du complexe échangeur d’histone Swr1, ne sont plus détectés dans la portion euchromatinienne suggérant un rôle clé de l’acétylation de la lysine 16 dans le processus d’incorporation de ce variant. |
|
Lysine 16 et définition de régions chromatiniennes « ouvertes » L’un des processus les plus significatifs lié à l’acétylation de K16 est la spécification de domaines chromatiniens favorables à la transcription sur de vastes portions du génome, voire sur des chromosomes entiers. Ainsi, chez la levure, l’acétylation de K16 est essentielle à la détermination de la position de la « frontière » entre les gènes transcrits dans l’euchromatine et ceux épigénétiquement réprimés au sein de l’hétérochromatine [9, 25,
26]. En effet, un gradient d’acétylation de K16 contribue à déterminer la position de cette frontière dans les régions télomériques (Figure 1), des cassettes sexuelles (locus HML et HMR) et des gènes codants les ARN ribosomiques [26]. Chez la drosophile, des données génétiques et moléculaires ont élégamment démontré le rôle central de l’acétylation de K16 dans le processus de compensation de dose. Chez le mâle, la transcription des gènes du chromosome X est doublée de manière à compenser le fait que les femelles possèdent deux chromosomes X. Sur le plan moléculaire, chez le mâle, le complexe MSL (male-specific lethal), dont Mof est la sous-unité catalytique, est spécialement recruté de manière à acétyler K16 à travers le chromosome X entier [10,
21]. Cette modification contribuerait, notamment, à inhiber la compaction du chromosome médiée par le facteur de remodelage de la chromatine ISWI [21, 23]. |
Lysine 16, compaction de la fibre chromatinienne et structure du noyau Il y a quelques années, il a été établi que la portion de H4 comprenant les résidus basiques 14 à 19 était essentielle à l’interaction des nucléosomes entre eux et à la compaction de la fibre chromatinienne [
27]. L’acétylation de K16 étant la seule modification détectée dans cette région, on en a déduit de manière spéculative qu’elle devait jouer un rôle prépondérant dans le processus d’inhibition de la compaction de la chromatine d’autant qu’elle était essentielle au maintien décondensé de la chromatine sur de grandes portions de génome. Récemment, en utilisant une technique de ligation chimique permettant de produire des histones H4 acétylé de façon homogène sur K16, une preuve expérimentale a été obtenue [23]. L’incorporation de ces histones in vitro dans des nucléosomes a montré que l’acétylation de la K16 inhibe la compaction de la fibre ainsi reconstituée mais aussi l’interaction entre deux fibres différentes [23]. De fait, aujourd’hui, l’acétylation de K16 est la seule modification des histones dont on a démontré la capacité d’affecter, de manière directe, le processus de compaction de la fibre chromatinienne. Plus encore, la déplétion de MOF dans des cellules humaines affecte la structure globale du noyau avec l’apparition de multiples lobes nucléaires [5]. Ce phénotype peut être reproduit par la surexpression d’un variant de MOF déficient pour son activité HAT [5]. Ainsi l’acétylation de K16 serait essentielle au processus de régionalisation et de compaction des chromosomes et probablement aussi au maintien de la structure générale du noyau. |
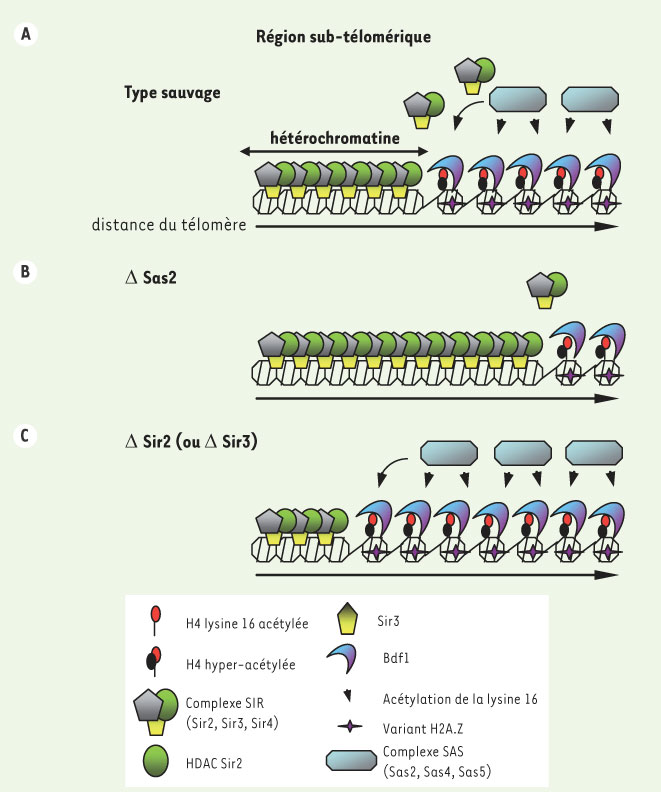
Lysine 16, cancer et croissance tumorale Comme elle intervient de manière déterminante dans de nombreux processus biologiques, l’altération de l’activité des HAT et HDAC est associée au dérèglement d’un nombre plus ou moins important de gènes ou de facteurs nucléaires pouvant jouer un rôle dans le processus de transformation et de cancérogenèse [2,
28]. Toutefois la preuve de l’implication directe de l’acétylation des histones dans ces processus n’a été acquise que récemment. Encore une fois K16 jouerait un rôle clé (Figure 2) [28–
30]. De manière remarquable, la perte sélective d’acétylation de K16 serait corrélée à la croissance tumorale d’au moins deux types de cancer : les adénocarcinomes du côlon et les hémopathies lymphocytaires [28]. L’acétylation de K16 aurait donc une fonction anti-transformation tumorale dans le noyau. Sur le plan moléculaire, dans diverses lignées cellulaires cancéreuses, l’association des HAT MOZ, MORF et MOF avec les régions d’ADN répétées péri-centromériques et sub-télomériques du génome (dont l’instabilité génétique est associée au cancer) est réduite voire abolie [28]. D’autre part, ces HAT avaient déjà été identifiées dans des réarrangements chromosomiques associés à des leucémies présentant une activité HAT K16 réduite [28]. À l’inverse de cette perte globale d’acétylation de K16, il a été montré que la ré-expression, au cours du processus tumoral, de gènes épigénétiquement réprimés coïncidait avec une augmentation de l’acétylation de K16 dans leurs régions promotrices bien que les marques épigénétiques de répression ne soient pas nécessairement éliminées [30,
31]. C’est pourquoi la détection de variations globales d’acétylation de K16 serait un des marqueurs de développement de certains cancers et constituerait même un moyen de prédire l’évolution clinique du cancer (par exemple, le cancer de la prostate [
29]). Le dosage de cette modification pourrait donc être à la base de systèmes de dépistage ou de diagnostic de plusieurs cancers.  | Figure 2.
Illustration de la perte globale et localisée d’acétylation de la lysine 16 dans des lignées cellulaires cancéreuses et certaines tumeurs humaines. A. Niveaux relatifs d’acétylation de la lysine 16 déterminés par HPCE ( high performance capillary electrophoresis) dans des biopsies de tumeurs primaires de côlon (barres vertes) et de tissus sains d’un même patient (barres grises). B. Quantification relative du niveau d’acétylation de la lysine 16 dans des cellules de côlon normales et HCT116 (lignée cellulaire d’adénocarcinome du côlon). C. Expérience similaire comparant des lymphocytes normaux et des lignées cellulaires de leucémies arborant les protéines chimères MOZ/CBP ou MORF/CBP. D. Expérience similaire comparant plusieurs lignées cellulaires d’un modèle de croissance tumorale de l’épiderme murin. On peut noter que la perte d’acétylation de la lysine 16 apparaît dès le début du processus tumoral et s’accentue tout au long de sa progression. Les lignées cellulaires PDV, PAM212, CarB et CarC sont classées dans un ordre croissant en fonction de leur propension à produire des tumeurs. E. Expérience de ChIP démontrant la perte localisée d’acétylation de la lysine 16 dans les régions d’ADN répétées péri-centromériques (Sat-2) et sub-télomériques (NBL-2 et D4Z4) du génome dans des lignées cellulaires cancéreuses (figure adaptée de données publiées dans [ 13]). |
|
Les récentes découvertes concernant le rôle de l’acétylation de K16 dans le processus de transcription, de compaction de la fibre chromatinienne et de croissance tumorale ont mis en lumière le rôle singulier de cette modification de la chromatine. Il apparaît désormais essentiel d’entreprendre l’étude fonctionnelle des HAT et HDAC, propres à K16, au cours d’autres processus nucléaires et cellulaires, de même qu’au cours du développement d’organismes eucaryotes supérieurs. Bien plus encore, la perspective de contrôler sélectivement le degré d’acétylation de K16 aussi bien dans des locus spécifiques que dans de vastes régions du génome laisse raisonnablement espérer l’émergence de nombreuses applications biotechnologiques et thérapeutiques. |
Les recherches du laboratoire du Dr Kevin Struhl sont subventionnées par le National Institutes of Health (NIH, États-Unis). Benoit Miotto est en stage post-doctoral après avoir réalisé une thèse en France, financièrement subventionnée par le ministère de la Recherche et des Technologies et l’Association pour la recherche contre le cancer, dans le laboratoire du Dr Jacques Pradel (CNRS, Laboratoire de génétique et physiologie du développement, Marseille). Cet article lui est d’ailleurs dédié en remerciement de son soutien et de ses multiples conseils.
|
1. Egger G, Liang G, Aparicio A, Jones PA. Epigenetics in human disease and prospects for epigenetic therapy. Nature 2004; 429 : 457–63. 2. Esteller M. Epigenetics provides a new generation of oncogenes and tumour-suppressor genes. Br J Cancer 2006; 94 : 179–83. 3. Jenuwein T, Allis CD. Translating the histone code. Science 2001; 293 : 1074–80. 4. Katan-Khaykovich Y, Struhl K. Dynamics of global histone acetylation and deacetylation in vivo: rapid restoration of normal histone acetylation status upon removal of activators and repressors. Genes Dev 2002; 16 : 743–52. 5. Taipale M, Rea S, Richter K, et al. hMOF histone acetyltransferase is required for histone H4 lysine 16 acetylation in mammalian cells. Mol Cell Biol 2005; 25 : 6798–810. 6. Dou Y, Milne TA, Tackett AJ, et al. Physical association and coordinate function of the H3 K4 methyltransferase MLL1 and the H4 K16 acetyltransferase MOF. Cell 2005; 121 : 873–85. 7. Smith ER, Cayrou C, Huang R, et al. A human protein complex homologous to the Drosophila MSL complex is responsible for the majority of histone H4 acetylation at lysine 16. Mol Cell Biol 2005; 25 : 9175–88. 8. Kimura A, Umehara T, Horikoshi M. Chromosomal gradient of histone acetylation established by Sas2p and Sir2p functions as a shield against gene silencing. Nat Genet 2002; 32 : 370–7. 9. Suka N, Luo K, Grunstein M. Sir2p and Sas2p opposingly regulate acetylation of yeast histone H4 lysine16 and spreading of heterochromatin. Nat Genet 2002; 32 : 378–83. 10. Akhtar A, Becker PB. Activation of transcription through histone H4 acetylation by MOF, an acetyltransferase essential for dosage compensation in Drosophila. Mol Cell 2000; 5 : 367–75. 11. Thorne AW, Kmiciek D, Mitchelson K, et al. Patterns of histone acetylation. Eur J Biochem/FEBS 1990; 193 : 701–13. 12. Turner BM, O’Neill LP, Allan IM. Histone H4 acetylation in human cells. Frequency of acetylation at different sites defined by immunolabeling with site-specific antibodies. FEBS Lett 1989; 253 : 141–5. 13. Zhang K, Williams KE, Huang L, et al. Histone acetylation and deacetylation: identification of acetylation and methylation sites of HeLa histone H4 by mass spectrometry. Mol Cell Proteomics 2002; 1 : 500–8. 14. Kurdistani SK, Tavazoie S, Grunstein M. Mapping global histone acetylation patterns to gene expression. Cell 2004; 117 : 721–33. 15. Dion MF, Altschuler SJ, Wu LF, Rando OJ. Genomic characterization reveals a simple histone H4 acetylation code. Proc Natl Acad Sci USA 2005; 102 : 5501–6. 16. Yoon HG, Choi Y, Cole PA, Wong J. Reading and function of a histone code involved in targeting corepressor complexes for repression. Mol Cell Biol 2005; 25 : 324–35. 17. Zhou Y, Grummt I. The PHD finger/bromodomain of NoRC interacts with acetylated histone H4K16 and is sufficient for rDNA silencing. Curr Biol 2005; 15 : 1434–8. 18. Jacobson RH, Ladurner AG, King DS, Tjian R. Structure and function of a human TAFII250 double bromodomain module. Science 2000; 288 : 1422–5. 19. Dhalluin C, Carlson JE, Zeng L, et al. Structure and ligand of a histone acetyltransferase bromodomain. Nature 1999; 399 : 491–6. 20. Clapier CR, Nightingale KP, Becker PB. A critical epitope for substrate recognition by the nucleosome remodeling ATPase ISWI. Nucleic Acids Res 2002; 30 : 649–55. 21. Corona DF, Clapier CR, Becker PB, Tamkun JW. Modulation of ISWI function by site-specific histone acetylation. EMBO Rep 2002; 3 : 242–7. 22. Makowski AM, Dutnall RN, Annunziato AT. Effects of acetylation of histone H4 at lysines 8 and 16 on activity of the Hat1 histone acetyltransferase. J Biol Chem 2001; 276 : 43499–502. 23. Shogren-Knaak M, Ishii H, Sun JM, et al. Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science 2006; 311 : 844–7. 24. Guccione E, Martinato F, Finocchiaro G, et al. Myc-binding-site recognition in the human genome is determined by chromatin context. Nat Cell Biol 2006; 8 : 764–70. 25. Shia WJ, Li B, Workman JL. SAS-mediated acetylation of histone H4 Lys 16 is required for H2A.Z incorporation at subtelomeric regions in Saccharomyces cerevisiae. Genes Dev 2006; 20 : 2507–12. 26. Meijsing SH, Ehrenhofer-Murray AE. The silencing complex SAS-I links histone acetylation to the assembly of repressed chromatin by CAF-I and Asf1 in Saccharomyces cerevisiae.
Genes Dev 2001; 15 : 3169–82. 27. Dorigo B, Schalch T, Bystricky K, Richmond TJ. Chromatin fiber folding: requirement for the histone H4 N-terminal tail. J Mol Biol 2003; 327 : 85–96. 28. Fraga MF, Ballestar E, Villar-Garea A, et al. Loss of acetylation at Lys16 and trimethylation at Lys20 of histone H4 is a common hallmark of human cancer. Nat Genet 2005; 37 : 391–400. 29. Seligson DB, Horvath S, Shi T, et al. Global histone modification patterns predict risk of prostate cancer recurrence. Nature 2005; 435 : 1262–6. 30. Pruitt K, Zinn RL, Ohm JE, et al. Inhibition of SIRT1 reactivates silenced cancer genes without loss of promoter DNA hypermethylation. PLoS Genet 2006; 2 : e40. 31. Ladurner AG, Inouye C, Jain R, Tjian R. Bromodomains mediate an acetyl-histone encoded antisilencing function at heterochromatin boundaries. Mol Cell 2003; 11 : 365–76. |