| |
| Med Sci (Paris). 2008 January; 24(1): 31–33. Published online 2008 January 15. doi: 10.1051/medsci/200824131.Dégradation protéasomique de Pax3 dans les cellules progénitrices musculaires Une ubiquitine fait l’affaire ! Stéphane C. Boutet1* and Thomas A. Rando1,2 1Department of Neurology and Neurological Sciences, Stanford University School of Medicine, Stanford, California 94305, États-Unis 2Neurology Service, Veterans Affairs Palo Alto Health Care System, Palo Alto, California 94304, États-Unis MeSH keywords: Humains, Modèles biologiques, Muscles squelettiques, Facteurs de transcription PAX, Proteasome endopeptidase complex, Ubiquitine |
Pax 3 et Pax 7, acteurs essentiels de la myogenèse Pax3 est un facteur de transcription essentiel à la myogenèse, processus de formation du muscle squelettique, pendant le développement embryonnaire [
1]. Pax3 permet également l’expression de c-Met, un récepteur à activité tyrosine kinase impliqué dans la délamination et la migration des cellules progénitrices à l’origine des muscles squelettiques des membres. Plus récemment, il a été montré que Pax3 et son homologue Pax7 sont exprimés par une population de précurseurs qui donne naissance aux cellules souches musculaires adultes positionnées sous la lame basale des myofibres et appelées cellules satellites [
2–
5]. Le rôle essentiel de Pax3 et Pax7 dans la spécification myogénique est aussi prouvé par l’observation que les cellules qui n’expriment plus Pax3 ou Pax7 meurent ou changent de lignage cellulaire [
3]. De plus, à l’âge adulte, alors que Pax7 continue à être exprimé dans les cellules satellites quiescentes, Pax3 ne semble présent que dans les cellules satellites de certains muscles [1]. Dans le muscle adulte, en réponse à une lésion, les cellules satellites quiescentes sont activées et deviennent des cellules progénitrices intermédiaires qui donnent naissance aux myoblastes qui, à leur tour, vont fusionner pour donner les nouvelles fibres musculaires [
6]. Lors de la myogenèse adulte, Pax3 est temporairement exprimé par la population de cellules progénitrices intermédiaires qui montre un fort potentiel prolifératif [
7] (Figure 1). Cette corrélation entre la présence de Pax3 et la forte capacité proliférative des cellules qui l’expriment caractérise les tumeurs comportant des caractéristiques musculaires (rhabdomyosarcomes) [
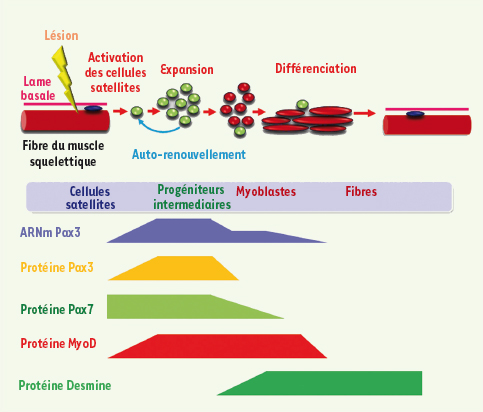
8]. Une abondante littérature suggère que la myogenèse embryonnaire et la myogenèse adulte dépendent de l’expression coordonnée, aussi bien temporelle que spatiale, de Pax3 et de Pax7. Alors qu’une attention considérable s’est portée sur l’étude de la régulation transcriptionnelle de Pax3 et de Pax7, aucune étude ne s’est intéressée à la régulation post-traductionnelle de ces facteurs de transcription.  | Figure 1.
Représentation schématique de la myogenèse dans le muscle adulte. Lorsqu’une fibre musculaire est lésée, les cellules souches du muscle squelettique (ou cellules satellites, en vert) résidant sous la lame basale sont activées. Après une phase de forte expansion caractérisée par l’expression de Pax3, les progéniteurs intermédiaires progressent dans le lignage myogénique en devenant des myoblastes (rouge). La différenciation myogénique terminale est caractérisée par la fusion des myoblastes en myotubes et l’expression des marqueurs terminaux de différenciation. Représentation schématique du niveau d’expression des marqueurs biochimiques associés aux différents stades d’activation et de la progression myogénique : Pax7 (protéine) dans les cellules satellites quiescentes et les progéniteurs intermédiaires, Pax3 (ARNm et protéine) et MyoD (protéine) dans les progéniteurs intermédiaires, Pax3 (ARNm) et MyoD (protéine) dans les myoblastes, et Desmine (protéine) dans les myoblastes et les myofibres différenciées. |
|
Contrôle des niveaux de Pax3 par le protéasome Lors de la myogenèse adulte, nous avons observé une divergence très marquée entre les niveaux d’ARN messager (ARNm) et de protéine Pax3 exprimés dans les cellules issues de la prolifération des progéniteurs intermédiaires (les myoblastes). Alors que le gène continue à être abondamment transcrit, la protéine n’est plus détectée, suggérant un mécanisme de régulation post-traductionnelle (Figure 1). En culture, la protéine Pax3 est détectée dans les cellules satellites activées mais seulement lorsque celles-ci sont préalablement traitées avec un inhibiteur du protéasome (MG132). De même, le traitement de cultures primaires de myoblastes avec MG132 empêche la dégradation de la protéine Pax3, alors que Pax7 est insensible à la dégradation par le protéasome [
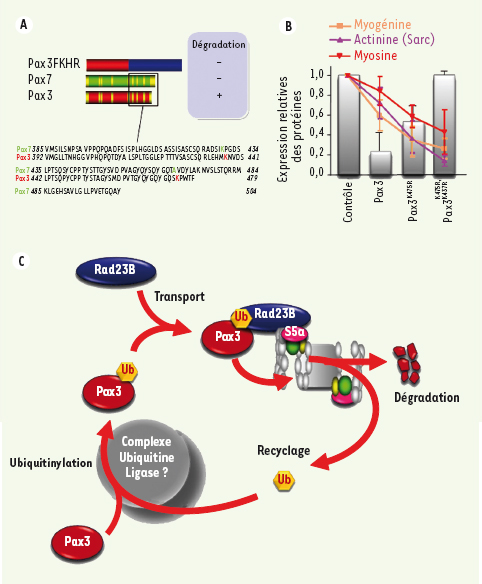
9]. De plus, l’utilisation d’une lignée cellulaire exprimant une ubiquitine ligase E1 dont l’activité est sensible à la température nous a permis de confirmer que la dégradation de Pax3 requiert un système ubiquitine/protéasome fonctionnel [9]. Pour étudier en détails les mécanismes moléculaires de la dégradation de Pax3, nous avons utilisé un système acellulaire couplant l’ubiquitinylation à la dégradation protéasomale. De manière surprenante, dans ce système, la protéolyse de Pax3 ne se produit que lorsque la protéine est mono-ubiquitinylée. Afin d’identifier les lysines ubiquitinylées entraînant la dégradation de Pax3 dans le protéasome, nous avons comparé la séquence et la stabilité des protéines Pax3, Pax7 et Pax3-FKHR, (la protéine fusion issue de la translocation chromosomique entre les gènes Pax3 et Forkhead). Nous avons identifié deux lysines susceptibles d’être ubiquitinylées, l’une spécifique de Pax3 (lysine 475), l’autre commune à Pax3 et Pax7 (lysine 437) (Figure 2A). Une mutation de la lysine 475 ou des deux lysines inhibe la dégradation protéasomale de Pax3 et augmente ainsi sa stabilité [9]. Pour démontrer que la lysine 475 propre à Pax3 suffit à expliquer la différence de stabilité entre Pax3 et Pax7, nous avons introduit dans Pax7 une lysine correspondant à la lysine 475 de Pax3 (Figure 2A). Cette protéine mutante est alors mono-ubiquitinylée et acquiert une sensibilité à la dégradation protéasomale [9]. La lysine 475 de Pax3 représente une signature de la différence de stabilité protéique entre Pax3 et Pax7. De plus, ces résultats suggèrent un mécanisme nouveau par lequel la mono-ubiquitinylation peut aussi être reconnue par le protéasome comme un signal protéolytique.  | Figure 2.
A. Représentation schématique des protéines Pax3-FKHR, Pax7 et Pax3. Seule Pax3 est sensible à la dégradation protéasomique. Les régions homologues entre Pax3 et Pax7 sont indiquées en jaune. L’analyse des régions carboxy-terminales de Pax3 et de Pax7 révèle la présence de deux lysines susceptibles d’être mono-ubiquitinylées : la lysine 475 spécifique à Pax3 et la lysine 437 commune à Pax3 et à Pax7. B. Quantification du niveau d’expression des marqueurs de la différenciation myogénique (lignes), comme la Myosine (chaîne lourde-en rouge), l’α-actinine sarcomérique (en violet) et la Myogénine (MyoG-en jaune) dans des myoblastes en cours de différenciation exprimant la GFP (témoin), Pax3, Pax3K475R ou Pax3K475R, K437R. Plus la protéine Pax3 est stable et plus l’expression des marqueurs de différenciation est inhibée. C. Modèle de la dégradation protéasomique de Pax3 par mono-ubiquitinylation. La protéine Pax3 est mono-ubiquitinylée par un mécanisme moléculaire resté indéterminé. Rad23B reconnaît spécifiquement la protéine Pax3 ubiquitinylée et transporte celle-ci jusqu’au protéasome en se liant à S5a. |
|
Reconnaissance de Pax3 ubiquitinylée En effet, une chaîne d’au moins quatre ubiquitines est généralement nécessaire pour que le protéasome reconnaisse son substrat. Les récepteurs de l’ubiquitine sont multiples et peuvent être soit parties intégrantes du complexe protéasomal (intrinsèques) comme la protéine S5a qui lie sélectivement les protéines poly-ubiquitinylées [
10], soit libres et indépendants du complexe protéasomal (extrinsèques) comme la protéine Rad23 [
11]. Nous avons montré qu’un complexe se forme entre les protéines Pax3 mono-ubiquitinylées, Rad23B et S5a, aussi bien in vitro que dans le contexte du protéasome fonctionnel [9]. En utilisant des cellules déficientes en Rad23A, Rad23B ou les deux, nous avons démontré que Rad23B est nécessaire à l’interaction entre Pax3 et S5a. De plus, Pax3 est dégradée par le protéasome uniquement dans les cellules exprimant Rad23B, suggérant que cette isoforme est essentielle pour le transport de Pax3 vers le protéasome, son interaction avec S5a, et finalement sa dégradation [9] (Figure 2C). Il est important de remarquer la spécificité de substrat entre les deux isoformes du récepteur de l’ubiquitine Rad23. En effet, Rad23 a une faible affinité pour l’ubiquitine monomérique [
12]. Mais la reconnaissance de Pax3 ubiquitinylée par Rad23B, et non par Rad23A, suggère que des interactions faibles entre Rad23B et l’ubiquitine d’une part, puis entre Rad23B et Pax3 d’autre part, permettent la reconnaissance et le positionnement de Pax3 mono-ubiquitinylée sur le protéasome. Dans la mesure où Pax3 peut inhiber la différenciation myogénique terminale [
13], nous avons fait l’hypothèse que la dégradation de Pax3 est une étape clé pour la progression du programme myogénique au-delà de l’étape d’engagement myogénique. En effet, l’expression de protéines Pax3 mutées dont la stabilité est accrue permet de maintenir les cellules sous forme mononucléée et d’empêcher l’expression des marqueurs tardifs de différenciation et la fusion en une fibre multinucléée [8] (Figure 2B). Ces résultats suggèrent que l’expression de Pax3 dans les cellules progénitrices musculaires permet de les maintenir dans un état non différencié et que la dégradation de Pax3 est essentielle à l’induction de la différenciation terminale. Cette étude nous a permis d’identifier un mécanisme par lequel la mono-ubiquitinylation d’une protéine peut être aussi reconnue comme un signal de dégradation par le protéasome (Figure 2C). Ce mécanisme de régulation de Pax3 permet de mieux comprendre les différences de profils d’expression entre Pax3 et Pax7 dans les cellules musculaires (Figure 1). Pour conclure, ces résultats apportent de nouvelles indications sur les mécanismes régulateurs de Pax3 dans les cellules souches du muscle et potentiellement sur la régulation des protéines Pax dans d’autres lignées de cellules souches. |
S.C.B. est soutenu par la Muscular Dystrophy Association. T.A.R. est soutenu par le NIH, le Department of Veterans Affairs, L’Ellison Medical Foundation et par le NIH Director’s Pioneer Award.
|
1. Buckingham M, Bajard L, Chang T, et al. The formation of skeletal muscle : from somite to limb. J Anat 2003; 202 : 59–68. 2. Gros J, Manceau M, Thome V, Marcelle C. A common somitic origin for embryonic muscle progenitors and satellite cells. Nature 2005; 435 : 954–8. 3. Manceau M, Marcelle C, Gros J. Une source unique de progéniteurs musculaires. Med Sci (Paris) 2005; 21 : 915–7. 4. Relaix F, Rocancourt D, Mansouri A, Buckingham M. A Pax3/Pax7-dependent population of skeletal muscle progenitor cells. Nature 2005; 435 : 948–53. 5. Lagha M, Rocancourt D, Relaix F. Origine du muscle squelettique : rôles de Pax3/Pax7. Med Sci
(Paris) 2005; 21 : 801–3. 6. Chazaud B, Chretien F, Gherardi RK. Les macrophages régulent les différentes phases de la régénération musculaire. Med Sci
(Paris) 2007; 23 : 794–5. 7. Conboy IM, Rando TA. The regulation of Notch signaling controls satellite cell activation and cell fate determination in postnatal myogenesis. Dev Cell 2002; 3 : 397–409. 8. Barr FG. Gene fusions involving PAX and FOX family members in alveolar rhabdomyosarcoma. Oncogene 2001; 20 : 5736–46. 9. Boutet SC, Disatnik MH, Chan LS, et al. Regulation of Pax3 by proteasomal degradation of mono-ubiquitinated protein in skeletal muscle progenitors. Cell 2007; 130 : 349–62. 10. Thrower JS, Hoffman L, Rechsteiner M, Pickart CM. Recognition of the polyubiquitin proteolytic signal. EMBO J 2000; 19 : 94–102. 11. Elsasser S, Finley D. Delivery of ubiquitinated substrates to protein-unfolding machines. Nat Cell Biol 2005; 7 : 742–9. 12. Raasi S, Orlov I, Fleming KG, Pickart CM. Binding of polyubiquitin chains to ubiquitin-associated (UBA) domains of HHR23A. J Mol Biol 2004; 341: 1367–79. 13. Epstein JA, Lam P, Jepeal L, et al. Pax3 inhibits myogenic differentiation of cultured myoblast cells. J Biol Chem 1995; 270 : 11719–22. |