| |
| Med Sci (Paris). 2008 October; 24(10): 847–852. Published online 2008 October 15. doi: 10.1051/medsci/20082410847.ADN immortel ou signature épigénétique ? Pierre Rocheteau and Shahragim Tajbakhsh* Cellules Souches et Développement, Département de Biologie du Développement, Institut Pasteur, CNRS URA 2578, 25, rue du Docteur Roux, 75724 Paris Cedex 15, France |
Les cellules souches sont présentes dans tous les tissus et organes. Elles sont indispensables pour assurer la croissance embryonnaire et post-natale, et sont également requises lors de la régénération tissulaire après une blessure. Le potentiel régénératif des cellules souches a suscité beaucoup d’intérêt en médecine ces dernières années. Toutefois, l’un des principaux handicaps à l’étude de ces cellules a été l’absence de marqueurs spécifiques permettant de discriminer un état cellulaire souche d’un état cellulaire engagé vers la différenciation. Pour cela, la rétention de thymidine tritiée ou d’autres analogues de la thymidine a été utilisée pour identifier et localiser les cellules souches putatives. La rétention du nucléotide radioactif est fondée sur l’hypothèse selon laquelle une cellule souche se diviserait peu ou très lentement, alors que les progéniteurs ou les cellules plus différenciées possèderaient un rythme de division plus rapide engendrant la dilution progressive du marqueur par ségrégation aléatoire des brins d’ADN marqué. Néanmoins, un nombre croissant d’expériences, dont certaines seront discutées dans cette revue, montrent que lors de sa division, la cellule souche pourrait transmettre les brins matrices de l’ADN, appelés « brins anciens ou immortels », à une seule des cellules filles [
1]. Ce mécanisme de co-ségrégation des brins matrices permettrait aux cellules souches de protéger leur ADN des erreurs produites lors de sa réplication. Cependant, ces observations pourraient également être expliquées par l’état épigénétique particulier de la cellule souche. |
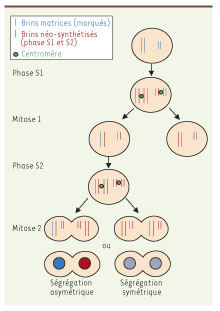
La co-ségrégation des brins matrices de l’ADN Dans l’hypothèse d’une réplication semi-conservative de l’ADN, les deux brins d’ADN des chromosomes sont identifiables, l’un étant défini comme « vieux brin » (ou matrice) et l’autre comme brin néo-synthétisé. L’hypothèse de ségrégation asymétrique suppose qu’il existe un mécanisme permettant la discrimination des brins anciens et néo-synthétisés, et la co-ségrégation des vieux brins de l’ADN à une seule des cellules filles lors de la division cellulaire (Figure 1). Le modèle prédit que la cellule héritant des brins anciens conservera son statut de cellule souche ; son génome serait ainsi protégé des erreurs réplicatives et des mutations.
 | Figure 1.
Modèle de division asymétrique du brin matrice de l’ADN. Lors d’une division asymétrique, les chromosomes contenant le brin matrice de l’ADN (bleu) co-ségrègent dans la cellule souche. La réplication étant semi- conservative, chaque chromosome contient un brin « ancien » et un brin néo-synthétisé. Ainsi les chromosomes contenant l’ancien brin d’ADN, en accord avec l’hypothèse du brin immortel, co-ségrègent pendant plusieurs divisions asymétriques, puis sont distribués de manière aléatoire lors de divisions symétriques. Une population de cellules souches contient donc proportionnellement plus d’anciens brins (bleu) que de brins néo-synthétisés (rouges) qui sont transmis à la population destinée à se différencier. |
Cette possible ségrégation asymétrique des brins d’ADN soulève plusieurs questions :
-
Ce phénomène est-il le fait de toutes les cellules souches ou seulement d’une partie d’entre elles ?
-
À quel stade de la vie cellulaire cela survient-il ? (stade embryonnaire, fœtal, ou adulte).
Les cellules souches peuvent aussi se diviser de manière symétrique. En effet, en période d’expansion cellulaire, comme c’est le cas au cours du développement, de la croissance, ou lors de la réparation d’une lésion, des divisons symétriques permettent d’augmenter de façon exponentielle la population cellulaire et de repeupler le tissu. En revanche, le mode de division asymétrique est souvent associé à un état homéostatique et à une expansion cellulaire linéaire.
-
Comment l’identité du brin matrice de l’ADN est-elle établie, et dans quelles circonstances s’effectue cette co-ségrégation ? Il faut souligner que pour répondre à ces questions, il est impératif de déterminer, pour chaque tissu et organe, la période à laquelle l’homéostasie est atteinte.
-
Quel mécanisme permet la co-ségrégation des brins matrices de l’ADN ? L’identité du brin matrice de l’ADN serait acquise lors des divisions cellulaires. Le modèle suppose qu’une fois cette identité acquise, elle serait reconnue et maintenue, au cours des divisions asymétriques. Il reste encore à déterminer si ce mécanisme est universel ou propre à certains tissus seulement, mais le démontrer, ou éventuellement le réfuter, est un vrai défi sur le plan expérimental. De même, il n’est pas exclu que ce phénomène ne soit pas restreint aux seules cellules souches, mais puisse, dans certaines conditions, s’appliquer également à d’autres types cellulaires.
|
Les implications pour la biologie des cellules souches La rétention des brins matrices de l’ADN dans une seule des cellules filles lors de la division cellulaire a des implications majeures. La première, bénéfique pour la cellule, est la conservation de la fidélité du génome puisque les éventuelles mutations survenant au cours des réplications seront distribuées dans les cellules qui vont se différencier et non pas dans les cellules souches [1]. Cela est important dans le contexte de la carcinogenèse, car ces cellules souches, contrairement aux cellules différenciées qui ont généralement une durée de vie limitée, sont destinées à perdurer dans l’organisme, et donneront naissance à de très nombreuses descendances au cours du temps. En contrepartie, cet « avantage » peut s’avérer néfaste pour une cellule souche qui subit une mutation pendant la phase d’expansion tissulaire, durant laquelle elle se multiplie alors via des divisions symétriques. Dans ce schéma, les éventuelles erreurs de réplication seront transmises systématiquement à leur descendance. L’accumulation de mutations dans le brin matrice compromettrait alors le fonctionnement et la viabilité de ces cellules souches, et expliquerait entre autres pourquoi la capacité de régénération des tissus diminue avec l’âge et l’incidence des cancers augmente. Finalement, ce modèle remet en question l’interprétation des expériences de « pulse-chase » où la rétention des molécules comme le 5-Bromo-2’-Déoxyuridine (BrdU), un analogue de la thymidine, par les cellules souches, était interprétée comme témoignant d’une sortie du cycle cellulaire. Or, selon l’hypothèse de co-ségrégation des brins matrices de l’ADN, une cellule souche pourrait se diviser tout en retenant le marqueur BrdU. |
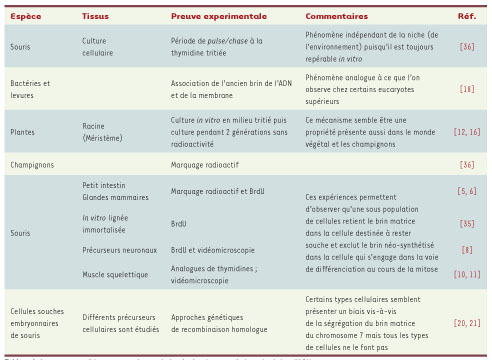
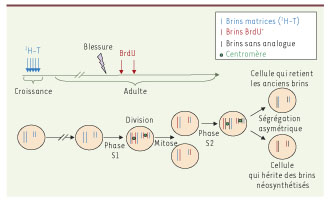
Preuves expérimentales de la ségrégation asymétrique des brins d’ADN Bien que postulée il y a une trentaine d’années, et en dépit des preuves expérimentales accumulées, l’existence de la co-ségrégation du brin matrice de l’ADN n’est pas universellement acceptée [
2,
3]. Cependant, de plus en plus de preuves expérimentales (Tableau I), obtenues notamment in vitro dans l’embryon de souris [
4], et dans l’intestin chez la souris [
5,
6], sont en accord avec ce modèle. Cette dernière étude, réalisée in vivo sur les cellules résidant dans les cryptes de l’intestin grêle, a eu un rôle majeur pour consolider l’hypothèse d’une ségrégation asymétrique des brins d’ADN. En effet, ces cellules se divisent un grand nombre de fois durant la vie de l’animal, assurant le renouvellement de la paroi épithéliale intestinale, et elles accumulent très peu de mutations cancéreuses. Dans ce travail, les souris ont reçu dans un premier temps une injection de thymidine tritiée (3H-thymidine) au cours de leur croissance (avant 6 semaines), marquant ainsi les brins de l’ADN. Puis à l’âge adulte, elles ont été exposées à une irradiation simulant une blessure de l’intestin et entraînant sa régénération, alors que dans le même temps, elles recevaient un pulse de BrdU (Figure 2). La présence de cellules marquées par les 2 analogues démontre que les cellules retenant la 3H-thymidine sont mitotiquement actives puisqu’elles ont incorporé le BrdU, et non pas dans un état quiescent. De plus, l’étude montrait qu’après un minimum de deux divisions (Figure 1), certaines cellules ont exclu le brin marqué par le BrdU (le brin néo-synthétisé) et ont conservé le brin marqué à la 3H-thymidine (brin matrice), en accord avec le modèle proposé de coségrégation des brins d’ADN immortels (Figure 2). Cependant une étude récente vient de montrer que, contrairement à ce que l’on pensait précédemment, les cellules souches de l’intestin grêle se trouvent au fond de la crypte intestinale et non pas quelques cellules au-dessus du fond, dans la position où les auteurs précédents ont fait leur étude [
7]. Cela ne remet pas en cause les observation d’une ségrégation asymétrique des brins d’ADN mais soulève la question du type cellulaire capable d’effectuer la co-ségrégation du brin matrice, qui peut ne pas être une cellule souche. Des études complémentaires réalisées in vivo et in vitro ont observé une ségrégation asymétrique des brins d’ADN dans d’autres types cellulaires comme les cellules neurales [
8], les cellules de la glande mammaire [
9] et les cellules satellites du muscle squelettique [
10,
11]. Chez les plantes Vicia faba ou Triticum boeoticum aussi, des biais ont été observés dans la distribution des chromosomes marqués au préalable par un traceur radioactif puis révélés par autoradiographie après un certain temps d’incubation dans un milieu de culture dépourvu de radioactivité [
12].
 | Tableau I.
Arguments expérimentaux en faveur de la ségrégation asymétrique des brins d’ADN.
|
 | Figure 2.
Schéma d’injection et observation du phénomène.
3H-T: thymidine tritiée en bleu correspondant ici au brin matrice marqué lors de la croissance de l’animal. BrdU : 5-Bromo-2’désoxyuridine en rouge correspondant ici au brin néo-synthétisé lors de l’activation de la cellule après une blessure et la synthèse de l’ADN. En noir correspond le brin néo-synthétisé en l’absence d’analogue. Dans ce cas de figure, la cellule souche va conserver les brins matrices marqués 3H-T alors que la cellule qui va entrer en différenciation va hériter des brins néo-synthétisés (BrdU). |
En revanche, dans d’autres systèmes, l’existence d’une co-ségrégation des brins matrices d’ADN n’a pas pu être démontrée (Tableau I). Ainsi, chez Caenorhabditis elegans, la règle est la ségrégation aléatoire, bien que le paradigme étudié correspondait au développement embryonnaire et non pas aux cellules souches adultes [
13]. Il semble aussi que l’absence de co-ségrégation des chromosomes prévaut dans les cellules souches du système hématopoïétique (CSH) [
14]. Cette étude bénéficie d’une connaissance approfondie des CSH ; cependant, il est vrai que les temps d’attente élevés par rapport au cycle cellulaire peuvent rendre les conclusions difficiles dans la mesure où des divisions symétriques peuvent avoir lieu, distribuant les brins matrices de l’ADN de façon aléatoire et diluant ainsi le marqueur, rendant la détection du phénomène impossible. Il est possible, donc, que les CSH ne ségrègent pas leur ADN de façon « immortelle ». Néanmoins, il est nécessaire de déterminer quand ces cellules souches s’engagent dans la division symétrique versus asymétrique pour consolider ces résultats. Ces analyses ne permettent donc pas d’affirmer que le processus de la co-ségrégation des brins matrices est impossible mais seulement qu’il est absent dans certains lignages cellulaires dans les conditions expérimentales utilisées. Cela indique que la ségrégation non-aléatoire des brins d’ADN n’est pas une règle absolue [
15–
17]. Ces conclusions sont en accord avec le modèle que nous proposons, dans lequel les cellules souches alternent entre des phases de divisions symétriques et de divisions asymétriques (sinon ces cellules, excluant toujours le brin néo-synthétisé, ne pourraient jamais incorporer un marqueur de l’ADN). Il est important de noter que, dans aucune de ces études, le phénomène de « l’immortalité » des brins d’ADN n’a été directement démontré sur de longues périodes, bien que de fortes preuves expérimentales utilisant différentes approches ont permis de documenter la co-ségrégation des brins matrices de l’ADN de manière indirecte. D’ailleurs, le choix du terme « immortel » initialement proposé par Cairns peut s’avérer inapproprié. En effet, la co-ségrégation des brins matrices n’implique pas que ce phénomène soit utilisé continuellement par la même cellule au cours de toute sa vie, mais plutôt définit un état qui peut être transitoire. Ce comportement cellulaire pourrait donc être plus complexe que celui qui a été proposé lors de sa description initiale ; la co-ségrégation du brin parental pourrait aussi être un phénomène stochastique rendant sa mise en évidence expérimentale très complexe sur de longues périodes. |
Considérations mécanistiques Indépendamment de la ségrégation des brins d’ADN, les cellules souches sont capables de se diviser de façon asymétrique ou symétrique, distribuant à parts égales ou inégales des déterminants cellulaires différents aux deux cellules filles à l’issue de la mitose. La levure S. pombe est un modèle de référence dans l’étude du mécanisme de division cellulaire. Cet eucaryote peut générer des descendants de sexe différents (P et M) via une division cellulaire asymétrique. Ce processus de division met en jeu des réarrangements géniques. L’expression des gènes responsables de ce phénomène est sous contrôle épigénétique. Or ces marques épigénétiques (au niveau du locus mat1) se trouvent sur un seul des deux brins de l’ADN [
18]. Il s’agit d’un cas de figure analogue à celui de la division asymétrique d’une cellule souche où une cellule fille est toujours identique à la cellule parentale alors que l’autre s’engage dans la différenciation et où les deux brins de l’ADN ne sont pas identiques. Les différences de destins cellulaires sont corrélées à la non équivalence des brins d’ADN. Cette division asymétrique est effectuée en partie par une machinerie cellulaire qui contrôle l’orientation du fuseau mitotique [
19]. La ségrégation active des chromosomes est probablement sous le contrôle de cette machinerie qui est composée d’éléments appartenant au cytosquelette. Un rôle clé de la dynéine a été proposé dans le mécanisme de co-ségrégation du brin de l’ADN dans les cellules, car l’utilisation d’ARN interférants qui bloquent l’expression de la dynéine abolit le phénomène [
20,
21]. La dynéine fait partie du complexe qui reconnaît les kinétochores, structures d’attachement des microtubules et des chromosomes durant la métaphase [
37]. Cela pourrait d’ailleurs constituer un autre « checkpoint » et expliquer la longueur de la métaphase par rapport à l’anaphase qui se déroule beaucoup plus rapidement, le temps supplémentaire étant utilisé pour la reconnaissance éventuelle des brins matrices. Les kinétochores sont en tout cas indispensables à la migration active des chromosomes aux pôles de la cellule [
22–
26]. Des défauts dans ces structures entraînent la ségrégation incorrecte des chromosomes pouvant contribuer à la formation de tumeurs ou à l’apparition de cellules aneuploïdes [
27,
28]. Le centromère (la région du chromosome où les chromatides sœurs sont liées et où se situent les kinétochores [
38]) est contrôlé par des marques épigénétiques pouvant être distinctes sur les deux chromatides du même chromosome [
29], qui pourront chacune transmettre des états épigénétiques différents dans les cellules filles après la division. Ces marques épigénétiques contrôlent la région péricentromérique composée d’un domaine hétérochromatique. Il est à noter que la formation de l’état hétérochromatique de ce domaine est régulé par ARN interférence chez S. pombe [
30] et qu’il affecte la ségrégation des chromosomes [
31,
32]. Des mécanismes similaires ont été observés chez le poulet [
33] et la souris [
34]. Des marques épigénétiques propres aux centrosomes pourraient être mises en place lors de l’acquisition de l’état « matrice » de l’ADN, et être conservées lors des divisions asymétriques. Par ailleurs, il a été proposé que p53, un facteur clé pour le maintien de l’intégrité du génome, soit un modulateur du phénomène de l’ADN « immortel » [
35]. Cependant, des expériences à court terme in vitro ont montré que le phénomène de co-ségrégation du brin matrice de l’ADN peut se produire en l’absence de p53 dans les cellules musculaires [11]. Il n’est néanmoins pas exclu que d’autres membres de cette famille, notamment p63 et p73, régulent ce phénomène. Ainsi le cytosquelette doit jouer un rôle clé dans la reconnaissance et la coségrégation des brins matrices des chromosomes. Les deux chromatides sœurs comportant la même information génétique, la reconnaissance doit se faire par le biais de marques épigénétiques. |
Régulation épigénétique des cellules souches et des progéniteurs : un modèle alternatif La régulation épigénétique correspond à des modifications transmissibles et réversibles de l’expression des gènes qui ne s’accompagnent pas de changements au niveau de la séquence nucléotidique [
39,
40]. Il est fort probable que l’identité des chromatides soit définie par le biais de marques épigénétiques. Quand l’hypothèse de co-ségrégation des brins matrices de l’ADN a été proposée il y a plus de 30 ans, la notion de régulation épigénétique du génome et sa corrélation avec l’état cellulaire n’étaient pas encore formulées. Aujourd’hui, de très nombreuses preuves expérimentales démontrent que l’état chromatinien et notamment l’état de méthylation de l’ADN ou des modifications des histones, jouent un rôle déterminant dans la régulation de l’expression de gènes. Ainsi, l’activation ou la répression des gènes par le biais de ces marques épigénétiques permet de distinguer un état souche d’un état plus différencié des cellules. Ces signatures moléculaires pourraient permettre aux brins matrices d’être reconnus et d’être co-ségrégés. Il est important de noter que la ségrégation asymétrique des brins matrices pourrait refléter des marques épigénétiques de l’ensemble des gènes des chromatides et ne soit pas simplement un moyen d’échapper aux erreurs de réplication comme cela a été postulé par Cairns [1]. De cette façon, l’expression des gènes impliqués dans l’auto-renouvellement et le destin cellulaire pourrait être respectivement maintenue ou réprimée dans les cellules souches et leurs descendants. L’utilisation de marqueurs comme des analogues de nucléotides pourrait dévoiler, par la co-ségrégation du brin matrice de l’ADN, la distribution inégale de ces signatures épigénétiques. Nous avions évoqué cette possibilité [11], qui a été récemment élaborée plus en détail [3]. Ainsi, les cellules filles qui conservent le brin matrice conservent aussi l’identité souche. Et par conséquent, les cellules s’engageant dans la voie de différenciation héritent des brins « néo-synthétisés ». La différence de comportement cellulaire entre les deux cellules filles peut provenir de la non-équivalence des brins de l’ADN, qui fournissent aux deux cellules filles la même information génétique mais des états épigénétiques différents se traduisant par un état d’activation des gènes différent entre les deux cellules. |
Conclusions et perspectives Le fait que les brins matrices de l’ADN co-ségrègent au cours de la division cellulaire intrigue les chercheurs depuis trente ans. Les progrès de nos connaissances en biologie cellulaire mais aussi des régulations épigénétiques, de même que le développement de méthodes de marquage in vitro et in vivo sophistiquées, ont permis d’apporter de nouveaux éléments. Il demeure cependant à répondre à des questions majeures, par exemple, ce processus est-il exclusif des cellules souches adultes ? Quel que soit le bénéfice que puisse tirer la cellule souche d’un tel mécanisme, le fait que les chromatides sœurs puissent être distinguées pendant la métaphase et ségrégées sélectivement dans l’une des cellules filles, ébranle un dogme et repousse les limites de la biologie cellulaire, que l’imagerie dynamique devrait dans les prochaines années aider à résoudre. |
Nous remercions les membres du laboratoire pour leurs commentaires sur le manuscrit. Le laboratoire est subventionné par l’AFM, l’ARC, la FRM, le CNRS et l’Institut Pasteur.
|
1. Cairns J. Mutation selection and the natural history of cancer. Nature 1975; 255 : 197–200. 2. Kuroki T, Murakami Y. Random segregation of DNA strands in epidermal basal cells. Jpn J Cancer Res 1989; 80 : 637–42. 3. Lansdorp PM. Immortal strands ? Give me a break. Cell 2007; 129 : 1244–7. 4. Lark KG, Consigli RA, Minocha HC. Segregation of sister chromatids in mammalian cells. Science 1966; 154 : 1202–5. 5. Potten CS, Hume WJ, Reid P, Cairns J. The segregation of DNA in epithelial stem cells. Cell 1978; 15 : 899–906. 6. Potten CS, Owen G, Booth D. Intestinal stem cells protect their genome by selective segregation of template DNA strands. J Cell Sci 2002; 115 : 2381–8. 7. Barker N, van Es JH, Kuipers J, et al. Identification of stem cells in small intestine and colon by marker gene lgr5. Nature 2007; 449 : 1003–7. 8. Karpowicz P, Morshead C, Kam A, et al. Support for the immortal strand hypothesis: Neural stem cells partition DNA asymmetrically in vitro. J Cell Biol 2005; 170 : 721–32. 9. Smith GH. Label-retaining epithelial cells in mouse mammary gland divide asymmetrically and retain their template DNA strands. Development 2005; 132 : 681–7. 10. Conboy MJ, Karasov AO, Rando TA. High incidence of non-random template strand segregation and asymmetric fate determination in dividing stem cells and their progeny. PLoS Biol 2007; 5 : e102. 11. Shinin V, Gayraud-Morel B, Gomes D, Tajbakhsh S. Asymmetric division and cosegregation of template DNA strands in adult muscle satellite cells. Nat Cell Biol 2006; 8 : 677–82. 12. Lark KG. Nonrandom segregation of sister chromatids in vicia faba and triticum boeoticum. Proc Natl Acad Sci USA 1967; 58 : 352–9. 13. Ito K, McGhee JD. Parental DNA strands segregate randomly during embryonic development of Caenorhabditis elegans. Cell 1987; 49 : 329–36. 14. Kiel MJ, He S, Ashkenazi R, et al. Haematopoietic stem cells do not asymmetrically segregate chromosomes or retain brdu. Nature 2007; 449 : 238–42. 15. Comings DE. The distribution of sister chromatids at mitosis in chinese hamster cells. Chromosoma 1970; 29 : 428–33. 16. Fernandez-Gomez ME, de la Torre C, Stockert JC. Random segregation of sister chromatids in meristematic cells. Exp Cell Res 1975; 96 : 156–60. 17. Morris VB. Random segregation of sister chromatids in developing chick retinal cells demonstrated in vivo using the fluorescence plus giemsa technique. Chromosoma 1977; 60 : 139–45. 18. Klar AJ. Lessons learned from studies of fission yeast mating-type switching and silencing. Annu Rev Genet 2007; 41 : 213–36. 19. Yamashita YM, Jones DL, Fuller MT. Orientation of asymmetric stem cell division by the apc tumor suppressor and centrosome. Science 2003; 301 : 1547–50. 20. Armakolas A, Klar AJ. Cell type regulates selective segregation of mouse chromosome 7 DNA strands in mitosis. Science 2006; 311 : 1146–9. 21. Armakolas A, Klar AJ. Left-right dynein motor implicated in selective chromatid segregation in mouse cells. Science 2007; 315 : 100–1. 22. Holleran EA, Karki S, Holzbaur EL. The role of the dynactin complex in intracellular motility. Int Rev Cytol 1998; 182 : 69–109. 23. Pfarr CM, Coue M, Grissom PM, et al. Cytoplasmic dynein is localized to kinetochores during mitosis. Nature 1990; 345 : 263–5. 24. Starr DA, Williams BC, Hays TS, Goldberg ML. Zw10 helps recruit dynactin and dynein to the kinetochore. J Cell Biol 1998; 142 : 763–74. 25. Steuer ER, Wordeman L, Schroer TA, Sheetz MP. Localization of cytoplasmic dynein to mitotic spindles and kinetochores. Nature 1990; 345 : 266–8. 26. Vaisberg EA, Koonce MP, McIntosh JR. Cytoplasmic dynein plays a role in mammalian mitotic spindle formation. J Cell Biol 1993; 123 : 849–58. 27. Jacobs CW, Adams AE, Szaniszlo PJ, Pringle JR. Functions of microtubules in the Saccharomyces cerevisiae cell cycle. J Cell Biol 1988; 107 : 1409–26. 28. Saunders WS, Shuster M, Huang X, et al. Chromosomal instability and cytoskeletal defects in oral cancer cells. Proc Natl Acad Sci
USA 2000; 97 : 303–8. 29. Ekwall K. Epigenetic control of centromere behavior. Annu Rev Genet 2007; 41 : 63–81. 30. Volpe TA, Kidner C, Hall IM, et al. Regulation of heterochromatic silencing and histone h3 lysine-9 methylation by rnai. Science 2002; 297 : 1833–7. 31. Provost P, Silverstein RA, Dishart D, et al. Dicer is required for chromosome segregation and gene silencing in fission yeast cells. Proc Natl Acad Sci USA 2002; 99 : 16648–53. 32. Volpe T, Schramke V, Hamilton GL, et al. RNA interference is required for normal centromere function in fission yeast. Chromosome Res 2003; 11 : 137–46. 33. Fukagawa T. Assembly of kinetochores in vertebrate cells. Exp Cell Res 2004; 296 : 21–7. 34. Kanellopoulou C, Muljo SA, Kung AL, et al. Dicer-deficient mouse embryonic stem cells are defective in differentiation and centromeric silencing. Genes Dev 2005; 19 : 489–501. 35. Rambhatla L, Bohn SA, Stadler PB, et al. Cellular senescence: ex vivo p53-dependent asymmetric cell kinetics. J Biomed Biotechnol 2001; 1 : 28–37. 36. Rosenberger RF, Kessel. Nonrandom sister chromatid segregation and nuclear migration in hyphae of Aspergillus nidulans. J Bacteriol 1968; 1 : 1208–13. 37. Castro A, Vigneron S, Lorca T, Labbé JC. La mitose sous surveillance. Med Sci (Paris) 2003; 19 : 309–17. 38. Labbé JP. Élasticité du centromère. Med Sci (Paris) 2005; 21 : 261–6. 39>. Ray-Gallet D, Gérard A, Polo S, Almouzni G. Variations sur le thème, du code histone. Med Sci (Paris) 2005; 21 : 384–9. 40. Képès F. L’épigénétique comme aspect de la postgénomique. Med Sci (Paris) 2005; 21 : 371–6. |