| |
| Med Sci (Paris). 2009 May; 25(5): 505–512. Published online 2009 May 15. doi: 10.1051/medsci/2009255505.La bataille du silence Mécanisme et inhibition du RNA silencing au cours des interactions plante/virus Patrice Dunoyer* Institut de Biologie Moléculaire des Plantes du CNRS, IBMP-CNRS, Université de Strasbourg, 12, rue du Général Zimmer, 67084 Strasbourg Cedex, France |
Découvert au début des années 1990 [
1,
2], le mécanisme du « RNA silencing » a complètement révolutionné notre façon d’appréhender la régulation de l’expression du génome chez les eucaryotes. Le terme « RNA silencing » (que nous préférons garder dans son appellation anglophone) englobe, de manière générale, l’ensemble des mécanismes conduisant à la suppression de l’expression de gènes, au niveau transcriptionnel ou post-transcriptionnel, via des interactions séquence-spécifiques médiées par l’ARN. Présent de l’algue unicellulaire Chlamydomonas reinhartii à l’homme, et hautement conservé chez la quasi-totalité des organismes eucaryotes, le RNA silencing joue un rôle essentiel dans un grand nombre de processus biologiques, par exemple le maintien de la stabilité des génomes (en réprimant le mouvement des éléments mobiles du génome que sont les transposons) ou le contrôle de l’expression de gènes au cours du développement, nécessaire à leur bon déroulement et inversement participe à de nombreuses pathologies comme le cancer (→) [
3,
4,
61]. (→) Voir l’article de Yannick Ladeiro et Jessica Zucman-Rossi, page 467 de ce numéro
Dans tous les cas, l’ARN double brin (ARNdb) est la molécule clé initiatrice du silencing [
5]. L’ARNdb peut être produit in vivo soit par transcription de séquences génomiques organisées en répétitions inversées, à partir de promoteurs convergents ou via l’action d’ARN polymérases ARN-dépendantes (RdRp) d’origine endogène ou virale qui convertiront un ARN simple brin (ARNsb) en ARNdb. Cet ARNdb est reconnu par une enzyme de type RNase III appelée Dicer [
6] qui va produire la seconde catégorie de molécules ubiquitaires du RNA silencing que sont les petits ARN (small ARN ou sARN) [
7,
8] (Figure 1). Ces sARN, de 21-24nt de longueur, vont ensuite être incorporés dans un complexe multiprotéique appelé RISC (RNA-induced silencing complex) où ils serviront de guide pour lui permettre de retrouver, de manière séquence-spécifique, tout espèce d’acides nucléiques présentant une séquence complémentaire [
9]. Selon la nature du complexe RISC impliqué, cette reconnaissance entraînera une répression de l’expression du gène, soit post-transcriptionnelle, par clivage endonucléolytique ou inhibition de la traduction de l’ARN messager (ARNm) ciblé [
10], soit transcriptionnelle, par méthylation de l’ADN ou de la chromatine [
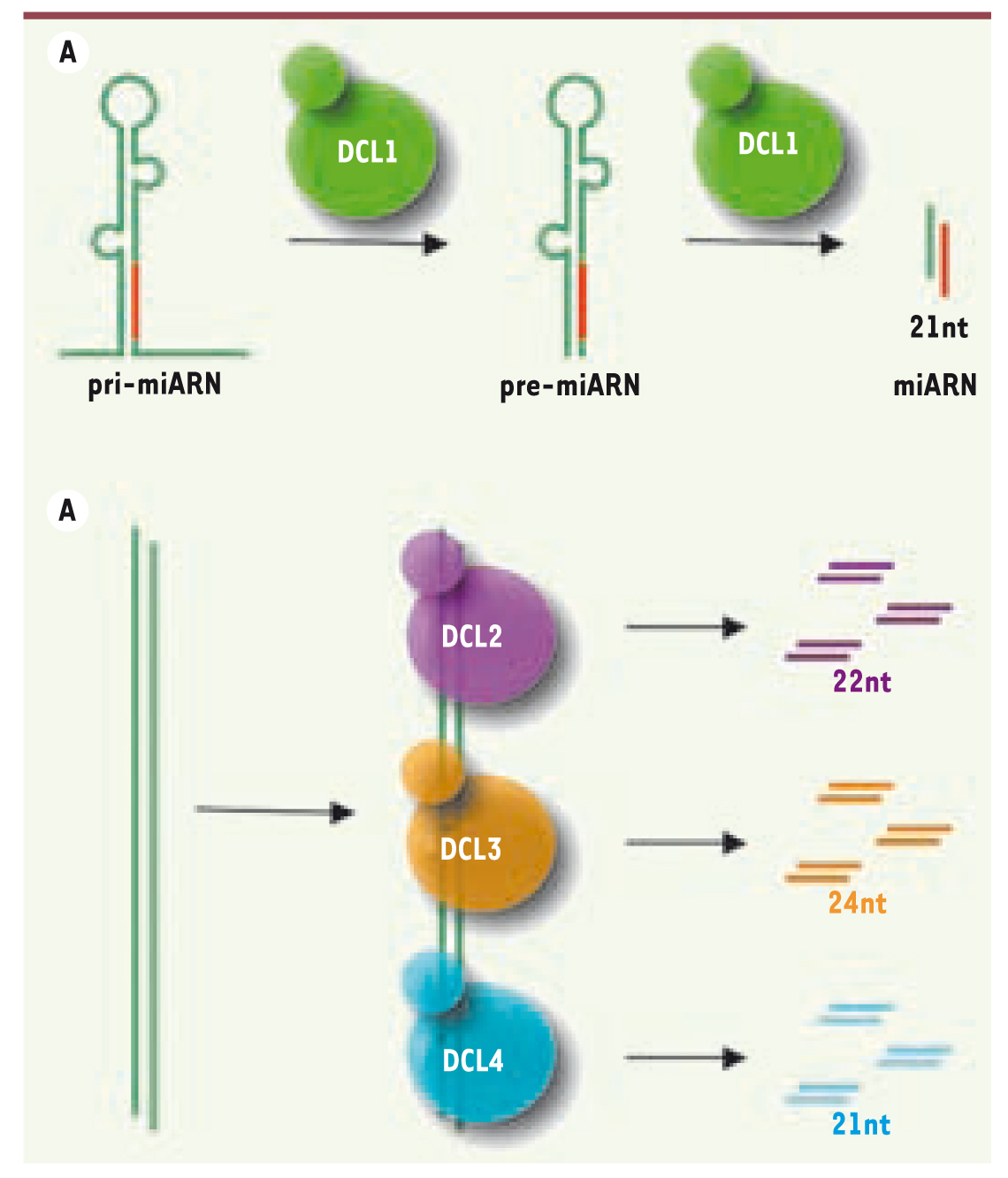
11].  | Figure 1.
Biogenèse des miARN et siARN chez les plantes. A. Biogenèse des miARN. Les transcrits primaires des miARN (pri-miARN) sont convertis séquentiellement en précurseurs de miARN (pre-miARN) puis en duplexes de miARN. Ces deux étapes sont catalysées par l’activité de DCL1 dans le noyau des cellules végétales. B. Biogenèse des siARN. Les précurseurs des siARN sont de longs ARN double brin qui seront reconnus par DCL2, DCL3 ou DCL4, enzymes qui produiront des siARN de 22nt, 24nt ou 21nt, respectivement. |
|
RNA silencing et protection antivirale Chez les plantes et les invertébrés, plusieurs études montrent qu’un des rôles biologiques du RNA silencing est la défense antivirale [
12–
14]. Cette implication a été confirmée de manière non ambiguë par la détection systématique de quantités importantes de sARN dérivant du génome viral (virus-derived sRNA ou vsARN), dans des tissus de plantes infectées par des virus très variés. En effet, la plupart des virus infectant ces organismes possèdent un génome à ARN qui est répliqué dans la cellule hôte sous forme d’un intermédiaire constitué d’ARNdb. De plus, la plupart des ARN viraux possèdent des structures intramoléculaires sous forme d’ARNdb présentant un haut degré de complémentarité (Figure 2A). Ce sont ces deux formes d’ARNdb qui sont responsables de l’activation de la machinerie cellulaire du RNA silencing [
15,
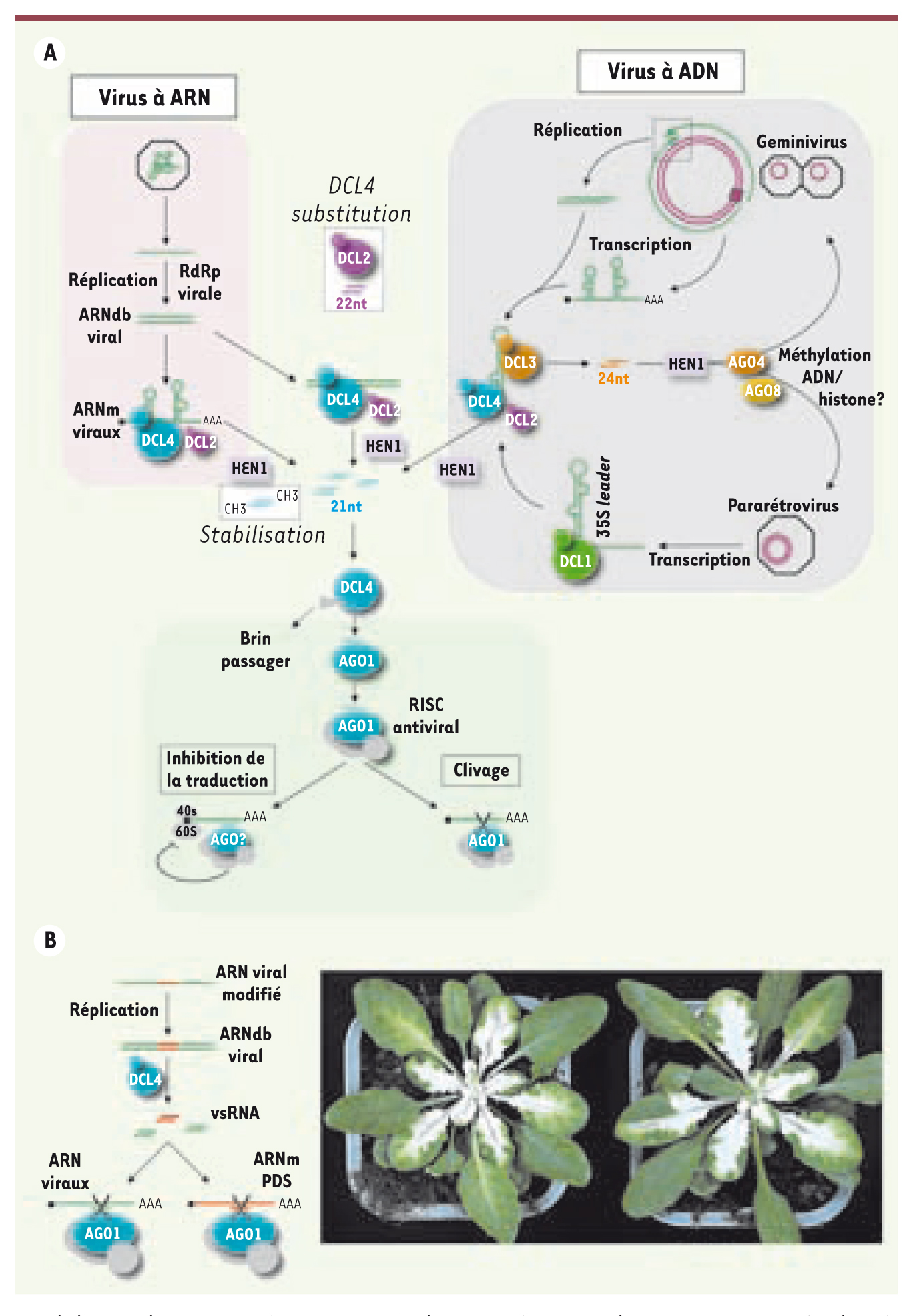
16] conduisant à la dégradation séquence-spécifique de tout ARN homologue à l’ARN viral initiateur (Figure 2B). 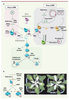 | Figure 2.
A. Mode d’action du RNA silencing dans la défense antivirale. Dans le cas des virus à ARN, DCL4 est l’enzyme Dicer principale de la réponse antivirale et DCL2 celle de substitution quand DCL4 n’est pas fonctionnel. Pour les virus à ADN, DCL1 facilite l’accès de DCL4 et DCL3 (les deux Dicer antiviraux principaux) à l’ARN double brin viral. DCL2 agit également de manière redondante avec DCL4 pour ces virus à ADN. Les vsARN produits sont tous stabilisés par l’ajout d’un groupement méthyl à leur extrémité 3’ catalysé par la protéine HEN1. Les deux brins du duplexe de vsARN sont ensuite séparés et seul l’un des deux brins est incorporé dans le RISC antiviral, l’autre brin étant dégradé. AGO1 est ici représentée comme l’Argonaute principale intervenant dans la réponse antiantivirale mais d’autres protéines AGO sont très probablement impliquées. Le complexe RISC va ensuite reconnaître les ARN messagers viraux complémentaires au brin guide incorporé ainsi que tout transcrit cellulaire possédant une homologie de séquence avec l’ARN viral initiateur. Cette reconnaissance entraînera le clivage ou l’inhibition de la traduction des messagers ciblés. Dans le cas des virus à ADN, les vsARN de 24nt produits par DCL3 peuvent également inhiber la réplication de l’ADN génomique viral en étant incorporés dans AGO4 et/ou AGO6, induisant un silencing transcriptionnel par méthylation de l’ADN ou des histones présents sur le génome viral qui se réplique dans le noyau sous forme de mini-chromosomes. B. Illustration du phénomène de virus-induced silencing (VIGS). Des plantes d’Arabidospsis ont été infectées avec un phytovirus modifié portant dans son génome un fragment du gène de la phytoene desaturase (PDS). La réplication du virus va activer le processus de RNA silencing antiviral et les vsARN produits, une fois incorporés dans le complexe RISC, assureront le clivage des transcrits viraux mais aussi celui de l’ARN messager PDS, conduisant à l’apparition d’un phénotype de blanchissement des feuilles là où le virus est présent. On peut ainsi imaginer qu’une partie des symptômes observés au cours d’une infection naturelle est potentiellement due au silencing de transcrits cellulaires par les vsARN dérivant du virus. |
Dans le cas des virus à ADN, les vsARN peuvent être produits par 2 mécanismes distincts selon la nature du génome viral. Pour les virus à ADN simple brin tels que les geminivirus, il a été suggéré que les vsARN sont produits à partir d’ARNdb formés par l’appariement intermoléculaire entre les transcrits sens et antisens produits durant la transcription de leur génome circulaire [
17]. Dans le cas des virus à ADN double brin, tels que le Cauliflower mosaic pararetrovirus (CaMV), l’extrémité 5’ du transcrit polycistronique, appelée la région « 35S leader » représente la source majoritaire de vsARN en raison de sa structure secondaire extensive qui sert de principal substrat pour Dicer [
18] (Figure 2A).
Ainsi, le RNA silencing peut être défini comme un système immunitaire dont un aspect fondamental est qu’il est totalement inné car la réponse est uniquement programmée par les caractéristiques et la séquence du génome infectieux et non par le génome de l’hôte lui-même. |
Base moléculaire du RNA silencing chez les plantes Chez les plantes et les animaux, on distingue 2 catégories de sARN selon leur biogenèse : les microARN (miARN) et les petits ARN interférents (siARN). Les miARN dérivent d’ARNsb précurseurs (pre-miARN) capables de se replier en une structure tige-boucle par appariement intramoléculaire. Les siARN dérivent de longues molécules d’ARNdb présentant un haut degré de complémentarité. Dans le génome d’Arabidopsis, on trouve 4 homologues de Dicer (Dicer-like ou DCL). DCL1 est majoritairement responsable de la maturation des pre-miARN et de la production des miARN matures [3–4,
19–
20]. DCL4, DCL2 et DCL3 sont quant à elles responsables de la production des siARN de 21nt, 22nt et 24nt, respectivement, à partir des longs ARNdb [
21] (Figure 1).
Les 2 brins du duplex des sARN vont ensuite être séparés et seul l’un des 2 brins (le guide) sera incorporé dans le complexe RISC, le brin complémentaire (le passager) sera quant à lui dégradé (Figure 2A). Les protéines de la famille Argonaute (AGO, dont il existe 10 membres chez Arabidopsis) jouent un rôle crucial dans le fonctionnement du complexe RISC. Ces protéines possèdent au moins un domaine PAZ (par référence aux protéines Piwi, Argonaute et Zwille) de fixation des ARNsb et un domaine PIWI conférant l’activité de clivage endonucléolytique (qui n’a pour l’instant été démontrée que pour AGO1, AGO4 et AGO7). Chez Arabidopsis, les miARN sont préférentiellement chargés dans AGO1 qui assure le clivage ou l’inhibition de traduction des transcrits ciblés [
22–
24]. Les siARN de 24nt sont, eux, chargés dans AGO4 ou AGO6 et sont responsables de la répression de la transcription par méthylation des histones et/ou de l’ADN au niveau des séquences promotrices [
25,
26]. De plus, au cours de leur synthèse, une méthyltransférase appelée HEN1 protège toutes les classes de sARN d’une dégradation précoce par l’ajout d’un groupement méthyl à leur extrémité 3’ [
27] (Figure 2A). |
Rôle des Dicer dans la défense antivirale ou comment transformer votre ennemi en milliers de petites sentinelles Le fait qu’aucun des simples mutants dcl chez Arabidopsis ne présentait une susceptibilité accrue à une infection virale suggérait fortement l’existence d’une redondance fonctionnelle au sein des DCL de plantes dans l’immunité antivirale. En accord avec cette hypothèse, une hypersusceptibilité aux virus à ARN n’a pu être observée que sur des plantes chez lesquelles on avait simultanément inactivé DCL4 et DCL2 [
28,
29]. DCL4 est le Dicer antiviral principal et produit les vsARN de 21nt qui s’accumulent majoritairement au cours de l’infection d’une plante sauvage. Cependant, en réponse à l’inactivation de DCL4, DCL2 prend en charge le silencing antiviral en produisant des vsARN de 22nt qui sont normalement sous le seuil de détection, indiquant son rôle de substitution dans la défense antivirale (Figure 2A). Par opposition, aucune contribution significative de DCL1 et DCL3 n’a pu être observée dans l’immunité contre ce type de virus. En effet, d’une part on a pu mettre en évidence que la quantité de vsARN produite par DCL1 est quasiment négligeable et d’autre part, les vsARN de 24nt produits par DCL3 ne sont pas capables d’induire la dégradation de transcrits homologues, vraisemblablement parce qu’ils guident normalement le silencing transcriptionnel au niveau de l’ADN [29]. De manière intéressante, l’implication des Dicer dans la protection antivirale change dans le cas des virus à ADN où il semble que les 4 Dicer agissent coopérativement pour assurer la défense antivirale [18,
30]. Ainsi, dans des plantes sauvages infectées par des virus à ADN, les vsARN s’accumulent majoritairement sous forme de 21 et 24nt produits par DCL4 et DCL3 respectivement, indiquant que ces 2 protéines sont les Dicer prévalents dans l’immunité contre ce type de virus (Figure 2A). Les vsARN de 22nt n’apparaissent qu’après l’inactivation de dcl4, renforçant encore le caractère redondant du rôle de DCL2 dans la défense antivirale. Ainsi, une hypersusceptibilité au CaMV n’a pu être observée que sur le triple mutant dcl2/dcl3/dcl4, suggérant que les vsARN de 24nt peuvent, dans ce cas, potentiellement déclencher du silencing transcriptionel sur le génome viral à ADN qui se réplique dans le noyau sous forme de mini-chromosomes.Enfin, l’inactivation de DCL1 conduit à une diminution générale des quantités de vsARN dérivant du CaMV. De manière surprenante, dans le triple mutant dcl2/dcl3/dcl4, la production de vsARN par DCL1 est quasiment négligeable comme cela a été précédemment observé pour les virus à ARN. À la suite de ces observations, il fut proposé que DCL1 agit précocement dans la cascade d’action des Dicer, probablement en excisant la région 35S leader du transcrit viral primaire dans un processus réminiscent de son activité nucléaire de maturation des pre-miARN [18]. Cette excision fait de l’ARNdb viral un meilleur substrat pour l’action des autres Dicer (Figure 2A). Cet effet ne s’observe pas sur les structures ARNdb présentes le long du génome des virus à ARN vraisemblablement à cause de leur localisation strictement cytoplasmique. Une fois produits, ces vsARN seront incorporés dans le RISC antiviral qui assurera le clivage ou l’inhibition de la traduction des transcrits viraux et de tout transcrit cellulaire présentant une homologie de séquence (on parle alors de virus induced gene silencing ou VIGS) [30,
31] (Figure 2B). Parmi les 10 AGO d’Arabidopsis, seule AGO1 a pour l’instant été impliqué dans la défense antivirale [14]. Toutefois, AGO1 s’associe préférentiellement aux sARN commençant par une uridine qui ne représentent qu’une fraction du répertoire complet des vsARN produits au cours de l’infection [24,
32,
33]. Étant donné qu’AGO2, AGO4 et AGO7 recrutent majoritairement les sARN avec une adénosine en 5’-terminal et AGO5 les sARN commençant par une cytosine, leur association avec le RISC antiviral n’est pas à exclure [24, 32, 33]. De même, AGO4 et AGO6 étant responsables du silencing transcriptionnel chez Arabidopsis, leur rôle potentiel dans la défense contre les virus à ADN est probable [
34,
35]. |
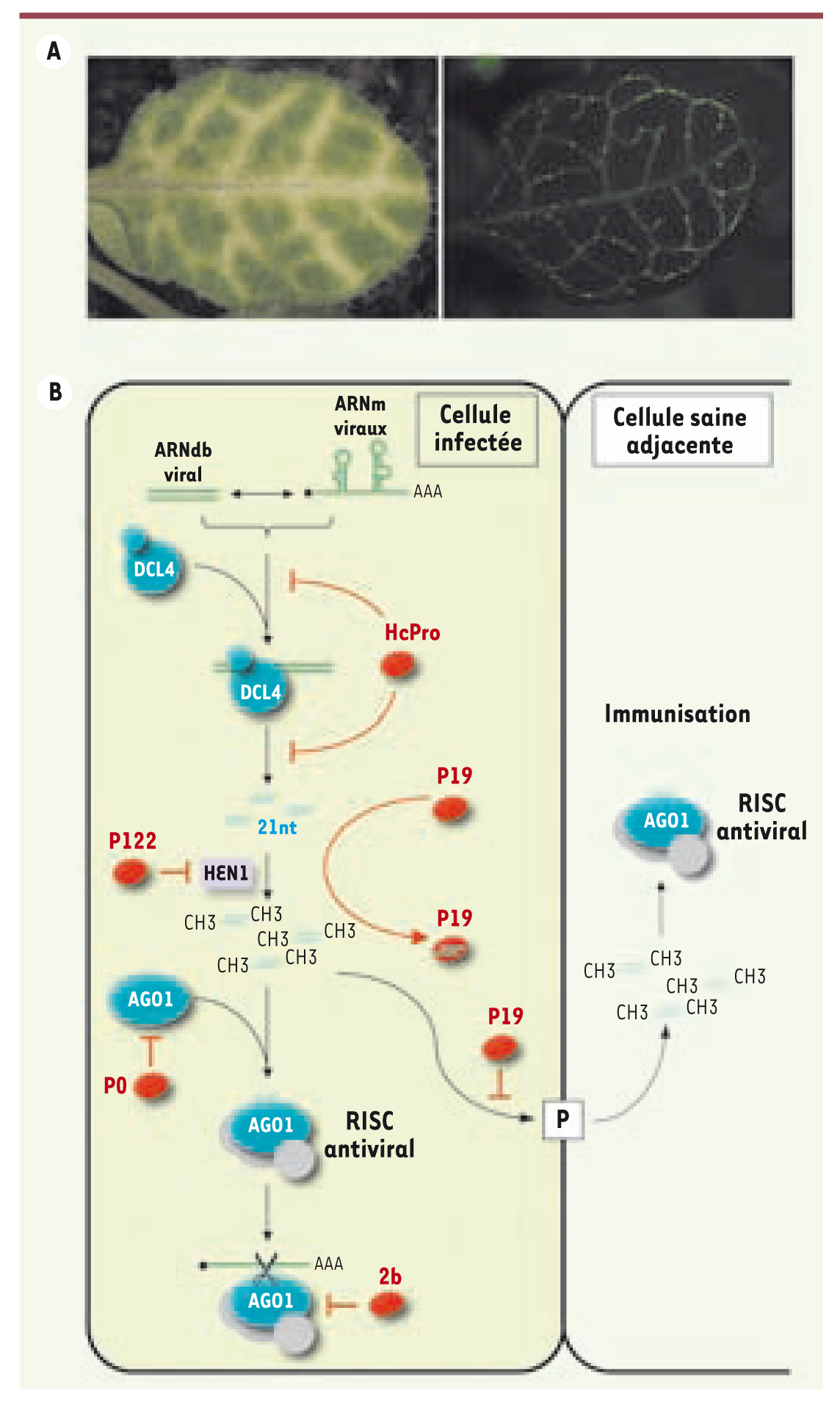
Variété et sophistication des stratégies de contre-défense virales La découverte que virtuellement tous les phytovirus codent pour des protéines qui suppriment le RNA silencing (viral suppressor of RNA silencing ou VSR) à des étapes très variées a renforcé l’idée selon laquelle ce processus représente le principal mécanisme de défense antivirale chez les plantes [12–14] (Figure 3B). De manière frappante, les VSR sont hautement divergentes en termes de séquence, de structure mais aussi des stratégies développées pour inhiber le RNA silencing, représentant un parfait exemple de convergence évolutive.  | Figure 3.
A. Illustration du mouvement de cellule-à-cellule du RNA silencing. Feuille d’Arabidopsis transgénique exprimant un ARNdb dirigé contre le messager cellulaire du gène Sulphur (gauche). L’expression de ce transgène est pilotée par un promoteur spécifique des cellules compagnes du phloème qui ne constituent qu’une seule assise cellulaire dans la feuille, comme on le voit lorsque ce même promoteur pilote l’expression de la green fluorescent protein (droite). Le silencing de ce gène entraîne un jaunissement des cellules exprimant cette construction, mais aussi celui des 10 à 15 cellules adjacentes grâce au mouvement de cellule-à-cellule du signal de silencing. Ce signal se propage d’une cellule à l’autre via les plasmodesmes (canaux reliant la quasi-totalité des cellules végétales) et il est majoritairement, si ce n’est exclusivement, constitué des siARN de 21nt issus de l’action de DCL4 sur l’ARNdb. B. Stratégies virales de suppression du processus de RNA silencing. Une stratégie pour supprimer le silencing consiste à inhiber la production des vsARN comme dans le cas de la protéine HCPro des Potyvirus. Cet effet s’exerce probablement par la fixation de l’ARN double brin (ARNdb) par HcPro bloquant ainsi son accès aux différents Dicer antiviraux. L’inhibition de l’activité de HEN1 par la P122 du Tobacco mosaic virus entraîne la déstabilisation des vsARN. Une autre stratégie consiste à séquestrer les vsARN, comme on l’observe pour la protéine P19 des Tombusvirus, empêchant ainsi leur incorporation dans le RISC antiviral. L’activité d’AGO1 associée au vsARN peut également être inhibée par une interaction directe avec le suppresseur viral comme cela a été démontré pour la protéine 2b du Cucumber mosaic virus. Une autre possibilité est que l’accumulation d’AGO1 soit ciblée, comme dans le cas de la P0 des Polerovirus qui stimulent la vitesse de dégradation des protéines Argonaute, probablement via une voie de protéolyse dépendante de l’ubiquitine. Les vsARN de 21nt produits par DCL4, le Dicer antiviral principal, se déplacent en aval du front d’infection et sont incorporés dans le RISC des cellules saines adjacentes, établissant une réponse antivirale puissante similaire à l’immunisation. La séquestration des vsARN par P19 empêche le mouvement de ce signal et permet ainsi au virus d’envahir les autres cellules. |
Parmi les différentes stratégies de contre-défense adoptées par les VSR, l’une des plus simples consiste à supprimer le RNA silencing en inhibant son initiation. C’est le cas par exemple de la protéine Hc-Pro (pour helper component-proteinase) des potyvirus dont l’expression est associée à une stabilisation de l’ARNdb et à une diminution concomitante de la production des sARN qui en dérivent [
36]. Comme Hc-Pro possède la capacité de fixer l’ARN de manière non séquence-spécifique, cette propriété peut expliquer la stabilisation de l’ARNdb en empêchant les Dicer d’y accéder. Alternativement, les VSR peuvent aussi accélérer le turn-over des sARN. En effet, les vsARN sont, comme leurs homologues endogènes, stabilisés par l’ajout d’un groupement méthyl à leur extrémité 3’, catalysé par la protéine HEN1. L’inhibition de l’activité de cette méthyltransférase, comme cela a été démontré entre autres pour la protéine p122 des tobamovirus [
37], augmentera donc potentiellement la vitesse de dégradation des vsARN. Une autre stratégie pour supprimer le RNA silencing repose sur la séquestration des sARN, empêchant ainsi leur incorporation dans le complexe RISC. L’un des meilleurs exemples de cette approche est fourni par la protéine P19 des tombusvirus dont le cristal en association avec des siARN a été obtenu [
38,
39]. La structure de ce cristal révèle que P19 fonctionne en homodimère qui se fixe spécifiquement aux sARN de 21nt de longueur. De ce fait, P19 fonctionne comme un calibreur moléculaire qui interagit sélectivement et indépendamment de toute séquence avec les duplexes de sARN dont la taille correspond à celle produite par le Dicer antiviral principal, DCL4 (Figure 3B). Les virus peuvent également cibler des facteurs cellulaires requis pour le RNA silencing tels que les protéines AGO. Cette stratégie a récemment été mise en évidence avec deux VSR distinctes : la protéine 2b des cucumovirus et la protéine P0 des luteovirus. La 2b interagit avec la protéine AGO1 au niveau de ses domaines PAZ et PIWI et inhibe son activité de clivage des ARN ciblés [
40]. La protéine P0 agit, elle, comme une protéine à F-BOX en induisant la dégradation, probablement via le protéasome, de différentes protéines de la famille Argonaute [
41]. |
Un aspect spectaculaire du RNA silencing chez les plantes est sa nature systémique. Activé localement dans une feuille, il peut se propager à travers toute la plante en empruntant les plasmodesmes (Figure 3A) et les canaux du phloème (tissu vasculaire assurant la conduction de la sève élaborée dans la plante) pour son transport intercellulaire et à longue distance, respectivement [
42,
43]. La molécule signal, compte tenu de la spécificité de séquence de son effet, doit nécessairement incorporer un acide nucléique, probablement un sARN. Cette composante systémique fait partie intégrante de la fonction antivirale du RNA silencing : activé dans une lésion locale, par réplication d’un virus, un signal séquence-spécifique du génome du pathogène se propage dans des tissus éloignés des zones virosées, les prémunissant ainsi contre l’infection à venir [
44] (Figure 3B). Pour contrer cette activation systémique du RNA silencing par la plante, les virus contre-attaquent en produisant des VSR inhibant spécifiquement la production ou le mouvement de ces signaux non cellule-autonome [
45]. L’étude de la P19 a mis en évidence l’importance de la suppression du RNA silencing sur la propagation virale systémique. Comme cela a été mentionné précédemment, P19 séquestre les siARN empêchant ainsi leur incorporation dans le complexe RISC. De manière surprenante, en dépit de cet effet sur le RNA silencing intracellulaire, des travaux précédents ont établi qu’un tombusvirus n’exprimant pas P19 s’accumule dans des cellules isolées à des niveaux identiques à ceux d’un virus sauvage, suggérant que P19 cible une étape du RNA silencing non cellule-autonome [
46]. Par la suite, il a été montré que ni la réplication de ce virus muté ni son mouvement à longue distance, via les tissus du phloème, n’étaient altérées [
47]. En revanche, l’absence de P19 empêche l’invasion par ce virus du reste de la feuille, qui reste ainsi confiné aux veines des feuilles systémiques. De plus, ces feuilles sont résistantes à une infection secondaire par le même virus ou par un virus possédant une homologie de séquence avec ce tombusvirus [
48]. Ces résultats suggèrent donc que P19 bloque très certainement la synthèse ou le mouvement du signal du silencing antiviral qui permet la destruction des ARN viraux en aval du front d’infection. En accord avec cette hypothèse, il est à noter que P19 fixe spécifiquement les sARN de 21nt produits par DCL4 qui ont été identifiés comme étant le principal, si ce n’est exclusif, signal du mouvement de cellule à cellule du RNA silencing [
49,
50] (Figure 3B). |
Perspectives et futures directions La réponse immunitaire innée des plantes relayée par des interactions protéine-protéine spécifiques implique des gènes de résistance (R) [
51]. Le produit de ces gènes veille sur l’intégrité de composés spécifiques assurant la défense de l’hôte et qui sont les cibles principales des facteurs de virulence des pathogènes. Tout changement dans l’état de ces composés stimule les protéines R qui déclenchent une réaction de défense de l’hôte pouvant culminer en une forme de mort cellulaire programmée (appelée réaction hypersensible ou HR) qui restreint le développement de la maladie. Les VSR sont par nature des facteurs de virulence dont un certain nombre est connu pour déclencher une réaction HR chez des hôtes spécifiques. On peut ainsi imaginer que des gènes R ont évolué pour détecter spécifiquement les dommages créés par les VSR sur des composants du RNA silencing antiviral, constituant ainsi une forme de « contre-contre » défense de la plante [
52]. Échapper à la reconnaissance des gènes R peut également constituer une forte pression de sélection guidant l’évolution des VSR et illustrant la course à l’armement sans fin opposant les plantes et leur pathogènes. De plus, le rôle du RNA silencing dans la défense des plantes face à leurs agresseurs ne semble pas restreint aux seuls virus. Ainsi, il a récemment été démontré que ce processus contribue à la défense contre différents types de bactéries phytopathogènes [
53,
54]. Enfin, la question du rôle antiviral du RNA silencing demeure entière chez les vertébrés et les mammifères où aucun vsARN n’a pu être détecté dans des cellules somatiques infectées, même par séquençage à haut débit de petits ARN cellulaires clonés. Ces expériences de séquençage ont cependant révélé un phénomène totalement inattendu, et spécifique des virus de mammifères, principalement les herpesviridae. On s’est en effet aperçu qu’une fraction parfois très importante des miARN de cellules infectées par de tels virus est en fait constituée de miARN produits à partir de transcrits viraux [
55,
56]. Cette découverte a des implications fondamentales en santé humaine et vétérinaire car ces miARN viraux semblent jouer un rôle très important, non seulement dans l’expression des génomes viraux, mais aussi dans l’établissement d’un terrain favorable à l’infection. Par exemple, cette usurpation de la voie miARN de l’hôte s’illustre dans le cas du virus SV40 par l’élimination de ses transcrits précoces, produisant l’antigène T, par des miARN viraux codés par son génome, lui permettant ainsi de contourner le système immunitaire [
57]. Il est enfin très probable que les patrons cellulaires de miARN de l’hôte jouent un rôle non négligeable dans le tropisme tissulaire de certains virus de vertébrés, comme cela a été démontré avec le virus de l’hépatite C et le rétrovirus foamy des primates [
58–
60]. |
1. Napoli C, Lemieux C, Jorgensen R. Introduction of a chimeric Chalcone Synthase gene into petunia results in reversible co-suppression of homologous genes in trans. Plant Cell 1990; 2 : 279–89. 2. Van der Krol AR, Mur LA, Beld M, et al. Flavonoid genes in petunia: addition of a limited number of gene copies may lead to a suppression of gene expression. Plant Cell 1990; 2 : 291–9. 3. Bartel DP. MicroRNAs: genomics, biogenesis, mechanism and function. Cell 2004; 116 : 281–97. 4. Jones-Rhoades MW, Bartel DP, Bartel B. MicroRNAs and their regulatory roles in plants. Annu Rev Plant Biol 2006; 57 : 19–53. 5. Fire A, Xu S, Montgomery MK, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature 1998; 391 : 806–11. 6. Bernstein E, Caudy AA, Hammond SM, Hannon GJ. Role for a bidentate ribonuclease in the initation step of RNA interference. Nature 2001; 409 : 363–6. 7. Hamilton AJ, Baulcombe DC. A species of small antisense RNA in posttranscriptional gene silencing in plants. Science 1999; 286 : 950–52. 8. Elbashir SM, Lendeckel W, Tuschl T. RNA interference is mediated by 21- and 22-nucleotide RNAs. Genes Dev 2001; 15 : 188–200. 9. Hammond SM, Bernstein E, Beach D, Hannon GJ. An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells. Nature 2000; 404 : 293–6. 10. Mallory AC, Bouché N. MicroRNA-directed regulation: to cleave or not to cleave. Trends Plant Sci 2008; 13 : 359–67. 11. Ekwall K. The RITS complex-A direct link between small RNA and heterochromatin. Mol Cell 2004; 13 : 304–5. 12. Voinnet O. Induction and suppression of RNA silencing : insights from viral infections. Nat Rev Genet 2005; 6 : 206–20. 13. Li F, Ding SW. Virus counterdefence: diverse strategies for evading the RNA-silencing immunity. Annu Rev Microbiol 2006; 60 : 503–31. 14. Ding SW, Voinnet O. Antiviral immunity directed by small RNAs. Cell 2007; 130 : 413–26. 15. Molnár A, Csorba T, Lakatos L, et al. Plant virus-derived small interfering RNAs originate predominantly from highly structured single-stranded viral RNAs. J Virol 2005; 79 : 7812–8. 16. Ho T, Pallett D, Rusholme R, et al. A simplified method for cloning of short interfering RNAs from Brassica juncea infected with Turnip mosaic potyvirus and Turnip crinkle carmovirus. J Virol Meth 2006; 136 : 217–23. 17. Chellappan P, Vanitharani R, Pita J, Fauquet CM. Short interfering RNA accumulation correlates with host recovery in DNA virus-infected hosts, and gene silencing targets specific viral sequences. J Virol 2004; 78 : 7465–77. 18. Moissiard G, Voinnet O. RNA silencing of host transcripts by cauliflower mosaic virus requires coordinated action of the four Arabidopsis Dicer-like proteins. Proc Natl Acad Sci USA 2006; 103 : 19593–98. 19. Rajagopalan R, Vaucheret H, Trejo J, Bartel DP. A diverse and evolutionarily fluid set of microRNAs in Arabidopsis thaliana. Genes Dev 2006; 20 : 3407–25. 20. Vazquez F, Blevins T, Ailhas J, et al. Evolution of Arabidopsis MIR genes generates novel microRNA classes. Nucleic Acids Res 2008; 36 : 6429–38. 21. Chapman EJ, Carington JC. Specialization and evolution of endogenous small RNA pathways. Nat Rev Genet 2007; 8 : 884–96. 22. Vaucheret H, Vazquez F, Crété P, Bartel DP. The action of ARGONAUTE1 in the miRNA pathway and its regulation by the miRNA pathway are crucial for plant develoment. Genes Dev 2004; 18 : 1187–97. 23. Brodersen P, Sakvarelidze-Achard L, Bruun-Rasmussen M, et al. Widespread translational inhibition by plant miRNAs and siRNAs. Science 2008; 320 : 1185–90. 24. Mi S, Cai T, Hu Y, et al. Sorting of small RNAs into Arabidopsis argonaute complexes is directed by the 5’ terminal nucleotide. Cell 2008; 133 : 116–27. 25. Zilberman D, Cao X, Jacobsen SE. ARGONAUTE4 control of locus-specific siRNA accumulation and DNA and histone methylation. Science 2003; 299 : 716–9. 26. Zheng X, Zhu J, Kapoor A, Zhu JK. Role of Arabidopsis AGO6 in siRNA accumulation, DNA methylation and transcriptional gene silencing. EMBO J 2007; 26 : 1691–701. 27. Yang Z, Ebright YW, Yu B, Chen X. HEN1 recognizes 21-24 nt small RNA duplexes and deposits a methyl group onto 2’ OH of the 3’ terminal nucleotide. Nucleic Acids Res 2006; 34 : 667–75. 28. Bouché N, Lauressergues D, Gasciolli V, Vaucheret H. An antagonistic function for Arabidopsis DCL2 in development and a new function for DCL4 in generating viral siRNAs. EMBO J 2006; 25 : 3347–56. 29. Deleris A, Gallego-Bartolome J, Bao J, et al. Hierarchical action and inhibition of plant Dicer-like proteins in antiviral defense. Science 2006; 313 : 68–71. 30. Blevins T, Rajeswaran R, Shivaprasad PV, et al. Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing. Nucleic Acids Res 2006; 34 : 6233–46. 31. Ruiz MT, Voinnet O, Baulcombe DC. Initiation and maintenance of virus-induced gene silencing. Plant Cell 1998; 10 : 937–46. 32. Takeda A, Iwasaki S, Watanabe T, et al. The mechanism selecting the guide strand from small RNA duplexes is different among argonaute proteins. Plant Cell Physiol 2008; 49 : 493–500. 33. Montgomery TA, Howell MD, Cuperus JT, et al. Specificity of ARGONAUTE7-miR390 interaction and dual functionality in TAS3 trans-acting siRNA formation. Cell 2008; 133 : 128–41. 34. Raja P, Sanville BC, Buchmann RC, Bisaro DM. Viral genome methylation as an epigenetic defense against geminiviruses. J Virol 2008; 82 : 8997–9007. 35. Rodriguez-Negrete EA, Carrillo-Tripp J, Rivera-Bustamante RF. RNA silencing against geminivirus: complementary action of posttranscriptional gene silencing and transcriptional gene silencing in host recovery. J Virol 2009; 83 : 1332–40. 36. Dunoyer P, Lecellier CH, Parizotto EA, et al. Probing the miRNA and small interfering RNA pathways with virus-encoded suppressors of RNA silencing. Plant Cell 2004; 16 : 1235–50. 37. Vogler H, Akbergenov R, Shivaprasad PV, et al. modification of small RNAs associated with suppression of RNA silencing by tobamovirus replicase protein. J Virol 2007; 81 : 10379–88. 38. Ye K, Malinina L, Patel DJ. Recognition of small interfering RNA by a viral suppressor of RNA silencing. Nature 2003; 426 : 874–8. 39. Vargason JM, Szittya G, Burgyan J, Hall TM. Size selective recognition of siRNA by an RNA silencing suppressor. Cell 2003; 115 : 799–811. 40. Zhang X, Yuan YR, Pei Y, et al. Cucumber mosaic virus-encoded 2b suppressor inhibits Arabidopsis Argonaute1 cleavage activity to counter plant defense. Genes Dev 2006; 20 : 3255–68. 41. Baumberger N, Tsai CH, Lie M, et al. The Polerovirus silencing suppressor P0 targets ARGONAUTE proteins for degradation. Curr Biol 2007; 17 : 1609–14. 42. Voinnet O. Non-cell autonomous RNA silencing. FEBS Lett 2005; 579 : 5858–71. 43. Dunoyer P, Voinnet O. RNA as a signalling molecule. In : Fleming A, ed. Intercellular communication in plants. Annual Plant Reviews, vol. 16. Sheffield, UK : Blackwell Publishing, 2005 : 49–84. 44. Voinnet O, Lederer C, Baulcombe DC. A viral movement protein prevents spread of the gene silencing signal in Nicotiana benthamiana. Cell 2000; 103 : 157–67. 45. Himber C, Dunoyer P, Moissiard G, et al. Transitivity-dependent and -independent cell-to-cell movement of RNA silencing. EMBO J 2003; 22 : 4523–33. 46. Silhavy D, Molnar A, Lucioli A, et al. A viral protein suppresses RNA silencing and binds silencing-generated 21- to 25-nucleotide double-stranded RNAs. EMBO J 2002; 21 : 3070–80. 47. Havelda Z, Hornyik C, Crescenzi A, Burgyan J. In situ characterization of Cymbidium ringspot tombusvirus infection-induced posttranscriptional gene silencing in Nicotiana benthamiana. J Virol 2003; 77 : 6082–6. 48. Szittya G, Molnar A, Silhavy D, et al. Short defective interfering RNAs of tombusviruses are not targeted but trigger post-transcriptional gene silencing against their helper virus. Plant Cell 2002; 14 : 359–72. 49. Dunoyer P, Himber C,Voinnet O. DICER-LIKE 4 is required for RNAi and produces the 21nt siRNA component of the plant cell-to-cell silencing signal. Nat Genet 2005; 37 : 1356–60. 50. Dunoyer P, Himber C, Ruiz-Ferrer V, et al. Intra- and inter-cellular RNA interference in Arabidopsis requires components of the microRNA and heterochromatic silencing pathways. Nat Genet 2007; 39 : 848–56. 51. Jones JD, Dangl JL. The plant immune system. Nature 2006; 444 : 323–9. 52. Li HW, Lucy AP, Guo HS, et al. Strong host resistance targeted against a viral suppressor of the plant gene silencing defence mechanism. EMBO J 1999; 18 : 2683–91. 53. Dunoyer P, Himber C, Voinnet O. Induction, suppression and requirement of RNA silencing pathways in virulent Agrobacterium infections. Nat Genet 2006; 38 : 258–263. 54. Navarro L, Dunoyer P, Jay F, et al. A microRNA contributes to arabidopsis basal resistance by repressing auxin-signalling. Science 2006; 312 : 436–439. 55. Pfeffer S, Zavolan M, Grässer FA, et al. Identification of virus-encoded microRNAs. Science 2004; 304 : 734–6. 56. Dölken L, Perot J, Cognat V, et al. Mouse cytomegalovirus microRNAs dominate the cellular small RNA profile during lytic infection and show features of posttranscriptional regulation. J Virol 2007; 81 : 13771–82. 57. Sullivan CS, Grundhoff AT, Tevethia S, et al. SV40-encoded microRNAs regulate viral gene expression and reduce susceptibility to cytotoxic T cells. Nature 2005; 435 : 682–6. 58. Lecellier CH, Dunoyer P, Arar K, et al. A cellular microRNA mediates antiviral defense in human cells. Science 2005; 308 : 557–60. 59. Jopling CL, Yi M, Lancaster AM, et al. Modulation of hepatitis C virus RNA abundance by a liver-specific microRNA. Science 2005; 309 : 1577–81. 60. Chang J, Guo JT, Jiang D, et al. Liver-specific microRNA miR-122 enhances the replication of hepatitis C virus in nonhepatic cells. J Virol 2008; 82 : 8215–23. 61. Ladeiro Y, Zucman-Rossi J. Micro-ARN (miARN) et cancer : le cas des tumeurs hépatocellulaires. Med Sci (Paris) 2009; 25 : 467–72. |