| |
| Med Sci (Paris). 2009 June; 25(6-7): 601–607. Published online 2009 June 15. doi: 10.1051/medsci/2009256-7601.Biologie des systèmes appliqués aux cancers Emmanuel Barillot,* Laurence Calzone, and Andrei Zinovyev Institut Curie, 26, rue d’Ulm, F-75248 Paris Cedex 05 Inserm, U900, F-75248 Paris École des Mines ParisTech, F-75248 Paris, France |
Pourquoi une biologie des systèmes appliqués aux cancers ? Le terme de biologie des systèmes est aujourd’hui si couramment utilisé qu’il importe de préciser l’acception qu’on lui prête avant de l’employer. Il est admis que la discipline vise à construire un modèle mathématique qui rende compte du fonctionnement d’un système biologique. Mais la démarche de biologie des systèmes présente certaines spécificités. Tout d’abord, elle suppose d’étudier un système biologique dans sa globalité, et non de se limiter à une partie comme dans les approches classiques où l’on se focalise sur un seul gène. Elle requiert qu’on s’intéresse à plusieurs niveaux d’échelle, du moléculaire au cellulaire, à l’organe voire à l’individu dans notre cas. Elle s’appuie sur des allers-retours entre théorie et expérience, qui permettent d’affiner progressivement les modèles et de les valider. Enfin, elle intègre nos connaissances préalables sur le fonctionnement du système. Sans exiger une description complètement mécaniste (qui par exemple détaillerait explicitement toutes les interactions moléculaires et serait donc irréaliste), la modélisation systémique ne se contente pas des stratégies purement statistiques. Ces dernières opèrent typiquement en régressant des variables cibles (par exemple phénotypiques) sur les variables des niveaux inférieurs (par exemple l’expression des gènes), même de niveaux de descriptions multiples (ADN, ARN, protéines, cellules, etc.). Si l’approche statistique s’est montrée très fructueuse et reste incontournable en l’absence de connaissance préalable sur le comportement du système étudié, nos progrès dans la compréhension des mécanismes moléculaires et cellulaires qui régissent le fonctionnement des cellules normales et tumorales nous amènent aujourd’hui à proposer une nouvelle approche de ces systèmes. Durant les trois dernières décennies, notre connaissance des mécanismes à l’origine de la tumorigenèse et de la progression tumorale s’est considérablement affinée. Dans un premier temps, quelques gènes qui participent à ces mécanismes ont été identifiés. On a même pu croire pendant un temps avoir découvert le gène ou les gènes du cancer. Citons comme exemples célèbres RB (rétinoblastome), Src, Ras ou P53. Fort logiquement, la communauté scientifique s’est alors attachée à caractériser ces gènes, leurs interactions et leur rôle individuel dans différents contextes. Après quelques succès, cette approche a montré rapidement ses limites : il est apparu que le cancer revêtait une plus grande complexité étiologique et que sa survenue et son développpement supposaient le dysfonctionnement de plusieurs gènes, sans pour autant que les raisons de cette complexité ne soient comprises. Progressivement, l’apparition de la maladie a été reliée non plus directement à des gènes, mais à des fonctions cellulaires, bien entendu sous contrôle génétique : prolifération cellulaire, différenciation, mort cellulaire, angiogenèse, invasion tissulaire (ce qui a été brillamment résumé dans un article de Hanahan et Weinberg) [
1]. À ce stade, le déchiffrage de ces fonctions en voies moléculaires, c’est-à-dire en réseaux d’interactions moléculaires entre gènes et protéines, et notre capacité récente à mesurer le fonctionnement de ces systèmes aux niveaux génomique, transcriptomique, protéomique et épigénomique grâce aux nouvelles technologies, ont ouvert la voie à l’approche de biologie des systèmes appliqués aux cancers qui s’appuie sur la modélisation mathématique explicite de ces réseaux. Citons comme types de modèles mathématiques possibles les systèmes d’équations différentielles, les réseaux bayésiens, les réseaux de Petri, ou les modèles logiques (voir [
2] pour une présentation assez exhaustive). Pourtant, il subsiste encore un certain scepticisme quant au bien-fondé d’une telle approche pour l’étude d’un système aussi complexe qu’une tumeur : peut-on escompter des prédictions quantitatives dans une situation de si grande incertitude, alors que les paramètres du système sont dans leur très grande majorité inconnus et difficiles à mesurer, et que le nombre de variables est de plusieurs milliers ? De telles approches n’ont-elles pas par le passé montré leurs limites accouchant au mieux de prédictions triviales qui ne nécessitaient nullement le recours à une telle complexité ? Cette position ignore ou sous-estime les rôles que la modélisation mathématique peut jouer dans l’étude d’un système. C’est précisément de ces rôles dont il est question dans la suite de cet article, exemples à l’appui. Notons qu’ils touchent à la fois à la communication scientifique, aux progrès conceptuels, à la modélisation proprement dite, et aux applications concrètes, cliniques par exemple. On attend en particulier de la biologie des systèmes appliqués aux cancers qu’elle éclaire sur le choix des cibles thérapeutiques à la lumière des propriétés systémiques de la pathologie cancéreuse [
3]. Bien entendu cela suppose une démarche de modélisation appropriée pour les données disponibles (réseaux, mesures d’expression, mutations, phénotypes observés, etc.), mais aussi une planification d’expériences rigoureuses afin de collecter les informations nécessaires à la question systémique posée. Une interaction très forte entre expérimentateurs et théoriciens, dès la définition du projet d’étude, est donc indispensable. |
Un langage de description non équivoque du système Le langage des réseaux d’interaction La description précise des réseaux d’interactions moléculaires à l’œuvre dans la cellule saine comme au cours de la progression tumorale est typiquement rapportée le texte des articles parus dans la littérature scientifique ou sous forme de diagrammes dont la compréhension requiert une légende. C’est également le cas dans les bases de données traditionnelles, telle KEGG ( Kyoto encyclopedia of genes and genomes) [
4]. Ainsi, selon le type de graphe par exemple, une flèche peut indiquer l’influence d’une protéine (ou d’un gène) sur une (ou un) autre, une activation, une modification, une complexation, une translocation, etc. De même, dans le texte d’un article d’expérimentations, certains termes évasifs tels que « activation » ou « médiation » peuvent avoir différentes définitions selon le contexte et ne sont pas toujours explicités, ce qui rend l’interprétation difficile pour les néophytes. D’une part, ces formulations souffrent d’ambiguïté, et d’autre part, elles ne fournissent pas directement une description formelle du système qui peut être manipulée par un ordinateur et traduite en modèle mathématique. Pour y remédier, la communauté scientifique a fait plusieurs propositions aujourd’hui largement acceptées pour la description des systèmes biologiques, et qui de surcroît fournissent un langage précis et commun aux expérimentateurs et aux théoriciens. Parmi ces langages, on trouve : (1) le langage Systems Biology Graphical Notation (SBGN, http://www.sbgn.org) qui propose un standard de représentation graphique de diagrammes exprimant notre connaissance des interactions et régulations moléculaires ; (2) le langage Biological Pathways Exchange (BioPAX, http://www.biopax.org) qui est basé sur une ontologie visant à décrire formellement les voies métaboliques, les interactions moléculaires, les voies de transduction du signal, la régulation de l’expression des gènes et les interactions génétiques ; (3) le langage Systems Biology Markup Language (SBML) qui propose une description standard des modèles mathématiques de systèmes biochimiques [
5]. De nombreux modèles de réseaux biologiques exprimés en SBML sont regroupés dans la base BioModels [
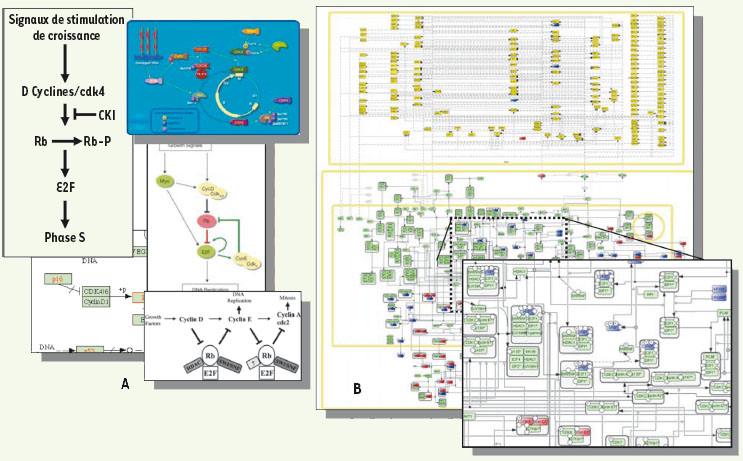
6]. Diagramme de la régulation du cycle cellulaire par la protéine retinoblastoma À l’Institut Curie, nous avons entrepris à partir de la littérature scientifique la description précise de la régulation du cycle cellulaire par la protéine retinoblastoma (RB/RB1) [
7], un des premiers suppresseurs de tumeurs découverts. RB, acteur agissant de manière négative sur le cycle cellulaire, est impliqué dans la plupart des cancers [
8]. Notre description exhaustive est partie du diagramme simple, incomplet et ambigu, proposé dans des publications et bases de données (Figure 1A). C’est autour de cette représentation très insuffisante que nous avons détaillé les interactions régulant l’activité de RB mais aussi celles de protéines de la même famille que RB (p107, p130) ainsi que leurs partenaires principaux : les facteurs de transcription E2F et leurs gènes cibles. La voie RB/E2F est reliée à bien d’autres et la définition que nous avons donnée de ses limites n’échappe pas à un certain arbitraire. Dans sa forme actuelle, elle contient 80 protéines qui existent sous la forme de 208 espèces chimiques (en incluant les complexes et les modifications post-traductionnelles). Le diagramme a été décrit en langage SBGN, à l’aide du logiciel CellDesigner, l’un des principaux éditeurs de réseaux biologiques incluant des liens vers les bases de données et les références bibliographiques [
9].  | Figure 1.
Diagramme de représentations de la voie RB/E2F. A. Différents types de représentations de la voie RB/E2F utilisant des formats et des hypothèses variés [
31,
32] ; KEGG, http://www.genome.jp/kegg/ ; BioCarta, http://www.biocarta.com.
B. Reconstruction détaillée du réseau RB/E2F en standard SBGN ( Systems Biology Graphical Notation) et détail d’une partie du réseau. Le diagramme est divisé en compartiments : gènes régulés par les facteurs de transcription E2F en haut et les interactions protéiques prenant place dans le cytoplasme et le noyau au-dessous. Les nœuds rouges sont les oncogènes connus et les bleus les suppresseurs de tumeurs. |
Le diagramme construit est utile à plusieurs égards. Il donne d’abord de la voie de RB/E2F une description détaillée et formelle, c’est-à-dire immédiatement disponible à l’analyse mathématique. Il permet, par exemple, de rechercher les chemins entre deux molécules, de déterminer les modules fonctionnels ou d’identifier toutes les rétroactions de régulation. De simples raisonnements basés sur la théorie des graphes fournissent alors des explications plausibles à certains phénomènes observés qui apparaissaient contre-intuitifs sous l’éclairage des diagrammes simplifiés de la voie de RB/E2F. Grâce à la carte détaillée et au modèle mathématique associé, nous comptons proposer des explications logiques à certains de ces phénomènes inexpliqués. Par exemple, dans le cancer de la vessie, la perte de RB entraîne une amplification du facteur de transcription E2F3 mais pas de E2F1 ou E2F2, ou encore une surexpression de p16/INK4a, un inhibiteur des CDK (kinases cyclines-dépendantes) probablement régulé transcriptionnellement par RB ; ces observations sont contraires a priori à notre compréhension intuitive du système. Le diagramme est aussi utile à l’analyse des données « omiques » (typiquement le transcriptome d’une série de tumeurs), par exemple pour déterminer les régions de la voie RB/E2F les plus enrichies en gènes dérégulés. Il peut également servir pour orienter la construction de règles de prédiction pour classifier les tumeurs, comme pour distinguer celles qui développeront des métastases des autres [
10]. Enfin il constitue un point de départ indispensable à la création d’un modèle mathématique dynamique de la voie RB/E2F, et ce quel que soit le formalisme retenu (cinétique chimique, discret, par réseau de Petri, etc. [2]). Ce dernier est destiné à prédire le comportement de la voie sous l’effet de perturbations diverses (génétiques, épigénétiques, chimiques, etc.). |
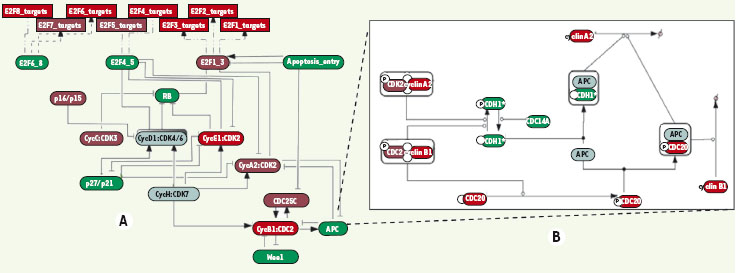
Un modèle pour orienter l’étude des réseaux biologiques Ce diagramme (Figure 1B) est difficilement exploitable en l’état, et il est nécessaire d’en donner une représentation plus abstraite et plus condensée, c’est-à-dire d’en réduire la complexité, sans perdre d’informations pour autant. Il s’agit donc de décomposer le diagramme en modules fonctionnels [7] (Figure 2A). Chaque nœud dans ce graphe représente un module (ou sous-réseau) du diagramme, baptisé du nom de la protéine qui domine le module. Les arcs résument l’influence positive ou négative d’un module sur l’activité d’un autre. Cette activité est liée directement à la fonction du module, qui peut être activée ou réprimée selon la distribution des états de ses composants. Une définition mathématique de cette fonction peut être formulée si elle est bien comprise, mais on s’appuie en général sur les données « omiques » (expression des protéines et des gènes) pour inférer une activité sur des bases statistiques [
11]. La Figure 2 illustre cette démarche à partir de données d’expression génique d’une série de tumeurs de la vessie [
12]. Chaque échantillon est caractérisé par son profil d’expression de modules de la voie RB/E2F. La structure d’un des modules, APC (anaphase promoting complex), est montrée en détail (Figure 2B) : il en ressort que la suppression de la fonction du module APC dans les tumeurs invasives de vessie est associée à une expression moindre des gènes CDH1 (FZR1 homologue) et CDC14A (cell division cycle 14 homolog A).
 | Figure 2.
A. Représentation modulaire du réseau RB/E2F. Les modules résultent de l’extraction des composantes fortement connexes du graphe de réactions, suivie d’une analyse des cycles élémentaires [ 7]. Les couleurs correspondent aux différentes activités des modules dans des cancers invasifs de la vessie. Les activités sont calculées en fonction des activités des échantillons sains. Dans les modules en vert, l’activité relative est diminuée alors que dans les modules en rouge, elle est amplifiée. B. Chaque nœud en A correspond à un sous-réseau de la carte complète et détaillée présenté dans la Figure 1B. Sont montrés ici la structure interne du module APC et le niveau de transcription moyen et relatif de chaque gène impliqué dans le module avec le même code de couleur qu’en A. Notons que la variation individuelle d’expression des gènes entre tumeurs invasives et tissu normal peut ne pas être significative, alors même que le changement d’activité du module le sera. |
|
Le modèle, laboratoire d’expérimentation biologique in silico
Le but de la modélisation est atteint quand le modèle permet de prédire le comportement du système étudié dans différents contextes, et donc lorsqu’il est soumis à différentes perturbations. Ce rôle prédictif ne peut être validé que dans des situations vérifiées expérimentalement, par exemple si le modèle prédit des comportements phénotypiques. Différents types de perturbations peuvent être simulés mathématiquement, par exemple une mutation qui entraînera l’arrêt du cycle cellulaire (non-dégradation de cyclines, marqueurs de l’une des phases du cycle cellulaire) ou induira l’apoptose (activation de composantes typiques de l’apoptose telles les caspases). Le modèle mathématique peut jouer ici un rôle sinon de substitut, du moins de planification de l’expérimentation biologique : on parle d’expérience in silico. L’un des exemples les plus convaincants à cet égard a concerné le cycle cellulaire de la levure. Dans leurs travaux, Chen et al. [
13] rendent compte du fonctionnement normal du cycle cellulaire de la levure Saccharomyces cerevisiae de type sauvage, ainsi que des propriétés de plus de 100 mutations génétiques. Ils prédisent les phénotypes de nombreuses combinaisons de mutants (de simple à quadruple) placés dans différents milieux (glucose et galactose) et soumis à des conditions diverses, dont certains ont été validés expérimentalement [
14]. De plus, par cette modélisation, certaines valeurs de paramètres (constantes biochimiques) difficiles à mesurer directement in vivo ont pu ainsi être estimées. Dans le cas de la biologie des systèmes appliqués aux cancers, l’affaire est sensiblement plus ardue, du fait de moyens d’investigation biologique plus restreints (knock-out de gènes plus problématique, techniques in vivo rares, hétérogénéité tumorale, règles éthiques, etc.). Pourtant, plusieurs modèles prédictifs de voies de signalisation et de régulation du cycle cellulaire ont été proposés qui concernent par exemple : TGFß (transforming growth factor β), NFkB, MAPK (mitogen-activated protein kinase), EGF (epidermal growth factor), etc. [
15–
18]. De même, un important effort a été entrepris pour intégrer nos connaissances sur la machinerie apoptotique dans un ensemble de modèles mathématiques [
19,
20]. Plusieurs arguments théoriques sont porteurs d’espoir dans l’approche de biologie des systèmes en dépit de la complexité du cancer. En premier lieu, une importance mesurée doit être accordée aux paramètres : le comportement d’un système dynamique n’est pas toujours très sensible aux valeurs des paramètres qui le contrôlent [
21]. Même si notre connaissance de ces paramètres est limitée, nos conclusions montrent qu’un même comportement de la cellule est conservé sur un domaine de validité assez large. Par ailleurs, des résultats qualitatifs sont plus accessibles que les quantitatifs et restent fort utiles d’un point de vue pratique : réduire la prolifération cellulaire ou provoquer l’apoptose peut déjà constituer un critère de choix de cibles, voire un objectif thérapeutique. Ces modèles mathématiques de voies de signalisation ne sont en fait qu’une première étape de la biologie des systèmes qui s’intéresse à la compréhension du comportement tumoral. D’autres défis restent à relever, comme l’élucidation du mystère de la spécificité tissulaire des tumeurs (comment la dérégulation de mécanismes cellulaires génériques, et bien compris comme tels, aboutit à des cancers spécifiques d’un tissu ?), ou de l’énigme de l’intégration des signaux de transduction par la cellule normale ou tumorale pour choisir son destin (mort ou division ?). Tout laisse à penser que la réponse à ces questions ne pourra venir que d’une approche systémique, prenant en compte toutes les interactions entre voies de signalisation qui étaient étudiées, jusqu’à ce jour, de façon indépendante. Enfin, le rôle de la plasticité du génome dans l’évolution tumorale, s’il est fermement établi, reste aussi à modéliser. De nombreux projets s’attachent aujourd’hui à collecter les informations nécessaires : altérations du génome (polymorphismes et mutations de tout ordre), du transcriptome, du protéome (profils d’expression et modifications post-traductionnelles), de l’épigénome (méthylation de l’ADN, modification des histones) [
22]. Elles devront être intégrées dans un modèle mathématique rendant compte de l’effet de ces perturbations sur les réseaux de régulation de l’expression et menant à la prédiction du phénotype tumoral. D’un point de vue pratique, les défis mentionnés ci-dessus ont des applications concrètes immédiates. La complexité de la pathologie cancéreuse s’exprime dans sa résistance aux traitements mono-médicamenteux, et a orienté la recherche pharmaceutique vers la voie des traitements multi-médicaments. Si l’on peut aujourd’hui cribler rapidement un grand nombre de molécules individuellement, les approches combinatoires doivent, quant à elles, s’appuyer sur la modélisation mathématique, qui doit limiter à un nombre raisonnable les combinaisons à valider expérimentalement. |
La modélisation, source d’avancées conceptuelles Ce rôle des modèles mathématiques comme source d’avancées conceptuelles dans le cadre de la biologie des systèmes n’est pas le moindre. Il touche à l’apparition de notions, concepts et paradigmes dans le domaine de la biologie du cancer résultant directement de la méthodologie mathématique mise en œuvre. Cet effet est potentiellement le plus profond et le plus durable, bien qu’il soit difficile à mettre en évidence et à apprécier pleinement. Par exemple, les résultats mathématiques reliant rétroactions positives et négatives dans les systèmes dynamiques avec la multi-stationnarité ou l’homéostasie du système conduisent à des nouveaux plans expérimentaux ciblant les rétroactions du système [
23]. Un autre exemple concerne la modélisation mathématique du cycle cellulaire, aujourd’hui partagée entre deux paradigmes : le premier conçoit le cycle cellulaire comme un oscillateur, c’est-à-dire un mécanisme à rétroaction négative [
24], tandis que le second le présente comme une séquence de transitions de phases irréversibles rythmées par l’évolution continue de la taille de la cellule qui constitue le paramètre critique de contrôle des transitions [
25]. À ces deux vues correspondent des stratégies différentes pour contrecarrer expérimentalement la progression du cycle cellulaire, en particulier en biologie des systèmes appliqués aux cancers. Les avancées conceptuelles découlent souvent de parallèles tracés entre disciplines. La notion de comportement émergent, ou propriétés émergentes (voir l’Encadré de l’article de A. Lesne, p. 586 de ce numéro), empruntée à la théorie des systèmes complexes, est aujourd’hui largement acceptée en recherche contre le cancer : l’idée est que la dérégulation d’une fonction biologique dans une tumeur n’est pas imputable à l’activité individuelle de quelques gènes mais résulte de perturbations collectives et orchestrées par des sous-réseaux ou des voies biologiques (ou plus généralement des modules). Ces différentes perturbations mènent à un même phénotype, alors que les activités individuelles de chaque composant du module peuvent varier significativement d’une tumeur à l’autre. Pour prendre en compte toutes ces variations, on a assisté à l’apparition de méthodes statistiques [
26] visant à déterminer et évaluer l’activité de modules fonctionnels [
27], méthodes qui sont devenues des standards de facto en recherche moléculaire contre le cancer [11]. La notion de robustesse est sans doute le meilleur exemple de concept que la modélisation mathématique permet d’introduire et de rendre opératoire dans l’étude des tumeurs. On parle de comportement robuste aussi bien pour les cellules normales que pour les cellules cancéreuses, les deux notions étant d’ailleurs pertinentes dans l’étude de la progression tumorale [
28]. Les systèmes biologiques remplissent en général leur fonction de façon stable, c’est-à-dire qu’ils s’accommodent de conditions externes (environnement) et internes (contenu cellulaire, y compris les variations génétiques) très variables ; c’est cette propriété des cellules normales, indispensable à leur bon fonctionnement, qu’on qualifie de robustesse. Cette même robustesse pose problème quand il s’agit de proposer des stratégies d’intervention pour faire sortir le système de son état stable, comme, par exemple, dans le traitement de pathologies systémiques (au sens de la biologie des systèmes : maladies immunitaires ou cancers). La résistance multi-drogues est un exemple typique d’échec de la pharmacopée devant la robustesse du système tumoral. Comprendre les raisons de la robustesse ouvrirait donc une voie prometteuse de recherches thérapeutiques. La robustesse soulève d’importantes questions théoriques, avec de fortes implications pratiques, par exemple, sur les stratégies thérapeutiques dans les tumeurs : mise en évidence de points de fragilité, cibles naturelles des interventions thérapeutiques [3], identification des caractéristiques structurelles de la robustesse d’un système [
29,
30], ou simplification robuste des modèles mathématiques du système biologique (par exemple des voies de transduction du signal), dont on peut déterminer les quelques paramètres critiques qui en gouvernent l’évolution [21]. |
La biologie des systèmes appliqués aux cancers est aujourd’hui une approche scientifique reconnue et porteuse d’espoirs en matière clinique. Elle s’appuie d’abord sur une formalisation rigoureuse des réseaux de régulation en des langages complets et non ambigus. Elle donne du système étudié (un réseau d’interaction gouvernant les fonctions cellulaires essentielles : prolifération, différenciation, mort cellulaire) une vue détaillée mais aussi une décomposition modulaire qui facilite l’interprétation des profils d’expression d’une tumeur. Ces modèles mathématiques nous permettent de prédire l’évolution du système, c’est-à-dire à la fois de proposer des points d’intervention pour le développement de médicaments, d’anticiper l’évolution tumorale et de classifier les tumeurs. Ces caractéristiques placent la biologie des systèmes appliqués aux cancers en acteur majeur de la médecine de demain, baptisée par Leroy Hood (Institute for Systems Biology, Seattle, États-Unis) du nom de « médecine P4 » : prédictive, personnalisée, préventive et participative. Les approches de biologie des systèmes promettent d’aider au développement de nouveaux biomarqueurs complexes, en utilisant à la fois les données de la biologie à haut débit et notre connaissance des réseaux biologiques. Les progrès technologiques, par exemple le séquençage massif qui rendra d’ici peu possible le séquençage du génome d’un patient pour le coût d’un test sanguin, ou la métabolomique, feront passer la biologie des systèmes du champ de la recherche scientifique et clinique à celui de la pratique médicale quotidienne. |
EB, LC et AZ sont membres de l’équipe « Biologie des systèmes du cancer » de l’U900, équipe labellisée par la Ligue nationale contre le cancer. Ce travail a été financé en partie par le programme de Projet incitatif et collaboratif de l’Institut Curie (PIC « Bioinformatique et Biostatistiques »).
|
1. Hanahan D, Weinberg RA. The hallmarks of cancer. Cell. 2000; 100 : 57–70. 2. De Jong H. Modeling and simulation of genetic regulatory systems: a literature review. J Comput Biol 2002; 9 : 67–103. 3. Kitano H. Cancer as a robust system: implications for anticancer therapy. Nat Rev Cancer 2004; 4 : 227–35. 4. Kanehisa M, Goto S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res 2000; 28 : 27–30. 5. Hucka M, Finney A, Sauro HM, et al. The systems biology markup language (SBML): a medium for representation and exchange of biochemical network models. Bioinformatics 2000; 19 : 524–33. 6. Le Novère N, Bornstein B, Broicher A, et al. Biomodels database: a free, centralized database of curated, published, quantitative kinetic models of biochemical and cellular systems. Nucleic Acids Res 2006; 34 : D689–91. 7. Calzone L, Gelay A, Zinovyev A, et al. A comprehensive modular map of molecular interactions in RB/E2F pathway. Mol Syst Biol 2008; 4 : 173. 8. Sherr CJ, McCormick F. The RB and p53 pathways in cancer. Cancer Cell 2002; 2 : 103–12. 9. Kitano H, Funahashi A, Matsuoka Y, Oda K. Using process diagrams for the graphical representation of biological networks. Nat Biotech 2005; 23 : 961–6. 10. Rapaport F, Zinovyev A, Dutreix M, et al. Classification of microarray data using gene networks. BMC Bioinformatics 2007; 8 : 35. 11. Song S, Black MA. Microarray-based gene set analysis: a comparison of current methods. BMC Bioinformatics 2008; 9 : 502. 12. Stransky N, Vallot C, Reyal F, et al. Regional copy number-independent deregulation of transcription in cancer. Nat Genet 2006; 38 : 1386–96. 13. Chen KC, Calzone L, Csikasz-Nagy A, et al. Integrative analysis of cell cycle control in budding yeast. Mol Biol Cell 2004; 15 : 3841–62. 14. Cross FR, Schroeder L, Kruse M, Chen KC. Quantitative characterization of a mitotic cyclin threshold regulating exit from mitosis. Mol Biol Cell 2005; 16 : 2129–38. 15. Zi Z, Klipp E. Constraint-based modeling and kinetic analysis of the Smad dependent TGF-beta signaling pathway. Plos One 2007; 2 : e936. 16. Lipniacki T, Kimmel M. Deterministic and stochastic models of NFkappaB pathway. Cardiovasc Toxicol 2007; 7 : 215–34. 17. Kholodenko BN. Negative feedback and ultrasensitivity can bring about oscillations in the mitogen-activated protein kinase cascades. Eur J Biochem 2000; 267 : 1583–8. 18. Kholodenko BN, Demin OV, Moehren G, Hoek JB. Quantification of short term signaling by the epidermal growth factor receptor. J Biol Chem 1999; 274 : 30169–81. 19. Fussenegger M, Bailey JE, Varner J. A mathematical model of caspase function in apoptosis. Nat Biotechnol 2000; 18 : 768–74. 20. Bentele M, Lavrik I, Ulrich M, et al. Mathematical modeling reveals threshold mechanism in CD95-induced apoptosis. J Cell Biol 2004; 166 : 839–51. 21. Radulescu O, Gorban AN, Zinovyev A, Lilienbaum A. Robust simplifications of multiscale biochemical networks. BMC Syst Biol 2008; 2 : 86. 22. Aebersold R, et al. Report on EU-USA workshop: how systems biology can advance cancer research. Mol Oncol 2009 (sous presse). 23. Thomas R, Kaufman M. Multistationarity, the basis of cell differentiation and memory. I. Structural conditions of multistationarity and other nontrivial behavior. Chaos 2001; 11 : 165–79. 24. Goldbeter A. A minimal cascade model for the mitotic oscillator involving cyclin and cdc2 kinase. Proc Natl Acad Sci USA 1991; 88 : 9107–11. 25. Novák B, Tyson JJ. Modelling the controls of the eukaryotic cell cycle. Biochem Soc Trans 2003; 31 : 1526–9. 26. Segal E, Friedman N, Koller D, Regev A. A module map showing conditional activity of expression modules in cancer. Nat Genet 2004; 36 : 1090–8. 27. Subramanian A, Tamayo P, Mootha VK, et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proc Natl Acad Sci USA 2005; 102 : 15545–50. 28. Kitano H. Towards a theory of biological robustness. Mol Syst Biol 2007; 3 : 137. 29. Alon U. An introduction to systems biology: design principles of biological circuits. Mathematical and computational biology. London : Chapman and Hall/CRC, 2006. 30. Wagner A. Robustness and evolvability in living systems. Princeton: Princeton University Press, 2005. 31. Harbour JW, Dean DC. the Rb/E2F pathway: expanding roles and emerging paradigms. Genes Dev 2000; 14 : 2393–409. 32. Yao G, Lee TJ, Mori S, et al. A bistable Rb-E2F switch underlies the restriction point. Nat Cell Biol 2008; 10 : 476–82. |