| |
| Med Sci (Paris). 2009 August; 25(8-9): 705–712. Published online 2009 August 15. doi: 10.1051/medsci/2009258-9705.Variations génétiques et environnementales dans un réseau de signalisation intercellulaire Marie-Anne Félix* Institut Jacques Monod, CNRS-Université Paris 7, Denis Diderot, 15, rue Hélène Brion, 75205 Paris Cedex 13, France |
Importance de la variation en biologie Les biologistes de laboratoire cherchent le plus souvent à réduire la variabilité dans leurs expériences, se désintéressant de la variation du système qu’ils étudient. Néanmoins, la variance, et plus précisément la distribution des variables, sont des caractéristiques du système, au même titre que les valeurs moyennes de ces variables. Par exemple, si une pathologie apparaît à partir d’un seuil dans la concentration d’une molécule, la distribution de la variation de cette concentration est cruciale pour caractériser le système. La variation entre individus est aussi la base de l’évolution. Dans la variation entre individus on distingue : (1) les variations génétiques héréditaires ; (2) les variations environnementales qui forment le contexte écologique dans lequel les organismes se développent et sont sélectionnés ; (3) les variations stochastiques (ou microenvironnementales), que l’on peut révéler lors du développement d’individus génétiquement identiques dans le même environnement [
1,
2]. En ce qui concerne la population humaine, la diversité des conditions de vie et la diversité génétique contribuent conjointement à la variation interindividuelle au cours de l’évolution de l’espèce et dans le contexte médical. |
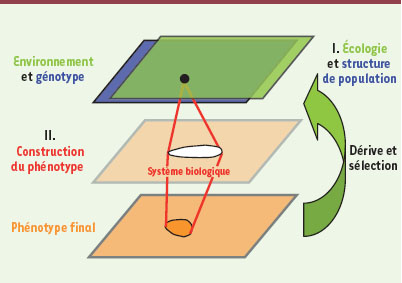
Relations entre génotype, environnement et phénotype Les systèmes biologiques s’articulent autour de la relation causale entre génotype et environnement d’une part, et phénotype d’autre part [
3–
5] (Figure 1). 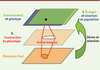 | Figure 1.
Génotype, environnement, phénotype intermédiaire, phénotype final. Schéma simplifié représentant des espaces de paramètres du génotype (bleu), de l’environnement (vert) et du phénotype final (orange). L’écologie et la structure des populations déterminent la position d’un organisme en développement dans l’espace des paramètres génétiques et environnementaux. La construction du phénotype final passe par un système biologique (interactions cellulaires, expression de gènes, etc.) qui peut lui-même être représenté par un espace de paramètres (phénotype intermédiaire ou endophénotype, saumon). Un phénotype intermédiaire peut par exemple être la position respective de cellules, ou la concentration d’une molécule. En une boucle de rétroaction, le phénotype final influe sur l’écologie et la structure génétique de la génération suivante, en particulier via la sélection naturelle. |
-
Le génotype, ou génome, est l’ensemble du matériel génétique.
-
Le phénotype est tout ce qui « apparaît », que ce soient la concentration d’une molécule, les assemblages de molécules, l’état du métabolisme d’une cellule, son mouvement, son adhésion à d’autres cellules, les signaux entre cellules, la morphologie d’un organisme, ou son comportement. La visibilité et l’étude d’un phénotype dépendent des méthodes employées pour le révéler.
La construction de tout phénotype dépend conjointement de l’environnement et du génotype (Figure 1). Comme exemples de dépendance de l’environnement, à l’extrême une mouche drosophile ne peut se développer à 100°C, et plus subtilement la taille de la mouche adulte dépend des conditions de nourriture de la larve. Comme exemples de dépendance du génotype, une mouche ne se développerait pas sans ADN, et des mutations de cet ADN peuvent produire des changements de phénotype. Le rôle de l’environnement dans le phénotype n’est pas du tout une curiosité de laboratoire mais est très fréquemment observé dans la nature. Au cours du développement des plantes, la lumière, la température ou l’humidité sont des facteurs clés pour la germination, la définition de la forme des branchements ou des racines, ou la floraison. Chez les animaux, le développement est généralement moins variable, mais cette plasticité du phénotype final en fonction des conditions de développement existe aussi. Un exemple extrême est celui des abeilles pour lesquelles la spécialisation en reine ou en ouvrière dans une ruche est le résultat d’une nutrition différente de femelles génétiquement identiques. De manière plus générale, environnement et génotype influent tous deux sur un grand nombre de phénotypes, tels que le niveau d’expression des gènes [
6]. La distribution de la variation dans un phénotype dépend beaucoup des conditions d’étude : telle population pour la variation génétique, un ensemble d’environnements pour la variation environnementale, un fonds génétique et un environnement déterminés pour la variation stochastique. Les études de l’influence d’un facteur génétique ou environnemental dépendent donc de façon cruciale de la population étudiée et des environnements dans lesquels cette population se trouve [3, 6,
7]. Par exemple, l’influence de tel facteur génétique peut être détectée dans une population et non dans une autre, soit parce qu’une certaine variation génétique est absente de la seconde, soit parce que les variables environnementales des deux populations diffèrent. Le phénotype influence ensuite la survie et la reproduction différentielle des organismes, ce qui forme le mécanisme de la sélection naturelle (Figure 1). L’évolution opère principalement par des variations de gènes ; par conséquent, la génétique est particulièrement importante pour comprendre l’évolution. Cette dernière dépend aussi de variations de l’environnement. L’apparition de l’oxygène dans l’atmosphère terrestre par exemple a permis la respiration ; la nutrition joue un rôle dans l’évolution récente du poids chez l’homme. En revanche, l’environnement ne modifie pas de façon héréditaire le matériel génétique. Il a néanmoins un rôle dans le processus de sélection des organismes car leur capacité de survie et de reproduction dépend de l’environnement dans lequel ils se trouvent, notamment des conditions de températures ou de nutrition. Enfin, l’organisme peut d’une part choisir son environnement et d’autre part le modifier par son comportement, ce qui a pour résultat une évolution de l’environnement et ainsi des conditions de développement de l’organisme (dans le cadre de l’évolution culturelle humaine, l’évolution de l’environnement est ainsi pratiquement devenue indépendante de l’évolution génétique). |
Épistasie génétique et environnementale dans la construction d’un phénotype Les phénotypes ne se construisent pas suivant les instructions d’un gène mais par l’assemblage et la dynamique de nombreuses molécules, protéines et cellules. Il résulte que l’effet sur le phénotype d’une variation ou d’une mutation dans un gène dépend : (1) de l’état des autres gènes dans le même génome ; (2) de l’environnement dans lequel est construit le phénotype [5, 7,
8]. Cette dépendance s’appelle aussi épistasie. On la détecte par une interaction non additive1 entre l’effet de plusieurs variations génétiques ou de paramètres environnementaux sur la variance du phénotype [
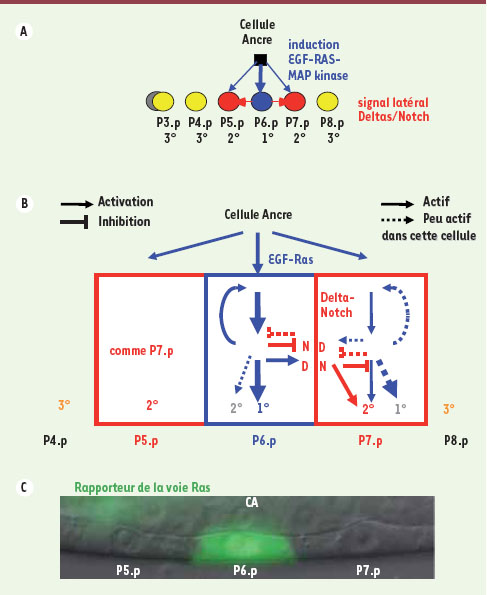
9]. Épistasie génétique Tout d’abord, l’effet d’une variation au niveau d’un locus dépend de l’état allélique des autres gènes. Prenons l’exemple de la formation de la vulve, l’organe de ponte chez le ver nématode Caenorhabditis elegans, qui est un système modèle de développement basé sur un réseau de signalisations intercellulaires bien connu au niveau moléculaire [
10]. Schématiquement, six cellules précurseurs sont alignées dans l’épiderme ventral. La cellule ancre de la gonade 2 envoie un signal EGF ( epidermal growth factor) relayé par une cascade Ras-MAP kinase dans ces cellules précurseurs ; la cellule précurseur P6.p, située généralement le plus près de la cellule ancre, adopte une destinée vulvaire centrale ou primaire et exprime des ligands de type Delta (ligands des récepteurs Notch) ; ces ligands induisent la destinée secondaire dans les deux cellules voisines et y inhibent la voie Ras via la voie Notch (Figure 2). Soit deux gènes a et b, et deux formes variantes (alléliques) de chaque gène a1 et a2, b1 et b2. On peut trouver des exemples de combinaison d’allèles tels que le ver de génotype a1b1 ou a2b1 a une vulve normale, celui de génotype a2b1 n’a pas assez de différenciation vulvaire (moins de trois cellules précurseurs) et celui de génotype a2b2 un excès de différenciation vulvaire (plus de trois cellules) (Figure 3). Dans ce cas, l’effet de la mutation de a1 en a2 dépend fortement du génotype au gène b. Par exemple, une même mutation du récepteur à l’EGF réduit le nombre de cellules induites quand le gène gap-1 est sauvage, mais l’augmente quand le gène gap-1 est inactivé par une mutation nulle (Figure 3A) [
11]. Pour expliquer ce phénomène, on peut avancer l’hypothèse de la titration du ligand EGF par le récepteur. La mutation du récepteur à l’EGF appelée let-23(sy1) diminue la quantité de molécules de récepteurs à la surface des cellules. Dans un contexte par ailleurs sauvage, cette mutation diminue la signalisation dans les cellules centrales, mais quand la voie de signalisation est rendue hypersensible par une inactivation de l’inhibiteur gap-1, la diminution du dosage du récepteur permet à l’EGF d’agir à plus grande distance, activant ainsi la différenciation vulvaire de cellules additionnelles. On obtient ainsi un effet contraire de la même mutation du récepteur suivant l’état d’autres gènes.  | Figure 2.
Réseau de signalisation intercellulaire dans la spécification des cellules précurseurs de la vulve de Caenorhabditis. A. Ce système modèle de développement fait intervenir une rangée de cellules précurseurs compétentes pour former du tissu vulvaire : les cellules P3.p à P8.p chez C. elegans. Chez la plupart des animaux sauvages, P6.p adopte une destinée primaire (bleu, partie centrale de la vulve), P5.p et P7.p une destinée secondaire (rouge, parties latérales de la vulve). Les autres cellules adoptent une destinée épidermale non-vulvaire (jaune). Ce motif spatial de destinées cellulaires apparaît sous l’action d’un signal inducteur de la cellule ancre de l’utérus, qui sécrète un signal LIN-3 de type EGF (flèches bleues). L’activation du récepteur à l’EGF dans P6.p induit la transcription de plusieurs ligands de Notch de type Delta (flèches rouges) qui activent la destinée secondaire dans les cellules voisines et y inhibent la voie Ras [10]. La destinée de P(4-8).p est pratiquement invariante (voir l’article), alors que celle de P3.p est soumise à des variations stochastiques : P3.p peut adopter une destinée tertiaire ou ne pas se diviser et sortir précocément du groupe de compétence (gris) [18, 29]. B. Schéma du réseau moléculaire comprenant des boucles de rétroaction et des interactions multiples entre les voies Ras (en bleu) et Notch (en rouge). Le même réseau moléculaire est présent dans toutes les cellules, mais les différentes voies sont différentiellement activées sous l’influence de la cellule ancre (généralement près de P6.p). Les flèches en pointillés indiquent un faible niveau d’activation de la branche considérée. N : Notch ; D : Delta. C. Image en optique Nomarski et épifluorescence montrant le niveau du rapporteur transcriptionnel de la voie Ras egl-17 couplé à la GFP dans P5.p, P6.p et P7.p dans une larve de troisième stade de Caenorhabditis briggsae, espèce voisine de C. elegans. Noyau et nucléole sont visibles à l’intérieur de chaque cellule. CA : cellule ancre. |
 | Figure 3.
Exemples d’épistasies génétique et environnementale. A. Épistasie génétique entre des variations à deux locus a et b. L’état allélique (1 ou 2) au locus b interagit avec celui au locus a. Ceci correspond à l’exemple de la mutation let-23(sy1)/EGFR pour le locus a et gap-1(ga133)/GAP pour le locus b [11]. I = nombre de cellules Pn.p induites (± erreur standard). Le code de couleur des destinées cellulaires est le même que dans la Figure 2. Les variations stochastiques de destinées cellulaires entre individus sont représentées schématiquement : la destinée prédominante par un disque, une destinée de moindre fréquence par un croissant à gauche. B. Épistasie entre la variation à un locus a et le fonds génétique naturel d’une souche. L’exemple correspond à la mutation let-23(sy1) dans les fonds génétiques sauvages N2 et AB1 de C. elegans [12]. C. Épistasie entre la variation à un locus c et l’environnement. L’exemple correspond à la mutation lin-3(n378)/egf en environnement standard (nourris) et en carence nutritionnelle pendant le développement [15]. |
Ce que nous venons de voir avec la variation d’un gène b s’applique aussi en comparant l’effet d’une variation du gène a dans le contexte de génotypes naturels qui diffèrent à des milliers d’autres endroits du génome. L’effet de la mutation d’un gène dépend ainsi de l’ensemble du fonds génétique sauvage. La mutation let-23(sy1) a par exemple un effet important pour les vers ayant le fonds génétique standard de laboratoire (N2), et presque pas d’effet s’ils présentent un autre fonds génétique sauvage (AB1) (Figure 3B). À l’inverse, une mutation dans une autre voie moléculaire (Wnt) a un effet faible dans le fonds génétique standard (et pour cette raison, n’avait pas été isolée dans les cribles pour les mutants de la vulve de C. elegans), et plus fort dans d’autres fonds génétiques [
12]. Cet effet du fonds génétique se retrouve chez tous les organismes : par exemple chez la drosophile ou la souris, une mutation de l’EGFR aura des conséquences différentes en fonction du fonds génétique [
13,
14]. Chez l’homme, la connaissance d’un génotype à un locus peut ainsi suggérer des fréquences statistiques de développement d’une maladie dans une population donnée, mais l’effet de ce variant dépend de variations au niveau d’autres locus. Épistasie entre le génotype et l’environnement Le second facteur qui influence l’effet d’un variant génétique est l’environnement (Figure 3C). Dans l’exemple de la vulve de C. elegans, une souche portant une mutation inactivatrice d’un régulateur négatif léger de la voie Ras développe une vulve normale si elle est placée dans l’environnement standard, mais anormale quand on place les vers dans un autre environnement [
15]. L’absence d’effet observé d’une inactivation génique dans un environnement standard de laboratoire est très commune [
16]. Le maintien de ce régulateur au niveau évolutif est permis si les organismes rencontrent dans la nature un environnement qui conduit à un phénotype anormal (même à faible fréquence) si ce régulateur est muté. Dans ce cas précis, le paramètre de l’environnement qui a été modifié est la quantité de nourriture. Plus encore que le génotype, l’environnement est multifactoriel et il est possible que maints autres changements environnementaux produisent le même effet. La détermination des paramètres environnementaux importants pour l’évolution du génome requiert donc des études écologiques qui font particulièrement défaut pour les espèces modèles de laboratoire. De façon plus générale, différents paramètres environnementaux peuvent interagir entre eux comme le font différents variants géniques : par exemple, différents traitements médicamenteux entre eux, ou un médicament et un régime alimentaire. Le génotype et l’environnement influencent un ensemble commun de paramètres L’activité d’une protéine peut être modifiée par une variation du gène qui la code, ou par celle de gènes codant des répresseurs ou des activateurs de ce premier gène/protéine. Mais les mêmes paramètres d’activité de cette protéine peuvent aussi être modifiés par l’environnement, par exemple la température ou la nourriture, qui peuvent aussi conduire à l’activation de la protéine, ou à celle de répresseurs ou d’activateurs de cette protéine. Génotype et environnement peuvent donc en principe pareillement influencer le phénotype (Figure 3). Nous manquons cependant d’études détaillées de ce phénomène au niveau moléculaire. Des variations environnementales et génétiques naturelles peuvent ainsi avoir le même effet sur le phénotype. Par exemple, chez l’espèce de papillon africain Bicyclus anynana, la coloration des ocelles (tache arrondie) sur les ailes diffère selon que le papillon s’est développé pendant la saison sèche ou pendant la saison humide, et l’on peut trouver cette même différence de coloration sous la forme de variants génétiques dans la population [
17]. Comprendre la part de l’environnement dans la variation du phénotype a beaucoup d’importance dans le contexte de notre relation à notre propre génome. La chose nous paraît évidente si on parle de notre poids : la nutrition et le mode de vie d’une part (variables environnementales), et des variants génétiques présents dans les populations humaines d’autre part, peuvent avoir un effet. Mais on l’oublie souvent quand on parle de soi-disant « gènes de maladie ou de comportement ». Même pour des maladies comme des cancers qui sont évidemment sous l’influence de facteurs environnementaux et génétiques, nous avons une fâcheuse tendance à nous concentrer sur une seule variable à la fois. Cela nuit à la compréhension par le public des facteurs de la susceptibilité à une maladie [7]. |
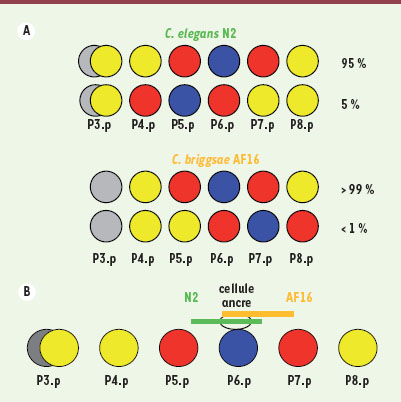
Robustesse d’un système et déviations rares dans le phénotype final Dans le souci de minimiser la variation, les systèmes modèles de laboratoire sont choisis parce qu’ils sont peu sensibles (ou robustes) à des variations stochastiques et environnementales souvent non contrôlables. C’est le cas du développement de la vulve chez C. elegans. L’invariance du système est importante, y compris au niveau cellulaire : on retrouve les mêmes cellules d’un ver à l’autre, avec les mêmes destinées développementales [
18–
20]. Bien que la robustesse soit une propriété des systèmes biologiques abondamment invoquée ces dernières années, elle est rarement mesurée. De plus, les types de variants sont rarement étudiés - s’ils le sont jamais -, sauf pour des variations quantitatives autour d’une moyenne. Pourtant, ces variants donnent de nombreuses informations sur le système étudié. Dans l’exemple du développement de la vulve de C. elegans, un variant relativement fréquent lorsque des conditions de carence nutritionnelle sont imposées à la souche N2 modifie le centrage du motif de destinées cellulaires vulvaires : au lieu que le motif soit centré sur la cellule P6.p, il l’est sur la cellule immédiatement antérieure, P5.p (Figure 4). Dans l’espèce proche, Caenorhabditis briggsae, les variations de centrage s’observent plutôt du côté postérieur sur P7.p [15]. De telles variations requièrent un nombre de cellules compétentes supérieur aux trois cellules précurseurs qui forment effectivement le tissu vulvaire dans ces espèces. L’étude de l’induction de variants par les modifications de l’environnement et de leur évolution est donc une riche source d’informations sur les systèmes aux niveaux mécanistique et évolutif. Par ailleurs, le caractère stochastique de ces variants rares doit être souligné : la fréquence à laquelle un variant apparaît parmi des individus dont le génotype et le macro-environnement sont fixés est très faible.  | Figure 4.
Exemples de déviations dans le motif spatial de destinées des cellules précurseurs de la vulve. A. Un variant fréquent chez C. elegans N2 placé dans un environnement de carence alimentaire est le centrage du motif vulvaire sur P5.p au lieu de P6.p (environ 5 % des individus). On ne trouve pas ce variant chez C. briggsae AF16, mais plutôt un centrage sur P7.p (à très faible fréquence dans la souche AF16, à plus forte fréquence dans d’autres souches sauvages de C. briggsae) [15]. Le code de couleur des destinées cellulaires est le même que dans la Figure 2. P3.p n’est pas compétent chez C. briggsae [29]. B. La position de la cellule ancre par rapport aux cellules Pn.p varie de manière stochastique d’un individu à l’autre. La distribution (schématisée par les traits verts et orange) est déplacée vers le côté antérieur (P5.p) chez C. elegans N2, vers le côté postérieur chez C. briggsae AF16. |
On peut étudier l’effet de variations génétiques spontanées sur la robustesse du système à l’aide de lignées où est permise l’accumulation de mutations spontanées dans des conditions où ces dernières sont soumises à une sélection minimale. Pour construire ces lignées, un seul ver juvénile est transféré sur une nouvelle boîte à chaque génération [
21]. On introduit ainsi un goulot d’étranglement de population qui minimise la sélection contre des mutations délétères qui se produit inévitablement lorsque les boîtes de culture abritent des populations plus importantes. Au-delà de 400 générations, les lignées maintenues en parallèles ont accumulé de nombreuses mutations spontanées. Chacune d’entre elles abrite plus de mutations qu’après une mutagenèse chimique de laboratoire [
22]. L’éventail de phénotypes obtenus est représentatif de ce qu’il advient en l’absence de sélection. La comparaison entre les phénotypes de ces lignées de laboratoire et ceux de lignées sauvages (plus divergentes entre elles génétiquement) démontre l’action de la sélection sur le système [
23]. Par exemple, les génotypes qui favorisent les variations de centrage de la vulve sont contre-sélectionnés dans les populations naturelles [15]. Nous pouvons faire l’hypothèse que ce décentrage allié à une autre déviation observée, par exemple une perte de cellule Pn.p ou une perte de leur compétence, conduit à une vulve incomplète. Cette dernière ne permet pas la ponte des œufs et la lignée est contre-sélectionnée. Que se passe-t-il au cours du développement de ces individus variants ? Pour le centrage de la vulve, la variation semble provenir d’une distribution variable de la position relative de la cellule ancre inductrice et des cellules Pn.p. La cellule ancre est placée à sa naissance dans différentes positions selon les individus d’une même souche, avec un biais vers P5.p chez C. elegans N2 et vers P7.p chez C. briggsae (Figure 4) [15]. L’évolution des rares variations de centrage indique ainsi une évolution dans le système de développement sous-jacent, alliée à une variation stochastique entre individus. Nous allons maintenant étudier d’autres manières de mettre en évidence des variations dans le système. |
Variations cryptiques dans la construction du phénotype De même que les variants dans le produit final de développement (qui réprésente le phénotype le plus apparent du système) d’un système ont été peu étudiés, on minimise aussi la variation qui existe pendant le processus de construction de ce phénotype final lorsqu’on schématise ce processus développemental. Pourtant, même dans les systèmes où le taux de variants est faible pour le produit final, d’amples variations peuvent exister dans le processus de construction du phénotype. Ces variations qui n’affectent pas le produit final, sont appelées « cryptiques » [
24]. En revanche, elles ne sont pas cryptiques si on emploie des méthodes pour révéler un phénotype « intermédiaire » (ou endophénotype) au cours de la construction du phénotype final (Figure 1) [
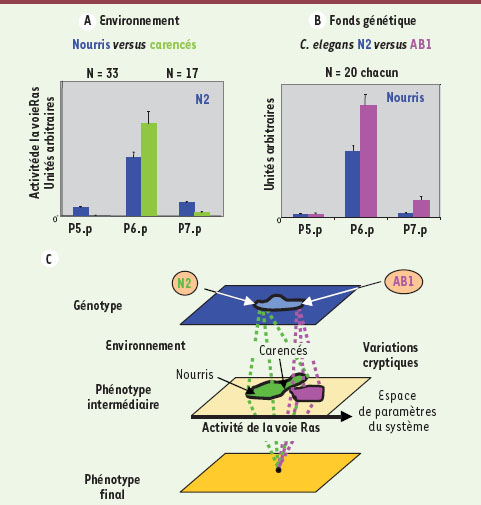
25]. Le premier type de méthode pour révéler des variations cryptiques dans un système invariant est de déplacer le système hors de la zone où les variations sont « tamponnées » [24]. On peut utiliser par exemple des mutations ou d’autres manipulations expérimentales contrôlées. Tout d’abord, les variations sous l’effet du bruit stochastique sont facilement révélées par l’augmentation de variance dans le produit du système. Par exemple, une souche isogénique portant une mutation qui inactive partiellement le récepteur à l’EGF présente une distribution d’index vulvaire allant de zéro à trois cellules induites. La variation interindividuelle dans le phénotype de mutants est très habituelle et bien connue des généticiens, mais rarement étudiée en tant que telle, c’est-à-dire comme conséquence de variations stochastiques non génétiques dans la construction du phénotype. Outre les variations stochastiques, ces mutations ou autres manipulations expérimentales peuvent révéler des variations cryptiques quand elles sont introduites dans différents fonds génétiques sauvages ou environnementaux. Par exemple, l’introduction du mutant du récepteur à l’EGF (1) dans différentes souches sauvages de C. elegans [12] ou (2) dans différents environnements de développement [15] révèle respectivement des variations génétiques et environnementales (voir plus haut à propos de l’épistasie ; Figure 3B, C). Le second type de méthode utilisé pour révéler une variation cryptique consiste à directement mesurer un phénotype « intermédiaire » (ou endophénotype) du système, comme la position de la cellule ancre (voir ci-dessus ; Figure 4B) ou le niveau d’activation d’une voie moléculaire (Figure 5). Dans le cas de la vulve, on peut quantifier l’expression de rapporteurs transcriptionnels des voies de signalisation EGF-Ras-MAP kinase et Delta-Notch. On peut ainsi révéler des différences entre des souches sauvages de C. elegans dans des conditions où le phénotype final est de type sauvage pour toutes ces souches (Figure 5).  | Figure 5.
Variation cryptique d’activité de la voie Ras dans différentes conditions, environnements et fonds génétiques sauvages. A. Activité de la voie Ras dans les cellules P5.p à P7.p dans la souche N2 de C. elegans placée dans des conditions d’environnement « vers nourris » (bleu) et « vers carencés » (vert), mesurée par quantification de fluorescence d’un rapporteur transcriptionnel egl-17 couplé à la CFP (moyenne + erreur standard). Alliée à des données telles que celles représentées sur la Figure 3C, cette quantification suggère une plus forte activation de la voie Ras dans un environnement carencé qui supprime des mutations hypomorphes de cette voie [ 15]. B. Activité de la voie Ras dans les fonds génétiques sauvages N2 (bleu) et AB1 (violet) dans des conditions d’environnement « vers nourris ». Associée à des données telles que celles représentées dans la Figure 3B, cette quantification suggère une plus forte activation de la voie Ras dans la souche AB1 que chez N2, qui compense des mutations hypomorphes de cette voie [ 12]. C. Schéma reprenant la représentation de la Figure 1, résumant la variation cryptique dans le système. L’activité de la voie Ras (A, B) est un phénotype intermédiaire variant dans le système en fonction de l’environnement et du fonds génétique sauvage, alors que le phénotype final (le nombre de cellules Pn.p induites) ne varie pas. |
Un exemple simple de variation cryptique est donné par la comparaison entre les souches sauvages de C. elegans N2 et AB1. Toutes deux présentent un phénotype final similaire de destinées vulvaires. Cependant, quand des mutations identiques sont introduites en parallèle dans les deux fonds génétiques, l’index vulvaire est toujours plus élevé dans le fonds génétique AB1. Lors d’expériences d’ablation au laser de la cellule ancre, le motif final de trois cellules induites apparaît plus tôt chez AB1 que chez N2. Enfin, le rapporteur transcriptionnel de la voie Ras a une activité plus élevée chez AB1 que chez N2 [12]. Ces éléments concordent pour indiquer une plus forte activité de la voie Ras chez AB1, qui supprime l’effet des réductions d’activité (il n’y a pas de perte totale) dans la voie et amplifie celui des gains d’activité. La comparaison entre les souches N2 et CB4856 offre un exemple plus complexe. Dans ce cas, plusieurs variations, dont certaines sont sans doute compensatoires, apparaissent à différents niveaux de la voie Ras et entre différentes branches du réseau obtenu avec la voie Notch [12]. Dans les deux cas, les variations cryptiques que l’on peut observer sont « tamponnées » par le système. Pour cela, point n’est besoin de régulations sophistiquées. Dans une certaine mesure, un système de spécification cellulaire tel que celui du développement de la vulve peut s’accommoder de variations et cependant produire le même motif final. L’étude in silico de la robustesse et des variations possibles d’un système donne alors beaucoup d’informations [
26,
27]. |
Cette revue a tenté de mettre en valeur l’importance de l’étude de la variation d’un système biologique et de son évolution. L’utilisation d’organismes modèles de laboratoire permet une approche assez exhaustive et contrôlée, avec des implications en médecine. De façon générale, les effets de l’environnement et du génotype sont intrinsèquement liés lors de la construction du phénotype (sain ou malade). De plus, la variation cryptique peut être révélée, en particulier par un changement environnemental. Une hypothèse récente propose ainsi que de nombreuses maladies humaines soient apparues récemment avec une fréquence élevée en raison de changements environnementaux (mode de vie) qui poussent le système hors de la zone tamponnée, zone où des variants génétiques ne se traduisent pas par un phénotype pathologique [
28]. |
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Je remercie C. Braendle et J. Milloz dont le travail est rapporté ici. L’équipe est financée par le CNRS, l’ANR et l’ARC.
|
Footnotes |
1. Ancel Meyers L, Bull JJ. Fighting change with change: adaptove variation in an uncertain world. TREE 2002; 17 : 551–7. 2. Braendle C, Milloz J, Félix, MA. Mechanisms and evolution of environmental responses in Caenorhabditis elegans. Curr Top Dev Biol 2008; 80 : 171–207. 3. Lewontin RC. The analysis of variance and the analysis of causes. Am J Hum Genet 1974;26 : 400–11. 4. Maynard Smith J. Evolutionary genetics. Oxford : Oxford University Press, 1989 : 326 p. 5. Schlichting CD, Pigliucci M. Phenotypic evolution: a reaction norm perspective. Sunderland, MA : Sinauer Associates, 1998 : 388 p. 6. Gibson G. The environmental contribution to gene expression profiles. Nat Rev Gen 2008; 9 : 575–81. 7. Lewontin R. The triple helix-gene, organism, and environment. Boston : Harvard University Press, 2000 : 130 p. 8. West-Eberhard MJ. Developmental plasticity and evolution. Oxford : Oxford University Press, 2003 : 720 p. 9. Lynch M, Walsh, B. Genetics and analysis of quantitative traits. Sunderland, MA : Sinauer Associates, 1998 : 804 p. 10. Sternberg PW. Vulval development. The C. elegans research community ( http://www.wormbook.org/). Wormbook 2005; 25 juin : 1–28. 11. Hajnal A, Whitfield CW, Kim SK. Inhibition of Caenorhabditis elegans vulval induction by gap-1 and by let-23 receptor tyrosine kinase. Genes Dev 1997; 11 : 2715–28. 12. Milloz J, Duveau F, Nuez I, Félix MA. Intraspecific evolution of the intercellular signaling network underlying a robust developmental system. Genes Dev 2008; 22 : 3064–75. 13. Dworkin I, Palsson A, Birdsall K, Gibson G. Evidence that Egfr contributes to cryptic genetic variation for photoreceptor determination in natural populations of Drosophila melanogaster. Curr Biol 2003; 13 : 1888–93. 14. Threadgill DW, Dlugosz AA, Hansen LA, et al. Targeted disruption of mouse EGF receptor: effect of genetic background on mutant phenotype. Science 1995; 269 : 230–4. 15. Braendle C, Félix MA. Plasticity and errors of a robust developmental system in different environments. Dev Cell 2008; 15 : 714–24. 16. Tautz D. A genetic uncertainty problem. Trends Genet 2000; 16 : 475–7. 17. Brakefield PM, Gates J, Keys D, et al. Development, plasticity and evolution of butterfly eyespot patterns. Nature 1996; 384 : 236–42. 18. Sulston J, Horvitz HR. Postembryonic cell lineages of the nematode Caenorhabditis elegans. Dev Biol 1977; 56 : 110–56. 19. Félix MA. Un ver, 959 cellules et 13 000 gènes. Med Sci (Paris) 1997;13 : 156–65. 20. Félix MA, Labouesse M, Ségalat L. Caenorhabditis elegans. Un organisme modèle en biologie. Paris : Hermann, 2002 : 194 p. 21. Vassilieva LL, Hook AM, Lynch M. The fitness effects of spontaneous mutations in Caenorhabditis elegans. Evolution 2000; 54 : 1234–46. 22. Denver DR, Morris K, Lynch M, Thomas WK. High mutation rate and predominance of insertions in the Caenorhabditis elegans nuclear genome. Nature 2004; 430 : 679–82. 23. Dichtel-Danjoy ML, Félix MA. Phenotypic neighborhood and micro-evolvability. Trends Genet 2004; 20 : 268–76. 24. Gibson G, Dworkin I. Uncovering cryptic genetic variation. Nat Rev Genet 2004; 5 : 681–90. 25. Félix MA, Wagner A. Robustness and evolution: concepts, insights and challenges from a developmental model system. Heredity 2008;100 : 132–40. 26. Von Dassow G, Meir E, Munro EM, Odell GM. The segment polarity network is a robust developmental module. Nature 2000; 406 : 188–92. 27. Giurumescu CA, Sternberg PW, Asthagiri AR. Intercellular coupling amplifies fate segregation during Caenorhabditis elegans vulval development. Proc Natl Acad Sci USA 2006; 103 : 1331–6. 28. Gibson G. Decanalization and the origin of complex disease. Nat Rev Gen 2009; 10 : 134–40. 29. Delattre M, Félix MA. Polymorphism and evolution of vulval precursor cell lineages within two nematode genera, Caenorhabditis and Oscheius. Curr Biol 2001; 11 : 631–43. |