| |
| Med Sci (Paris). 2010 February; 26(2): 193–200. Published online 2010 February 15. doi: 10.1051/medsci/2010262193.Comprendre la mutagenèse somatique grâce à la cartographie des dommages à l’ADN Sandrine Lacoste,1,2 Patrick J. Rochette,1,3 and Régen Drouin1* 1Service de génétique, Département de pédiatrie, local 1428, Faculté de Médecine et des sciences de la santé, Université de Sherbrooke, 3001, 12e Avenue Nord, Sherbrooke, Québec, J1H 5N4 Canada 2Adresse actuelle : Manitoba Institute of Cell Biology, Cancer Care Manitoba, University of Manitoba, 675 McDermot Avenue, Winnipeg, Manitoba, R3E 0V9 Canada 3Adresse actuelle : Department of Therapeutic Radiology, Yale School of Medicine, 333 Cedar Street, HRT 210, New Haven, CT, 06520-8040 États-Unis |
L’ADN des cellules est constamment endommagé par des stress endogènes comme les espèces oxygénées réactives produites par le métabolisme cellulaire, et par des stress exogènes tels que les ultraviolets (UV) [
1]. Ces altérations peuvent mener à des mutations, c’est-à-dire à des modifications de l’information génétique codée par la molécule. Certains changements dans l’information génétique des cellules somatiques (mutations ponctuelles et/ou réarrangements chromosomiques) sont une cause première de transformation maligne des cellules. L’accumulation de ces altérations dans des gènes importants qui jouent un rôle dans le maintien de l’intégrité cellulaire, tel le contrôle du cycle cellulaire, la réparation et l’apoptose, peut entraîner la multiplication anarchique de cellules anormales et la formation d’une tumeur. Afin d’expliquer la relation qui existe entre les dommages à l’ADN, les mutations et la formation des tumeurs, on se fonde sur la théorie de la mutagenèse somatique énoncée par Thilly en 1983 [
2] puis améliorée par Holmquist et Gao en 1997 [
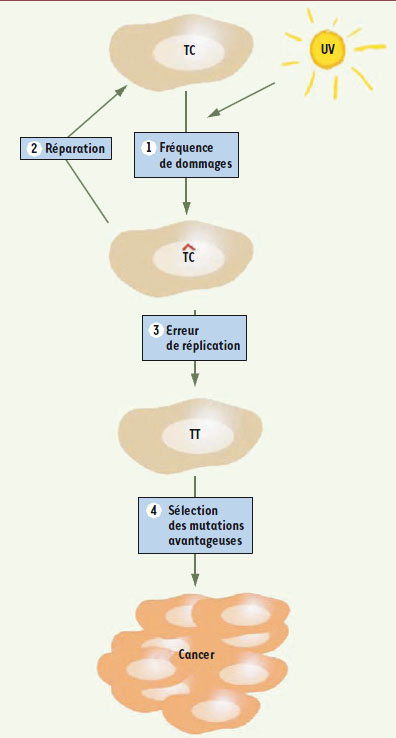
3]. Cette théorie tente de prédire la probabilité qu’a un dommage causé par un agent mutagène d’aboutir à la transformation maligne d’une cellule. Plusieurs facteurs interviennent successivement dans la mutagenèse somatique : (1) la fréquence des dommages, (2) la vitesse de leur réparation, (3) le comportement de la polymérase lors de la réplication de l’ADN endommagé et (4) la sélection des mutations « avantageuses » (Figure 1).
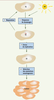 | Figure 1.
Représentation schématique de la théorie de la mutagenèse somatique. La probabilité qu’une mutation donnée soit associée à une transformation maligne donnée suppose : (1) que des dommages en quantité suffisante surviennent à cet endroit (fréquence des dommages) ; (2) qu’ils ne soient pas réparés ou qu’ils soient réparés suffisamment lentement (réparation) pour que (3) la polymérase de réplication de l’ADN ait le temps de venir répliquer cet ADN endommagé, qu’elle interprète mal cette base endommagée et ainsi qu’elle « fixe » la mutation (en incorporant un mauvais nucléotide en face de la base endommagée) (erreur de réplication) et finalement (4) que cette mutation influe sur le phénotype cellulaire dans une fonction associée à la transformation maligne (sélection des mutations « avantageuses »). |
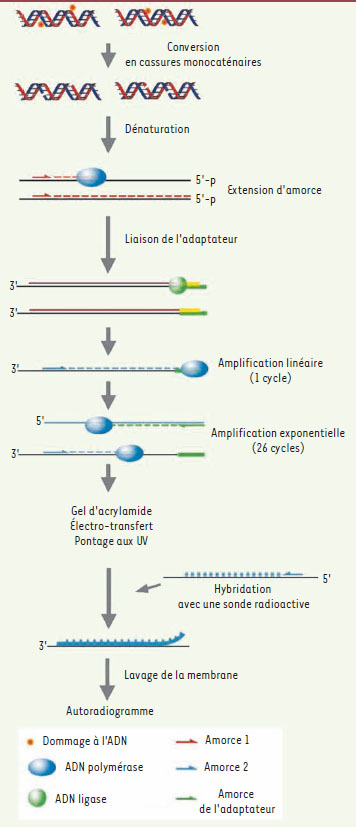
Il est donc fondamental de pouvoir mieux situer ces dommages (c’est-à-dire quelles sont les positions nucléotidiques) et mieux savoir à quelle vitesse ils sont réparés pour comprendre leur implication dans la transformation maligne des cellules. La technologie LMPCR (ligation-mediated PCR ou réaction de polymérisation en chaîne où intervient la liaison d’un adaptateur) permet de scruter ces deux paramètres. La technologie LMPCR n’est capable de mettre en évidence qu’un seul type de dommages : les cassures monocaténaires qui ne touchent qu’un seul brin de l’ADN ; il existe cependant des moyens enzymatiques ou chimiques de transformer d’autres types de dommages en cassures et ainsi élargir la gamme de lésions susceptibles d’être explorées (Figures 2 et 3).
 | Figure 2.
Représentation schématique de la technique LMPCR. La technique LMPCR permet d’étudier les cassures monocaténaires (une cassure sur un seul des 2 brins d’ADN). Certains agents lésionnels provoquent des cassures monocaténaires directement. Cependant, comme l’illustre le schéma, la majorité des dommages à l’ADN doivent être convertis de façon enzymatique ou chimique en cassure monocaténaire possédant un phosphate à son extrémité 5’. Les étapes de la technique sont les suivantes. Une extension est réalisée à partir d’une amorce spécifique du gène choisi (amorce 1) et s’arrête à la première cassure rencontrée qui correspond au lieu du dommage initial. L’amorce spécifique offre l’avantage d’un premier degré de spécificité à la technique. Cette première extension d’amorce produit un bout franc sur lequel on vient lier un adaptateur. Il en résulte des fragments d’ADN dont les deux extrémités sont connues et qu’on peut alors amplifier par PCR avec une amorce correspondant à l’adaptateur et une seconde amorce spécifique du gène étudié (amorce 2). Cette seconde amorce est choisie légèrement en aval de la première afin de donner un deuxième degré de spécificité à la technique. Les produits PCR ainsi obtenus comptent une taille et une quantité qui dépendent respectivement de la position nucléotidique des dommages de départ et de leur fréquence initiale à chaque position donnée. Une migration dans un gel d’acrylamide suivie d’un électro-transfert sur une membrane de nylon puis d’une hybridation avec une sonde spécifique radioactive permettent de visualiser l’ensemble des produits PCR. |
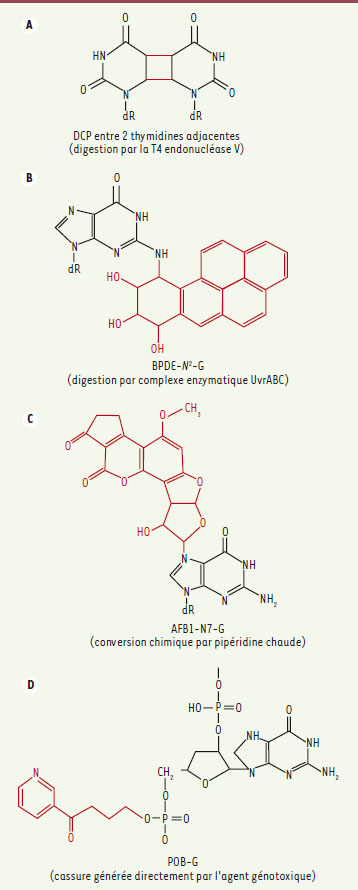
 | Figure 3.
Dommages à l’ADN examinés par LMPCR. Dommages fréquents à l’ADN consécutifs à une exposition (A)aux UV, (B)au BPDE, (C)
à l’AFB1 ou (D)
à un analogue de la NNK et qui ont pu être examinés à l’aide de la technologie LMPCR. La modification provoquée par l’agent génotoxique est indiquée en rouge et le mode de conversion du dommage en cassures monocaténaires pour son étude avec la technologie LMPCR est indiqué entre parenthèses. |
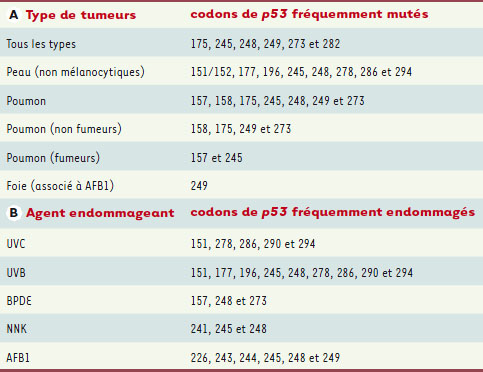
Depuis quelques années, des bases de données recensent les mutations propres aux tumeurs humaines (http://p53.free.fr ; http://www-p53.iarc.fr). Il s’agit essentiellement de mutations dans le gène p53 : ce gène est très fréquemment muté (dans plus de 50 % des tumeurs humaines) et il s’agit de mutations ponctuelles. Mais d’autres gènes comme ras, comptant un nombre plus restreint de mutations connues sont également analysés. Ces bases de données permettent de reconstituer des spectres de mutations indiquant les sites mutés dans un type de tumeur donné en fonction de la position nucléotidique. L’étude des spectres de mutations de p53 dans différents types de tumeurs a permis de faire apparaître des « points chauds » (hot spots) de mutations spécifiques de certains types de cancers, suggérant que ces spectres mutationnels sont en quelque sorte la signature du ou des agents mutagènes à l’origine de la transformation maligne [
4] (Tableau IA). La technologie LMPCR s’est avérée un outil essentiel pour vérifier si ces points chauds de mutations correspondaient effectivement à des positions nucléotidiques fréquemment endommagées et/ou réparées lentement. Ainsi, des liens étiologiques entre certains agents génotoxiques et certains types de cancer ont pu être établis à l’échelle de la structure moléculaire.
 | Tableau I.
A. Points chauds de mutations de p53 en fonction du type de tumeur. B. Positions fréquemment endommagées par les agents génotoxiques cartographiées par LMPCR. |
|
Le cancer de la peau et les différentes longueurs d’ondes des rayons UV Le cancer de la peau a depuis longtemps été associé à l’exposition aux rayons UV du soleil et plus particulièrement aux UVB (280-320 nm) qui, contrairement aux UVC (100-280 nm), ne sont pas arrêtés par la couche d’ozone. Les mutations de p53 les plus fréquentes dans ce type de cancer sont des transitions C → T (une cytosine est remplacée par une thymine) y compris quelques transitions en tandem CC → TT. Ces mutations sont la conséquence de la formation de dimères de pyrimidines (DCP), correspondant à des liaisons covalentes entre deux pyrimidines adjacentes (Figure 3A), après une irradiation UV. Huit points chauds de mutations ont été mis en évidence dans les cancers de la peau : ils sont situés aux codons 151/152, 177, 196, 245, 248, 278, 286 et 294 de la protéine p53 (Tableau IA) [
36]. Une première étude réalisée à l’aide de la LMPCR avec des cellules irradiées aux UVC, soit sous une longueur d’onde proche de celle qui est la mieux absorbée par l’ADN (260 nm), a montré que les points chauds de mutations aux codons 151, 278, 286 et 294 étaient fréquemment endommagés (Tableau IB) [
5]. Une expérience comparable réalisée dans notre laboratoire mais cette fois avec des UVB [
6], a révélé que les 8 points chauds de mutation sont alors fréquemment endommagés, et non plus seulement 4 (Tableau IB). Ainsi, le spectre mutationnel des cancers de la peau correspond mieux aux dommages consécutifs à une exposition aux UVB qu’à ceux dus aux UVC. D’autre part, il semblerait que les couches supérieures de l’épiderme soient suffisantes pour protéger les cellules de l’effet génotoxique des UVC mais pas de celui des UVB [
7]. Ainsi, bien que la longueur d’onde des UVC cause de façon très efficace des dommages à l’ADN, les résultats obtenus grâce à la technologie LMPCR ont permis de montrer qu’ils ne constituent pas le meilleur modèle expérimental pour simuler une exposition solaire de la peau. Les UVA représentent 5,1 % de l’énergie solaire terrestre (contre 0,3 % pour les UVB). On a longtemps considéré que leur nocivité était négligeable par rapport à celle des autres longueurs d’onde d’UV et qu’ils ne pouvaient qu’oxyder indirectement l’ADN via la production d’espèces réactives oxygénées. Or le spectre des mutations créées par une irradiation aux UVA montre une prédominance de transversions T → G [
8] ; ceci s’explique difficilement par les seuls dommages oxydatifs qui ne provoquent habituellement pas ce genre de mutations. Depuis longtemps, la formation de DCP par les UVA avait été soupçonnée ; elle a été détectée certes, mais reste un sujet de controverse [
9]. La cartographie des DCP consécutifs à une irradiation aux UVA réalisée à l’aide de la LMPCR a confirmé la formation préférentielle de DCP aux dipyrimidines TT (Figure 4), ce qui est cohérent avec le spectre mutationnel secondaire aux UVA mis en évidence chez les rongeurs [8,
10]. Ainsi, la cartographie des dommages a permis d’élucider l’origine d’une fraction des mutations provoquées par les UVA. L’usage de doses d’irradiation conformes à celles des rayons solaires tend cependant à confirmer le rôle prédominant des UVB dans l’étiologie des cancers non mélanocytiques [
11]. Toutefois, un point chaud de mutations au codon 600 du gène BRAF a récemment été associé spécifiquement aux cancers mélanocytiques provoqués par le soleil [
12]. Même si plusieurs longueurs d’ondes peuvent entraîner ce type de mutation, la capacité des UVA à atteindre des couches plus profondes de la peau suggère que leur responsabilité dans la formation de mélanomes pourrait être plus considérable qu’anticipé initialement [
13,
37].
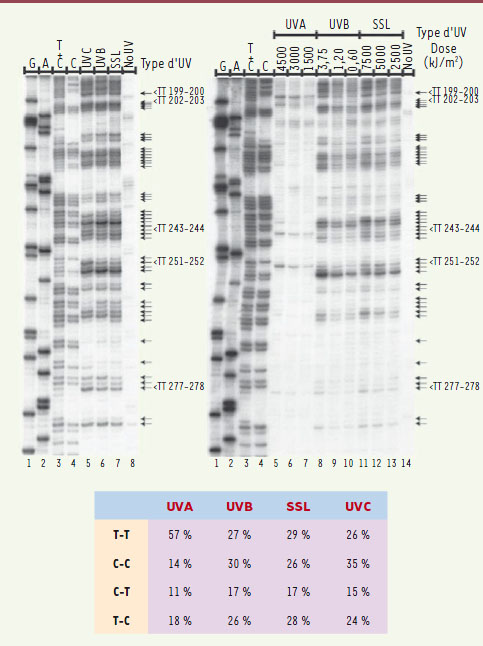
 | Figure 4.
Exemple d’application de la technique LMPCR. Ces autoradiogrammes montrent la cartographie des DPC obtenus sur le gène aprt (adénine phosphoribosyltransférase) après une irradiation aux UVA, UVB, UVC ou à la lumière solaire simulée (SSL). Les quatre premières colonnes de chaque autoradiogramme correspondent à la LMPCR appliquée sur de l’ADN spécifiquement cassé au niveau des G, des A, des T et C ou sur des C seuls. Elles permettent de suivre la séquence sur le gène et donc de situer le lieu des dommages. Chacune des flèches à droite représente un site dipyrimidinique, c’est-à-dire un site potentiel de DCP. Les bandes sur l’autoradiogramme correspondent à des sites effectivement endommagés. Leur intensité dépend du nombre de molécules d’ADN au départ qui étaient cassées à cette position nucléotidique. La colonne identifiée « NoUV » correspond à la LMPCR faite sur de l’ADN non irradié. Elle sert de contrôle négatif et constitue un indicateur du bruit de fond produit par la technique. On peut remarquer que les UVA provoquent presque exclusivement leurs DCP sur des sites TT alors que les autres longueurs d’ondes sont moins exclusives et endommagent tous les types de dipyrimidines. Le tableau recense les dommages quantifiés à partir de ces deux autoradiogrammes et des autoradiogrammes correspondants au brin opposé (non montrés). La localisation de ces dommages aux TT coïncide avec le spectre de mutations aux UVA précédemment publié [ 8]. (adapté de [ 10]). |
|
Influence de la méthylation des cytosines sur la mutagenèse par les rayons UV La détermination de la fréquence des dommages nucléotidiques a aussi permis de mettre en évidence l’influence de la méthylation des cytosines (Cm) sur la mutagenèse engendrée par les UV. Plusieurs phénomènes concourent à une surreprésentation des Cm dans les sites fréquemment mutés. Le premier phénomène indique que la formation des DCP est plus fréquente aux sites 5’-CCG et 5’-TCG où les cytosines sont méthylées [
14,
15] ; cette observation a été associée au fait qu’il y a plus de DCP formés quand les C sont méthylées, possiblement à cause d’une meilleure absorption directe des UV de la lumière solaire [14, 15]. Le second phénomène accrédite l’idée que la probabilité de désamination spontanée d’une cytosine méthylée, ce qui produit une thymine (T), serait plus grande lorsqu’elle participe à un DCP [
16,
17]. Or, une ADN polymérase qui réplique une molécule d’ADN comportant un DCP désaminé tend à incorporer des adénines en face des DCP, ce qui engendre les transitions C → T ou CC → TT caractéristiques des mutations trouvées dans les tumeurs cutanées [17–
19]. |
Étude de la vitesse de réparation du nucléotide La correspondance entre sites fréquemment endommagés et sites mutés n’est jamais parfaite (Tableau I). Ce constat témoigne évidemment de l’influence des autres étapes de la mutagenèse somatique (Figure 1). À l’aide de la technologie LMPCR, des variations de la vitesse de réparation à chaque site de 1 à 15 (quantifiée en temps nécessaire pour 50 % de réparation) ont pu être relevées entre les sites réparés les plus rapidement et ceux réparés les plus lentement [
20]. Le contexte de séquence aurait donc une influence déterminante sur la vitesse de réparation d’un dommage. On a ainsi déterminé que certains sites fréquemment mutés correspondaient en fait à des sites réparés lentement, notamment les codons 177, 196, 245, 248 et 278 de p53 [
21]. Cependant, la vitesse de réparation des DCP ne peut pas expliquer à elle seule le spectre mutationnel des tumeurs de la peau [
22]. |
Identification de carcinogènes à l’origine du cancer du poumon Le cancer du poumon continue d’être la principale cause de mortalité par cancer [
23]. Il a été associé depuis très longtemps à l’exposition au tabac et, en particulier, à la fumée des cigarettes. Celle-ci ne comprend pas moins de 20 carcinogènes différents susceptibles de provoquer des tumeurs pulmonaires chez des rongeurs [
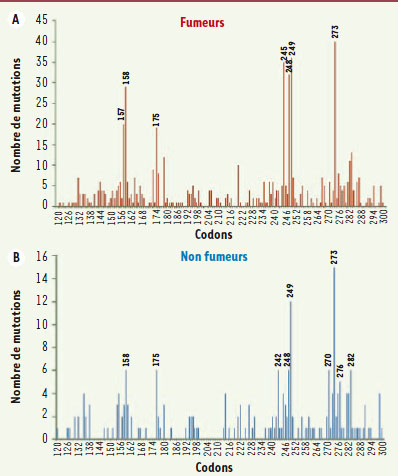
24]. Ces substances carcinogènes sont réparties dans plusieurs classes de composés dont les principales sont les hydrocarbures aromatiques polynucléés (HAP), contenus dans les goudrons de la cigarette, et les nitrosamines qui sont des dérivés de la nicotine. La technologie LMPCR a été utilisée pour cartographier les dommages causés par plusieurs carcinogènes présents dans la fumée du tabac. Le gène p53 est muté dans 70 % des cancers du poumon chez l’homme1,. Dans 40 % des cas, il s’agit de transversions G → T et dans 24 % de transitions G → A [4]. Les codons les plus fréquemment mutés dans les tumeurs pulmonaires associées à la cigarette sont les codons 157, 158, 175, 245, 248, 249 et 273 de p53. Certains sont aussi fréquemment mutés dans les cancers du poumon chez les non fumeurs (codons 158, 175, 249 et 273) tandis que d’autres sont plus spécifiques des cancers associés à la consommation de cigarettes : les codons 157 et 245 (Figure 5 et Tableau IA). Le gène K-ras est l’un des premiers gènes altérés dans le cancer pulmonaire et il est activé par des mutations ponctuelles dans 30 % des adénocarcinomes pulmonaires et des carcinomes à larges cellules [
25]. Les mutations de K-ras semblent particulièrement spécifiques des adénocarcinomes pulmonaires et la plupart affectent le codon 12. Elles sont composées à 80 % de transversions G → T et à 20 % de transitions G → A [25].
 | Figure 5.
Spectre des codons mutés de p53 dans les tumeurs pulmonaires. Les graphiques représentent le pourcentage de mutations ponctuelles recensées dans p53 à un codon donné dans les tumeurs pulmonaires chez des fumeurs (A) ou des non fumeurs (B). Ainsi, on peut déterminer les mutations de p53 qui sont spécifiquement associées à l’exposition à la fumée du tabac (http://www-p53.iarc.fr). |
Une étude par la technologie LMPCR en 1996 [
26] a montré une formation préférentielle dans les cellules traitées au BPDE (la forme activée du benzo[a]pyrène, un des HAP présent dans la fumée de cigarette) de dommages sur des guanines (Figure 3B) dans les points chauds de mutation de p53, en particulier les codons 157, 248 et 273. Les codons 248 et 273 sont fréquemment mutés dans tous les types de cancer, mais le codon 157 est, lui, très spécifique du cancer du poumon chez les fumeurs (Tableau IB et Figure 5). Cette étude correspond à la première démonstration à l’échelle de la structure moléculaire d’un lien étiologique entre un carcinogène donné et le cancer pulmonaire. D’autres HAP de la fumée de cigarette ont montré des profils comparables de dommages, avec des différences cependant concernant leur fréquence relative à une position donnée. En effet, plusieurs autres HAP ont entraîné une formation préférentielle de dommages à des sites correspondant aux points chauds de mutation des codons 157, 158, 245, 248 et 273 de p53 [
27]. Ainsi, les HAP en général semblent contribuer significativement au spectre mutationnel du cancer du poumon chez l’homme. Comme pour les DCP, la vitesse de réparation des dommages du BPDE semble avoir une influence sur la probabilité de formation des mutations. En particulier, la réparation aux points chauds de mutations des codons 157, 248 et 273 s’est avérée 2 à 4 fois plus lente que la réparation dans les autres sites endommagés [
28]. La cartographie des dommages causés par d’autres substances de la fumée de cigarette a également été réalisée à l’aide de la LMPCR. Nous avons montré dans notre laboratoire que des dérivés réactifs de la NNK, un carcinogène pulmonaire prouvé chez les animaux de laboratoire, créent des dommages (Figure 3D) à des codons fréquemment mutés dans le cancer du poumon, notamment le codon 12 de K-ras ainsi que les codons 241, 245 et, plus modérément, le codon 248 de p53 (Tableau IB) [
29]. Le même type d’analyse a été réalisé sur l’acroléine, un aldéhyde détecté en quantité très importante dans la fumée de cigarette. Ce composé induit des dommages aux mêmes codons que ceux qui sont affectés par le BPDE et semble même avoir un effet inhibiteur sur la réparation des dommages créés par le BPDE [
30]. Cependant, ces dommages ne produisent pas de mutations, probablement parce que l’ADN polymérase est dotée de la capacité de les répliquer sans faire d’erreur (Figure 1) ; néanmoins, sa contribution aux mutations provoquées par la fumée de cigarettes reste à déterminer [
31]. Ainsi, la technologie LMPCR aide peu à peu à éclaircir la contribution relative de différents composés du tabac dans les cancers pulmonaires. |
Lien étiologique entre l’aflatoxine B1 et le carcinome hépatocellulaire D’autres agents génotoxiques ont également été étudiés par LMPCR. C’est le cas de l’aflatoxine B1 (AFB1) dont on soupçonne la responsabilité dans la formation du carcinome hépatocellulaire (CHC). Cette mycotoxine est considérée comme la cause majeure de CHC dans les régions où la nourriture est fortement contaminée par le champignon qui la produit. Un point chaud de mutations uniques, une transversion G → T, a été observé dans ces tumeurs au codon 249 de p53. Denissenko et al. [
32] ont constaté que si l’AFB1 suscite effectivement des dommages sur de nombreuses guanines de p53 (Figure 3C), tel n’est pas particulièrement le cas dans le codon 249, même si l’on constate des dommages à cette position nucléotidique (Tableau 1B). Ainsi, l’exposition à l’AFB1 ne suffirait pas à elle seule à expliquer le fait que ce codon de p53 constitue un point chaud de mutations dans le CHC. Le rôle synergique d’une infection parallèle avec le virus de l’hépatite B a été suggéré ; celle-ci contribuerait à sélectionner les mutants au codon 249. |
La technologie LMPCR apporte donc des informations importantes qui permettent de mettre en rapport les dommages à l’ADN causés par certaines substances génotoxiques et des mutations caractéristiques trouvées dans certaines tumeurs. Cette technique est également fréquemment utilisée pour examiner de près d’autres phénomènes concernant notamment la réparation des dommages, le fait qu’un brin soit transcrit ou non (réparation couplée à la transcription) [4], l’éloignement d’une région par rapport au promoteur [
33] ou encore le rôle exercé par différentes protéines sur certains aspects précis de la réparation [
34,
35]. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
Les travaux présentés dans cette synthèse ont été financés par l’Institut national du cancer du Canada et les Instituts de recherche en santé du Canada. Régen Drouin détient la chaire de recherche du Canada en « Génétique, mutagenèse et cancer ».
|
Footnotes |
1. Hoeijmakers JH. Genome maintenance mechanisms for preventing cancer. Nature 2001; 411 : 366–74. 2. Thilly WG. Analysis of chemically induced mutation in single cell populations. Basic Life Sci 1983; 23 : 337–78. 3. Holmquist GP, Gao S. Somatic mutation theory, DNA repair rates, and the molecular epidemiology of p53 mutations. Mutat Res 1997; 386 : 69–101. 4. Greenblatt MS, Bennett WP, Hollstein M, Harris CC. Mutations in the p53 tumor suppressor gene: clues to cancer etiology and molecular pathogenesis. Cancer Res 1994; 54 : 4855–78. 5. Tornaletti S, Rozek D, Pfeifer GP. The distribution of UV photoproducts along the human p53 gene and its relation to mutations in skin cancer. Oncogene 1993; 8 : 2051–7. 6. Drouin R, Therrien JP. UVB-induced cyclobutane pyrimidine dimer frequency correlates with skin cancer mutational hotspots in p53. Photochem Photobiol 1997; 66 : 719–26. 7. Therrien JP, Rouabhia M, Drobetsky EA, Drouin R. The multilayered organization of engineered human skin does not influence the formation of sunlight-induced cyclobutane pyrimidine dimers in cellular DNA. Cancer Res 1999; 59 : 285–9. 8. Drobetsky EA, Turcotte J, Chateauneuf A. A role for ultraviolet A in solar mutagenesis. Proc Natl Acad Sci USA 1995; 92 : 2350–4. 9. Sage E, Perdiz D, Grof P, et al. DNA damage induced by UVA radiation: role in solar mutagenesis. In : Sage E, Drouin R, Rouabhia M, eds. From DNA photolesions to mutations, skin cancer and cell death. Cambridge, UK : The Royal Society of Chemistry, 2005 : 33–47. 10. Rochette PJ, Therrien JP, Drouin R, et al. UVA-induced cyclobutane pyrimidine dimers form predominantly at thymine-thymine dipyrimidines and correlate with the mutation spectrum in rodent cells. Nucleic Acids Res 2003; 31 : 2786–94. 11. Besaratinia A, Kim SI, Pfeifer GP. Rapid repair of UVA-induced oxidized purines and persistence of UVB-induced dipyrimidine lesions determine the mutagenicity of sunlight in mouse cells. FASEB J 2008; 22 : 2379–92. 12. Hocker T, Tsao H. Ultraviolet radiation and melanoma: a systematic review and analysis of reported sequence variants. Hum Mutat 2007; 28 : 578–88. 13. Besaratinia A, Pfeifer GP. Sunlight ultraviolet irradiation and BRAF V600 mutagenesis in human melanoma. Hum Mutat 2008; 29 : 983–91. 14. Tommasi S, Denissenko MF, Pfeifer GP. Sunlight induces pyrimidine dimers preferentially at 5-methylcytosine bases. Cancer Res 1997; 57 : 4727–30. 15. You YH, Pfeifer GP. Similarities in sunlight-induced mutational spectra of CpG-methylated transgenes and the p53 gene in skin cancer point to an important role of 5-methylcytosine residues in solar UV mutagenesis. J Mol Biol 2001; 305 : 389–99. 16. Lemaire DG, Ruzsicska BP. Kinetic analysis of the deamination reactions of cyclobutane dimers of thymidylyl-3’,5’-2’-deoxycytidine and 2’-deoxycytidylyl-3’,5’-thymidine. Biochemistry 1993; 32 : 2525–33. 17. Tu Y, Dammann R, Pfeifer GP. Sequence and time-dependent deamination of cytosine bases in UVB-induced cyclobutane pyrimidine dimers in vivo. J Mol Biol 1998; 284 : 297–311. 18. Taylor JS. New structural and mechanistic insight into the A-rule and the instructional and non-instructional behavior of DNA photoproducts and other lesions. Mutat Res 2002; 510 : 55–70. 19. Lee DH, Pfeifer GP. Deamination of 5-methylcytosines within cyclobutane pyrimidine dimers is an important component of UVB mutagenesis. J Biol Chem 2003; 278 : 10314–21. 20. Gao S, Drouin R, Holmquist GP. DNA repair rates mapped along the human PGK1 gene at nucleotide resolution. Science 1994; 263 : 1438–40. 21. Tornaletti S, Pfeifer GP. Slow repair of pyrimidine dimers at p53 mutation hotspots in skin cancer. Science 1994; 263 : 1436–8. 22. Rodin SN, Rodin AS, Juhasz A, Holmquist GP. Cancerous hyper-mutagenesis in p53 genes is possibly associated with transcriptional bypass of DNA lesions. Mutat Res 2002; 510 : 153–68. 23. Remontet L. Estimations nationales : tendance de l’incidence et de la mortalité par cancer en France entre 1978 et 2000. BEH 2003; 41-42 : 190–3. 24. Hecht SS. Tobacco smoke carcinogens and lung cancer. J Natl Cancer Inst 1999; 91 : 1194–210. 25. Devereux TR, Taylor JA, Barrett JC. Molecular mechanisms of lung cancer. Interaction of environmental and genetic factors. Giles F. Filley Lecture. Chest 1996; 109 : 14S-9S. 26. Denissenko MF, Pao A, Tang M, Pfeifer GP. Preferential formation of benzo[a]pyrene adducts at lung cancer mutational hotspots in P53. Science 1996; 274 : 430–2. 27. Smith LE, Denissenko MF, Bennett WP, et al. Targeting of lung cancer mutational hotspots by polycyclic aromatic hydrocarbons. J Natl Cancer Inst 2000; 92 : 803–11. 28. Denissenko MF, Pao A, Pfeifer GP, Tang M. Slow repair of bulky DNA adducts along the nontranscribed strand of the human p53 gene may explain the strand bias of transversion mutations in cancers. Oncogene 1998; 16 : 1241–7. 29. Cloutier JF, Drouin R, Weinfeld M, et al. Characterization and mapping of DNA damage induced by reactive metabolites of 4-(methylnitrosamino)-1-(3-pyridyl)-1-butanone (NNK) at nucleotide resolution in human genomic DNA. J Mol Biol 2001; 313 : 539–57. 30. Feng Z, Hu W, Hu Y, Tang MS. Acrolein is a major cigarette-related lung cancer agent: Preferential binding at p53 mutational hotspots and inhibition of DNA repair. Proc Natl Acad Sci USA 2006; 103 : 15404–9. 31. Kim SI, Pfeifer GP, Besaratinia A. Lack of mutagenicity of acrolein-induced DNA adducts in mouse and human cells. Cancer Res 2007; 67 : 11640–7. 32. Denissenko MF, Koudriakova TB, Smith L, et al. The p53 codon 249 mutational hotspot in hepatocellular carcinoma is not related to selective formation or persistence of aflatoxin B1 adducts. Oncogene 1998; 17 : 3007–14. 33. Tu Y, Tornaletti S, Pfeifer GP. DNA repair domains within a human gene: selective repair of sequences near the transcription initiation site. EMBO J 1996; 15 : 675–83. 34. Tu Y, Bates S, Pfeifer GP. Sequence-specific and domain-specific DNA repair in xeroderma pigmentosum and Cockayne syndrome cells. J Biol Chem 1997; 272 : 20747–55. 35. Therrien JP, Drouin R, Baril C, Drobetsky EA. Human cells compromised for p53 function exhibit defective global and transcription-coupled nucleotide excision repair, whereas cells compromised for pRb function are defective only in global repair. Proc Natl Acad Sci USA 1999; 96 : 15038–43. 36. Dalle S, Martin-Denavit T, Thomas L. Hypervariabilité génotypique des mélanomes : un défi thérapeutique. Med Sci (Paris) 2006; 22 : 178–82. 37. Douki T, Leccia MT, Béani JC, et al. Effets néfastes du rayonnement UVA solaire : de nouveaux indices dans l’ADN. Med Sci (Paris) 2007; 23 : 124–6 |