| |
| Med Sci (Paris). 2010 March; 26(3): 238–241. Published online 2010 March 15. doi: 10.1051/medsci/2010263238.L’acétylation des cohésines Un sésame pour le passage des fourches de réplication Marie-Emilie Terret* UMR7622, CNRS/université Pierre et Marie Curie, bâtiment C, 9, quai Saint-Bernard, 75005 Paris, France MeSH keywords: Acétylation, Protéines du cycle cellulaire, génétique, métabolisme, Division du noyau cellulaire, Chromatides, Protéines chromosomiques nonhistones, Réplication de l'ADN, Délétion de gène, Humains, Mitose, Transduction du signal |
La cohésion entre les chromatides sœurs : le cycle des cohésines Pour distribuer de manière égale le matériel génétique dans leur descendance, les cellules dupliquent leurs chromosomes en phase S du cycle cellulaire, reconnaissent le produit dupliqué en tant que chromatides sœurs, puis les ségrégent entre les deux cellules filles lors de la mitose [
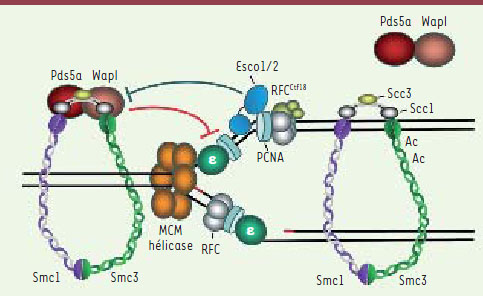
13]. À la fin de la période de réplication, les chromatides dupliquées sont appariées sur toute leur longueur. Ce lien, appelé cohésion, assure leur identification en tant que chromatides sœurs. D’un point de vue moléculaire, la cohésion dépend d’un complexe composé de quatre protéines, les cohésines Scc1, Scc3, Smc1, et Smc3 (Figure 1A). Les cohésines constituent un anneau qui promeut la proximité physique des molécules d’ADN synthétisées lors de la réplication. Les cohésines s’associent à l’ADN en phase G1, lors de la phase d’initiation de la cohésion. À ce stade, les cohésines associées à l’ADN ne sont pas encore fonctionnelles. Elles doivent être activées lors de la phase d’établissement de la cohésion. Ceci a lieu en phase S lors du passage de la fourche de réplication (Figure 1C) [
14]. Chez les vertébrés, les cohésines sont dissociées de l’ADN lors de la mitose en deux temps : en prophase au niveau des bras des chromosomes, puis lors de la transition métaphase-anaphase après clivage par la séparase. Le cycle des cohésines (association à l’ADN, activation, dissociation) peut alors recommencer (Figure 1B). Des protéines comme Wapl et Pds5 peuvent s’associer aux cohésines et réguler leur fonction, notamment en les déstabilisant. Le travail présenté dans cette nouvelle porte sur les liens entre établissement de la cohésion et réplication.
 | Figure 1.
Le cycle des cohésines. A. La cohésion entre les chromatides sœurs est assurée par les cohésines Scc1, Scc3, Smc1 et Smc3 qui forment un anneau de 45 nm de diamètre capable d’entourer les deux chromatides sœurs synthétisées lors de la phase S. Des protéines comme Wapl et Pds5 peuvent s’associer aux cohésines et réguler leur fonction, notamment en les déstabilisant. B. Les cohésines s’associent à l’ADN en phase G1, lors de la phase d’initiation de la cohésion. Les cohésines associées à l’ADN ne sont pas fonctionnelles. Elles doivent être activées lors de la phase d’établissement de la cohésion. Ceci a lieu lors du passage de la fourche réplicative en phase S grâce à des facteurs comme Eco1 et le complexe RFCCtf18. En mitose chez les vertébrés, les cohésines sont dissociées de l’ADN en deux temps : en prophase au niveau des bras des chromosomes grâce a Wapl (dissociation 1), puis à la transition métaphase-anaphase après clivage par la séparase (dissociation 2). Le cycle des cohésines (association à l’ADN, activation, dissociation) peut alors recommencer. |
|
L’établissement de la cohésion entre les chromatides sœurs : rôle d’Eco1 et du complexe RFC Ctf18
L’établissement de la cohésion se fait en phase S de façon concomitante à la fourche de réplication. Établissement de la cohésion et réplication sont donc deux processus intimement liés, même si peu de choses sont connues sur ce lien. Le diamètre de l’anneau formé par les cohésines est supposé être assez grand pour laisser passer la fourche de réplication (Figure 1A et C). Cependant, rien ne permet de déterminer si la fourche passe à travers l’anneau, ou bien si l’anneau est ouvert puis refermé au passage de la fourche. Les protéines impliquées dans l’établissement de la cohésion sont connues, essentiellement grâce aux cribles génétiques menés chez la levure. Deux d’entre elles font l’objet de cette étude (pour une revue générale des autres facteurs, voir [
1]). |
Eco1 est la seule protéine essentielle à l’établissement de la cohésion chez la levure. Chez l’homme, ses deux homologues, Esco1 et Esco2, sont aussi essentiels à ce processus [
2]. Une corrélation a été établie entre la mutation d’Esco2 et une maladie touchant le développement des membres et associée à un retard mental : le syndrome de Roberts [
3]. Eco1 est une acétyl transférase dont la cible est Smc3. C’est l’acétylation de Smc3 qui permet d’établir la cohésion [
4–
6]. Eco1 et l’acétylation de Smc3 s’opposent à l’activité déstabilisatrice de Wapl et Pds5a sur les cohésines [4,
7,
8]. |
Les complexes RFC canoniques sont des composants des fourches de réplication, capables de fixer un clamp autour de l’ADN, le PCNA (proliferating cell nuclear antigen), qui permet l’échange des polymérases alpha primase et delta. Ces complexes sont donc essentiels au processus de réplication. Le complexe alternatif RFCCtf18 impliqué dans l’établissement de la cohésion est constitué de sept protéines, dont trois différentes du complexe RFC canonique : Ctf18, Ctf8 et Dcc1 [
9]. Comme le complexe RFC canonique, il peut aussi recruter PCNA sur l’ADN [9,
10]. Eco1 est capable d’interagir avec PCNA et aussi directement avec le complexe RFCCtf18 [
11]. Le complexe RFCCtf18 pourrait donc être en amont d’Eco1, permettant son recrutement sur l’ADN au moment du passage des fourches de réplication et ainsi l’acétylation de Smc3 de manière coordonnée à la réplication. Cependant, le complexe RFCCtf18 n’est pas essentiel chez la levure, contrairement à Eco1. Cela suggère donc plutôt que Eco1 et le complexe RFCCtf18 pourraient être dans deux voies différentes, la première établissant la cohésion, et la seconde ayant un rôle dans la réplication. |
Liens entre réplication et établissement de la cohésion entre les chromatides soeurs Afin d’élucider les liens entre réplication et établissement de la cohésion, nous avons décidé d’invalider de manière conditionnelle Dcc1 dans des cellules humaines [
12]. La délétion de Dcc1 est très efficace et assure une inhibition complète de l’expression du complexe RFCCtf18. Les cellules qui n’expriment pas le complexe RFCCtf18 ne sont pas viables : elles accumulent des lésions de l’ADN et entrent en sénescence après 7 à 10 divisions cellulaires. Elles présentent des défauts de réplication (fourches lentes ayant tendance à s’arrêter spontanément) et d’établissement de la cohésion (Smc3 est hypoacétylée et les chromatides sœurs n’ont pas de cohésion). Ces deux phénotypes peuvent être liés ou être au contraire complètement indépendants. En effet, le complexe RFCCtf18 peut jouer un rôle à la fois dans l’établissement de la cohésion et dans la réplication, selon le facteur qui est recruté en aval de PCNA : Eco1 (qui acétyle Smc3 et établit la cohésion), la polymérase delta (responsable de la progression de la réplication), ou encore d’autres facteurs. Le complexe RFCCtf18 pourrait donc être à l’origine soit d’une voie linéaire couplant établissement et réplication, soit de deux voies distinctes conduisant respectivement à l’établissement de la cohésion et à la réplication. Afin de discriminer ces deux hypothèses, nous avons analysé la réplication dans des cellules défectives pour l’établissement de la cohésion entre les chromatides sœurs, autres que les cellules Dcc1−/−.
Trois types de cellules défectives pour l’établissement de la cohésion ont été analysés : des cellules provenant de patients atteints du syndrome de Roberts causé par des mutations d’Esco2 [3], des cellules invalidées pour Esco1 ou Esco2 par ARN interférence, et des cellules exprimant de manière inductible une forme non acétylable de Smc3 [6]. Dans tous les cas, les fourches de réplication sont plus lentes, comme dans les cellules Dcc1−/−. Eco1 s’oppose à l’activité déstabilisatrice de Wapl et Pds5a sur les cohésines [4, 7–8]. Nous avons donc testé si Wapl et/ou Pds5a s’opposaient au ralentissement des fourches de réplication observé dans les cellules défectives pour l’établissement de la cohésion et dans les cellules Dcc1−/−
. Nos résultats montrent que c’est le cas, l’invalidation de Wapl et Pds5a corrigeant également les défauts de cohésion observés entre les chromatides sœurs. Ces résultats valident donc l’hypothèse d’une voie unique partant du complexe RFCCtf18 et couplant établissement de la cohésion et réplication, et identifient la voie moléculaire supportant cette hypothèse [12]. |
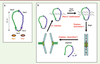
Conclusions et perspectives Ce travail a permis de démontrer pour la première fois que le réseau de protéines permettant l’établissement de la cohésion entre les chromatides sœurs couple l’acétylation de Smc3 et la progression des fourches de réplication [12]. Le modèle suivant peut être proposé : avant l’établissement de la cohésion, Wapl et Pds5a sont fixées sur Scc3, ce qui modifie peut-être sa conformation, empêchant le passage des fourches de réplication à travers l’anneau des cohésines. Quand la fourche rencontre une cohésine, le complexe RFCCtf18 s’associe à l’ADN, ce qui entraîne le recrutement de PCNA et d’Eco1, avec pour conséquence l’acétylation de Smc3. L’acétylation de Smc3 déloge alors Wapl et Pds5a, changeant la conformation des cohésines, ou permettant l’ouverture de l’anneau, afin de laisser passer la fourche de réplication (Figure 2) [12].
 | Figure 2.
Modèle couplant l’acétylation des cohésines par Eco1 au passage des fourches de réplication. Avant l’établissement de la cohésion, Wapl et Pds5a sont fixées sur Scc3, ce qui modifie peut-être sa conformation, de telle manière que les fourches ne puissent pas passer à travers l’anneau des cohésines. Quand la fourche rencontre une cohésine, le complexe RFCCtf18 est chargé sur l’ADN, ce qui entraîne le recrutement de PCNA et d’Eco1, avec pour évènement terminal l’acétylation de Smc3. L’acétylation de Smc3 déloge Wapl et Pds5a, et change alors la conformation des cohésines, ou bien permet d’ouvrir l’anneau, afin de laisser passer la fourche de réplication. Scc : sister chromatid cohesion ; Smc : structural maintenance of chromosomes ; Wapl : wings apart-like ; Pds5 : precocious dissociation of sisters ; Eco : establishment of cohesion ; RFC : replication factor C ; PCNA : proliferating cell nuclear antigen ; Ctf18 : chromosome transmission fidelity ; Dcc1 : defective in sister chromatid cohesion. MCM : minichromosome maintenance. |
Plusieurs questions restent cependant sans réponse et de nombreux aspects du modèle couplant l’acétylation des cohésines par Eco1 et le passage des fourches de réplication devront être testés. Que deviennent les cohésines lors du passage des fourches ? Est-ce que l’acétylation change la conformation de l’anneau afin de laisser passer les fourches ? L’acétylation permet-elle l’ouverture de l’anneau ? Comment Wapl et Pds5 modifient les cohésines en les rendant non permissives au passage des fourches ? Comment l’acétylation de Smc3 permet-elle de déloger Wapl et Pds5a des cohésines ? Des études structurales, bien que difficiles à réaliser, pourront mettre en lumière les changements potentiels de conformation des cohésines en fonction de leur état d’acétylation. Des modèles de réplication in vitro ont été mis au point chez les procaryotes. De tels modèles n’existent pas encore chez les eucaryotes, mais ils permettraient d’apporter les réponses à ces questions et de déterminer la composition et l’architecture des fourches de réplication. |
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Les travaux de recherche exposés ici ont été effectués dans le laboratoire du Dr Prasad V. Jallepalli, Molecular Biology Program, Memorial Sloan-Kettering Cancer Center, 1275 York Avenue, New York, New York 10065, USA. Ils ont été subventionnés par le National Institutes of Health (NIH) et par une bourse Pew Scholar in the Biochemical Sciences allouée à Prasad V. Jallepalli. Je remercie Stéphane Brunet et Marie-Hélène Verlhac pour leur lecture critique de cette nouvelle.
|
1. Uhlmann F. A matter of choice: the establishment of sister chromatid cohesion. EMBO Rep 2009; 10 : 1095–102. 2. Hou F, Zou H. Two human orthologues of Eco1/Ctf7 acetyltransferases are both required for proper sister-chromatid cohesion. Mol Biol Cell 2005; 8 : 3908–18. 3. Vega H, Waisfisz Q, Gordillo M, et al. Roberts syndrome is caused by mutations homolog of yeast ECO1 that is essential for the establishment cohesion. Nat Genet 2005; 37 : 468–70. 4. Ben-Shahar TR, Heeger S, Lehane C, et al. Eco1-dependent cohesin acetylation during establishment of sister chromatid cohesion. Science 2008; 321 : 563–6. 5. Unal E, Heidinger-Pauli JM, Kim W, et al. A molecular determinant for the establishment of sister chromatid cohesion. Science 2008; 321 : 566–9. 6. Zhang J, Shi X, Li Y, et al.Acetylation of Smc3 by Eco1 is required for S phase sister chromatid cohesion in both human and yeast. Mol Cell 2008; 31 : 143–51. 7. Rowland BD, Roig MB, Nishino T, et al. Building sister chromatid cohesion: Smc3 acetylation counteracts an antiestablishment activity. Mol Cell 2009; 33 : 763–74. 8. Sutani T, Kawaguchi T, Kanno R, et al. Budding yeast Wpl1(Rad61)-Pds5 complex counteracts sister chromatid cohesion-establishing reaction. Curr Biol 2009; 19 : 492–7. 9. Bermudez VP, Maniwa Y, Tappin I, et al. The alternative Ctf18-Dcc1-Ctf8-replication factor C complex required for sister chromatid cohesion loads proliferating cell nuclear antigen onto DNA. Proc Natl Acad Sci USA 2003; 100 : 10237–42. 10. Lengronne A, McIntyre J, Katou Y, et al. Establishment of sister chromatid cohesion at the S.cerevisiae replication fork. Mol Cell 2006; 23 : 787–99. 11. Kenna MA, Skibbens RV. Mechanical link between cohesion establishment and DNA replication: Ctf7p/Eco1p, a cohesion establishment factor, associates with three different replication factor C complexes. Mol Cell Biol 2003; 8 : 2999–3007. 12. Terret ME, Sherwood R, Rahman S, et al. Cohesin acetylation speeds the replication fork. Nature 2009; 462 : 231–4. 13. Dorée M. Le déclenchement de la mitose chez les eucaryotes supérieurs. Med Sci (Paris) 2003; 19 : 299–307 14. Michel B, Baldacci G. Réplication (lexique). Med Sci (Paris) 1998; 14 : 1422–7. |