| |
| Med Sci (Paris). 2011 January; 27(1): 41–47. Published online 2011 February 10. doi: 10.1051/medsci/201127141.Autophagie et pathogènes « Bon appétit Messieurs ! » Pierre-Emmanuel Joubert,1 Isabel Pombo Grégoire,1 Grégory Meiffren,1 Chantal Rabourdin-Combe,1 and Mathias Faure1* 1Inserm U851, Université de Lyon 1, IFR128, 21, avenue Tony Garnier, 69365 Lyon Cedex 07, France |
L’autophagie est, avec le protéasome, une voie du catabolisme protéique chez les eucaryotes [
1]. L’autophagie, littéralement « se manger soi-même », est un mécanisme de dégradation lysosomale de portions cytoplasmiques. Il existe trois voies distinctes d’autophagie : (1) l’autophagie coordonnée par les chaperonnes adresse des protéines cytosoliques directement dans le lysosome ; (2) la micro-autophagie dégrade des fragments du cytosol par invagination de la membrane du lysosome ; (3) la macro-autophagie, plus communément appelée autophagie, séquestre de grosses portions du cytoplasme dans des vésicules appelées autophagosomes qui sont dégradées après fusion avec des lysosomes. Une autophagie de portions du noyau appelée piecemeal autophagy, qui utilise des éléments moléculaires de la macro-autophagie, a en outre été décrite. L’autophagie est un mécanisme ubiquitaire et constitutif, très conservé chez les eucaryotes [
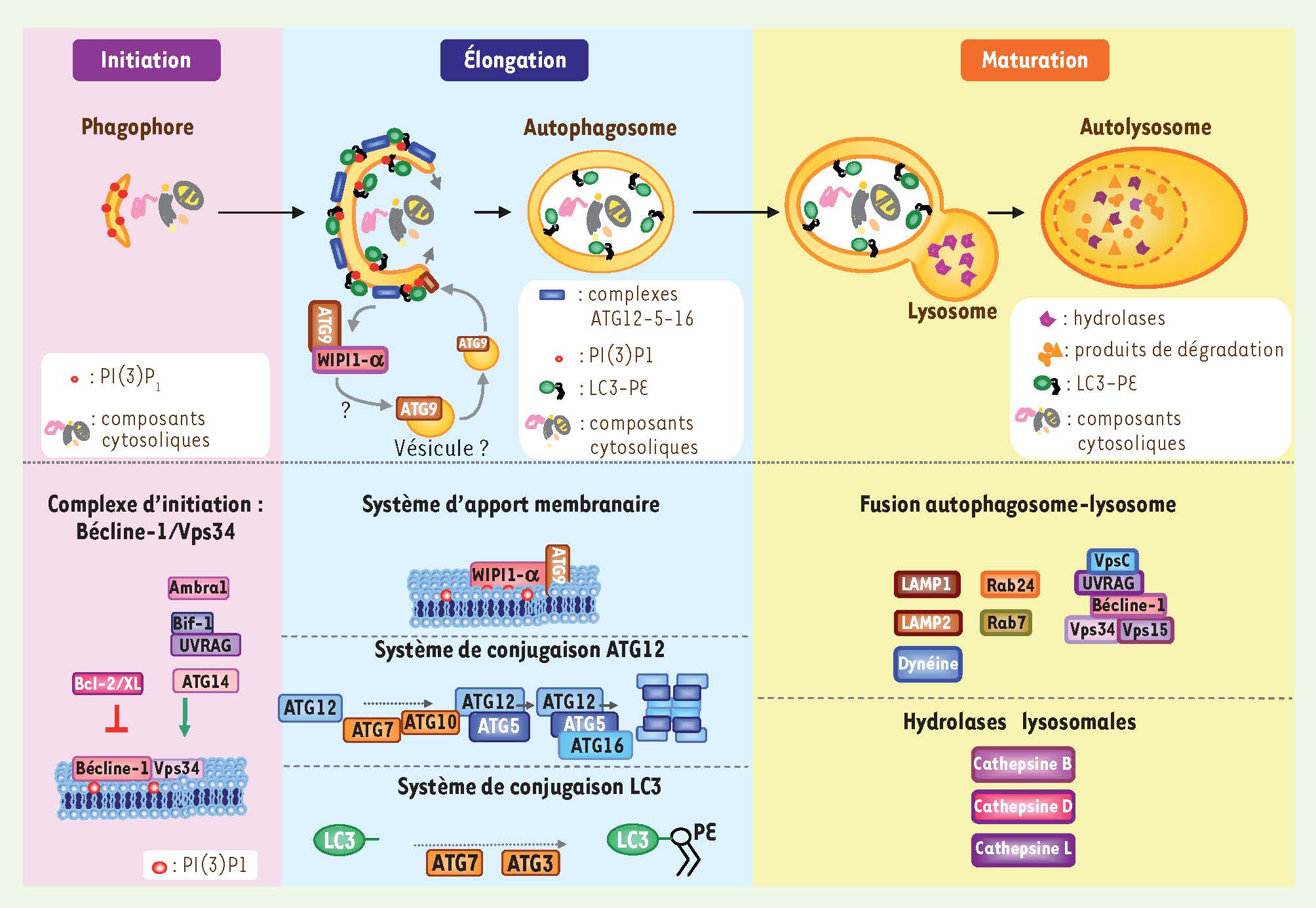
2]. Elle s’articule en trois étapes majeures, sous le dictat orchestré de protéines autophagiques, membres notamment de la famille ATG (autophagy-related genes) (
Figure 1
). Lors de l’étape d’initiation, le complexe de formation des autophagosomes Vps34/Bécline-1 (Vps : vacuolar protein sorting) assure la formation d’un croissant membranaire isolé dans le cytoplasme appelé phagophore. Le phagophore s’étend ensuite lors de l’étape d’élongation, sous le contrôle de deux systèmes de conjugaison impliquant respectivement les protéines ATG12 et LC3 (ATG8). L’autophagosome qui résulte de la fusion des deux extrémités du phagophore en croissance est une vésicule à double membrane qui séquestre en son sein un morceau du cytosol contenant protéines à longue durée de vie, organites sénescents et produits de dégradation. Enfin, au cours de l’étape de maturation, l’autophagosome fusionne avec des lysosomes pour former des autolysosomes qui digèrent le contenu cytoplasmique séquestré (
Figure 1
).  | Figure 1
La machinerie autophagique dans les cellules mammifères. Le complexe d’initiation des autophagosomes Vps34/Bécline-1 joue un rôle crucial dans la formation et l’élongation du phagophore. L’activation de ce complexe, qui est inhibée (trait rouge) ou activée (flèche verte) par les différents partenaires indiqués, génère des groupements phosphatidylinositol 3-phosphate (PI(3)P1) sur les membranes du phagophore. Ces groupements favorisent le recrutement de protéines nécessaires à l’apport de membranes au phagophore en croissance, impliquant WIPI1-α (WD repeat domain, phosphoinositide interacting-1a) et ATG9. Le complexe Vps34/Bécline-1 est également nécessaire à la formation de l’autophagosome par le recrutement des protéines autophagiques ATG12-ATG5 et LC3/ATG8. Aussi, l’élongation est assurée par le système de conjugaison ATG12-ATG5 (impliquant les protéines ATG7 et ATG10) qui mène à la formation de complexes macromoléculaires ATG12-ATG5-ATG16 essentiels au processus d’élongation et nécessaires au recrutement du complexe LC3-PE (LC3-II). Ce dernier résulte d’une modification post-traductionnelle de LC3 qui se lie alors de façon covalente à de la phosphatidyléthanolamine (PE) qui permet l’enchâssement dans la membrane de l’autophagosome en formation (ce processus nécessite ATG7 et ATG3). La fermeture de l’autophagosome s’achève par une réaction de séparation des protéines ATG associées à la membrane du phagophore. Seul LC3-PE lié à la membrane interne de l’autophagosome reste confiné dans ces vésicules. La maturation est régulée par des protéines facilitant les fusions vésiculaires, et la dégradation par des enzymes lysosomales. LAMP : lysosomal membrane proteins ; UVRAG : ultraviolet (UV) radiation resistance-associated gene.
|
Active au sein de chaque cellule, l’autophagie est une arme d’autodéfense cellulaire redoutable qui peut piéger puis dégrader des pathogènes intracellulaires. Ce processus est défini sous le terme de xénophagie. L’extinction expérimentale, in vitro et in vivo, de l’autophagie a démontré le rôle crucial de ce mécanisme dans le contrôle d’agents infectieux. Cependant, en raison de leur confrontation continuelle avec l’autophagie, de nombreux micro-organismes ayant développé des stratégies d’échappement ou de mise à profit de l’autophagie ont été sélectionnés au cours de l’évolution [
3]. |
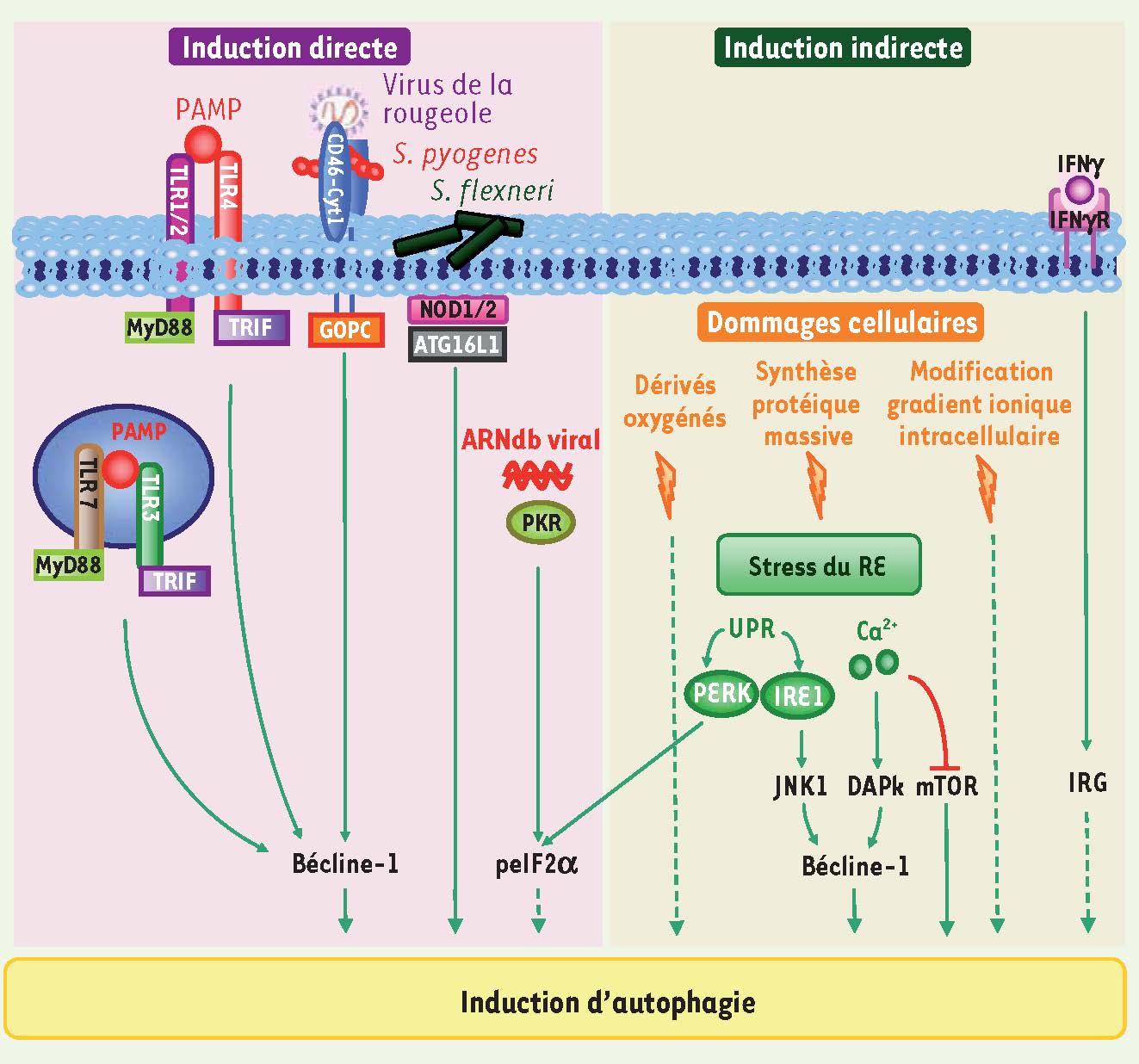
Détection des pathogènes et induction d’autophagie Plusieurs récepteurs de l’immunité innée (PRR pour pattern recognition receptor) stimulent l’autophagie au contact de leurs ligands microbiens (PAMP pour pathogen associated molecular pattern) (
Figure 2
). La sérine/thréonine kinase PKR est le premier PRR décrit pour exercer ce rôle. PKR reconnaît des ARN viraux double brin et active l’autophagie en phosphorylant eIF2α (eukaryotic initiation factor 2-α). Les mécanismes d’activation de PKR et de phosphorylation de eIF2α dans l’induction d’autophagie lors d’infections virales restent cependant inconnus [3]. Plus récemment, il a été décrit que la liaison de ligands exprimés par des pathogènes à des récepteurs de la famille des TLR (toll-like receptors) exprimés sur les membranes endosomales (TLR3/7) ou plasmiques (TLR1/2/4) conduit à l’induction d’autophagie [
4,
5]. Selon leur nature, ces récepteurs utilisent des voies signalétiques distinctes pour induire l’autophagie : TLR7 engage la protéine adaptatrice MyD88 (myeloid differentiation protein 88), alors que TLR4 implique TRIF (toll/interleukin-1 receptor [TIR]-domain containing adaptor inducing interferon-beta). Il a été montré que MyD88 et TRIF peuvent lier la protéine autophagique Bécline-1, ce qui pourrait soutenir l’induction d’autophagie par les TLR [
6]. 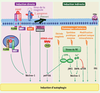 | Figure 2
Induction d’autophagie par détection de pathogènes intracellulaires. La détection directe de pathogènes par des senseurs moléculaires de la cellule, ou les signaux indirects de dommages cellulaires résultant de l’infection (flèches vertes) permettent l’induction de l’autophagie. Le stress du réticulum endoplasmique (RE) sert de sentinelle aux infections virales. L’activation des voies de signalisation dépendantes de IRE1 (inositol requiring enzyme 1) et PERK (PKR-related endoplasmic reticulum kinase) active l’autophagie, tout comme l’efflux de calcium. Les PAMP reconnus pas les différents TLR sont distincts. ARNdb : ARN double brin. |
Deux membres de la famille des NLR (nod-like receptor), NOD1 et NOD2, des PRR cytosoliques, induisent l’autophagie au contact de leurs ligands respectifs ou de bactéries les exprimant [
7]. Cette induction nécessite le recrutement d’ATG16L1 (L pour like) par NOD1/2 au site d’entrée des bactéries dans les cellules, bien que cette association physique ne suffise pas à l’achèvement du processus. L’autophagie peut aussi être induite par au moins un récepteur cellulaire pour de nombreux pathogènes distinct des PRR, CD46. L’engagement de l’une de ses isoformes (CD46-Cyt-1) par des ligands naturels comme les streptocoques de groupe A et le virus de la rougeole, induit l’autophagie. Cette induction met en jeu la protéine d’échafaudage GOPC (golgi-associated PDZ and coiled-coil motif-containing protein) qui lie CD46-Cyt-1 au complexe de formation des autophagosomes Vps34/Bécline-1 [
8]. Enfin, d’autres signaux permettent la mise en place d’une réponse autophagique dans un contexte infectieux (
Figure 2
). L’interféron gamma (IFN-γ) induit l’autophagie dans les macrophages par une voie de signalisation impliquant la petite GTPase IRG (immunity-related GTPase) [3]. De même, des signaux alertant sur des dommages cellulaires créés par des infections activent la machinerie autophagique : c’est le cas de la réponse au mauvais repliement des protéines (UPR pour unfolded protein response) consécutive au stress du réticulum endoplasmique provoqué par une production excessive de protéines lors d’infections virales, des modifications de gradients ioniques intracellulaires ou encore de la production de dérivés de l’oxygène [
9]. L’identification des senseurs de pathogènes capables d’induire l’autophagie et celle des intermédiaires moléculaires qui en sont responsables sont encore préliminaires et représentent un défi d’envergure pour la compréhension des relations entre autophagie et pathogènes. Cependant, il est probable que plusieurs de ces intermédiaires participent de concert à l’activation d’une autophagie antimicrobienne. |
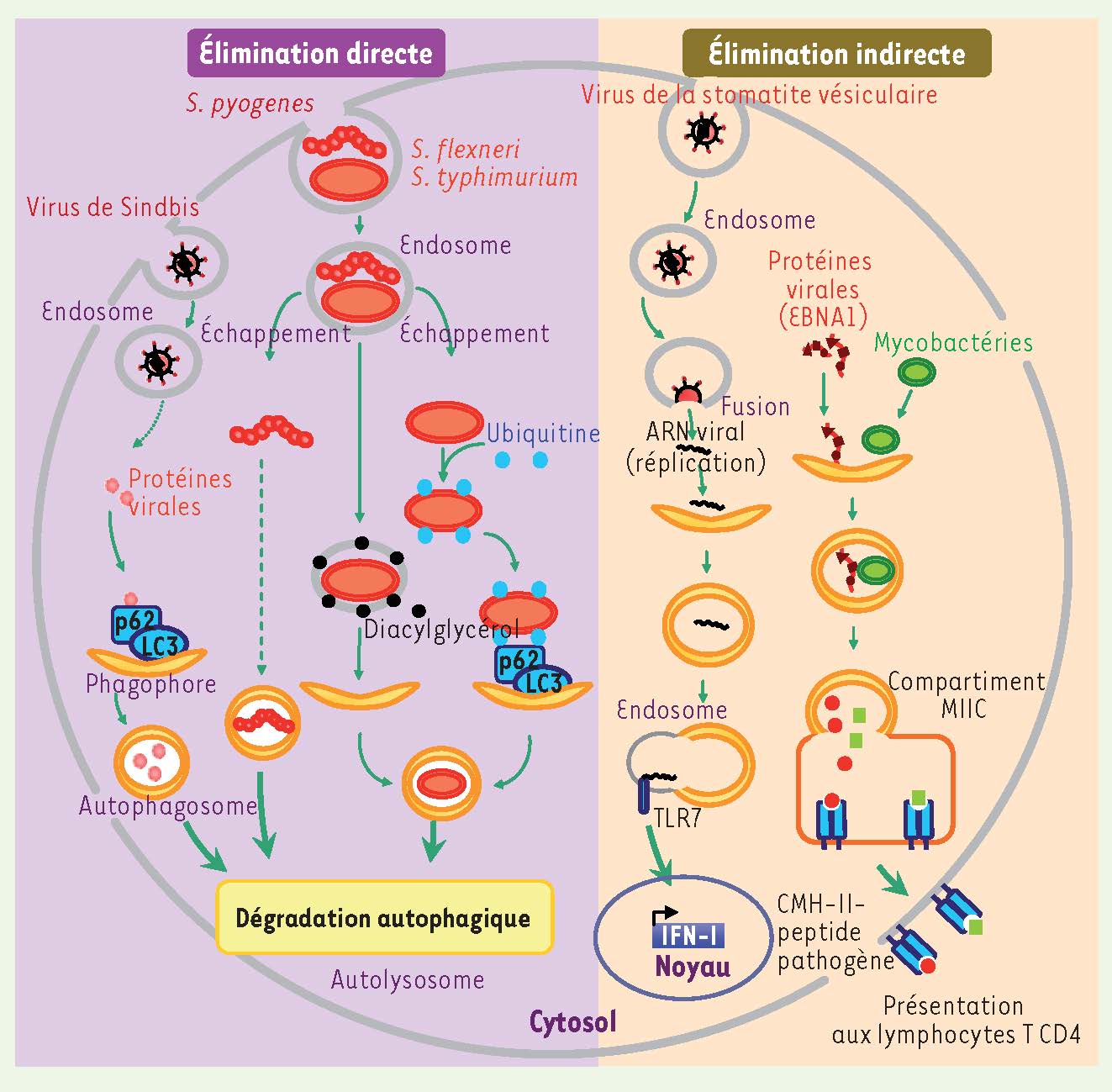
Rôle protecteur de l’autophagie contre les infections Présente à l’état basal, l’autophagie peut augmenter en réponse à la détection par la cellule de pathogènes pour dégrader ces derniers par xénophagie, et/ou servir d’intermédiaire à la mise en place d’autres réponses antimicrobiennes (
Figure 3
). 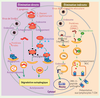 | Figure 3
Protection cellulaire contre les infections par autophagie. L’autophagie peut directement contrôler des infections par des pathogènes intracellulaires en les éliminant physiquement ou en dégradant certains de leurs composants essentiels. L’autophagie sert également d’intermédiaire à l’initiation d’autres réponses de lutte contre les infections, la réponse IFN-I ou l’activation d’une réponse immunitaire adaptative. Les protéines cellulaires sont indiquées en bleu, les protéines de pathogènes par des couleurs variées. MIIC : vésicules riches en molécules du CMH-II. |
L’autophagie : un mécanisme de dégradation directe des pathogènes L’autophagie a été décrite comme un mécanisme de dégradation non spécifique. Ainsi, l’augmentation de ce processus en réponse à la détection des pathogènes présents dans le cytoplasme pourrait suffire à la capture et à l’élimination de ces derniers. Cependant, de récents travaux décrivent un adressage sélectif de pathogènes ou de certains de leurs composants vers les autophagosomes (
Figure 3
). Salmonella typhimurium est une bactérie intracellulaire gram négative et, dans les cellules infectées, une partie des bactéries sont dégradées par autophagie. Deux processus permettent l’adressage de S. typhimurium vers cette voie : (1) certaines bactéries sont rapidement recouvertes d’ubiquitine dans le cytosol, puis dirigées vers la voie autophagique grâce au pontage de la protéine cellulaire p62 qui possède un domaine de liaison pour l’ubiquitine et un autre pour la protéine autophagique LC3 [
10] ; (2) d’autres bactéries sont adressées vers l’autophagie via des signaux impliquant la formation de diacylglycérol (DAG) qui se lie aux membranes endommagées des vésicules d’internalisation des bactéries [
11]. Par ailleurs, p62 peut permettre la dégradation autophagique sélective de protéines virales accumulées via un processus indépendant de l’ubiquitine [
12]. Ainsi, plusieurs mécanismes semblent coexister et contribuent à diriger des pathogènes intracellulaires, ou certains de leurs composants, vers la machinerie autophagique (
Figure 3
). Quel que soit le mécanisme d’induction et/ou de ciblage autophagique, ce processus contrôle nombre d’agents infectieux (
Figure 3
) [3]. La dégradation autophagique de Streptococcus pyogenes est la première à avoir été démontrée [
13]. Cette bactérie extracellulaire à Gram positif peut pénétrer dans les cellules épithéliales par internalisation. Les bactéries s’échappent alors des endosomes et, dans le cytosol, sont prises en charge par l’autophagie. Rapidement après infection, près de 80 % d’entre elles se retrouvent dans des autophagosomes. De plus, alors qu’elles sont très efficacement éliminées dans des cellules saines, elles ne le sont plus dans des cellules dont la machinerie autophagique est déficiente, et peuvent même y proliférer. La place de la dégradation autophagique dans les infections virales reste en revanche plus floue [
31]. La réplication de plusieurs virus est exacerbée dans des cellules dont l’autophagie est bloquée, ce qui indique un rôle protecteur, contre le virus, de l’autophagie dans ce contexte. Le seul exemple décrit à ce jour d’une dégradation de protéines virales par autophagie montre que ce processus protège d’un effet délétère dû à l’accumulation de protéines virales dans les cellules infectées, mais n’a pas de conséquences sur la réplication virale elle-même [12]. Il est néanmoins probable que l’autophagie puisse dégrader des particules virales ou des composants viraux afin d’en limiter la capacité réplicative. Cela a été montré pour une souche recombinante du virus herpes simplex 1 (HSV-1) qui n’exprime pas le facteur de virulence ICP34.5 : elle est détectée dans des autophagosomes avant d’être dégradée [
14]. L’autophagie : un relais pour d’autres réponses antimicrobiennes Les interférons de type I (IFN-I) sont des cytokines essentielles pour le contrôle des infections virales. Elles sont produites par les cellules infectées lorsque celles-ci détectent la présence de composants viraux, via l’activation de PRR membranaires ou cytosoliques. Lors de l’infection des cellules dendritiques plasmacytoïdes par le virus de la stomatite vésiculaire (VSV), le matériel génétique viral libéré dans le cytosol est capturé au sein d’autophagosomes puis, après fusion avec des endosomes, délivré à TLR7 qui enclenche la transduction de signaux menant à la production d’IFN-I (
Figure 3
) [
15]. Il reste à déterminer si l’autophagie régule positivement la production d’IFN-I dans d’autres types cellulaires et/ou via l’engagement d’autres PRR. L’autophagie facilite également le déclenchement de réponses immunitaires adaptatives. Elle permet l’apprêtement de protéines de pathogènes et le chargement de peptides sur des molécules du complexe majeur d’histocompatibilité de classe II (CMH-II), après fusion des autophagosomes avec des vésicules spécialisées riches en CMH-II (
Figure 3
) [
16]. Ces dernières présentent alors les échantillons peptidiques issus de protéines de pathogènes aux lymphocytes T CD4 naïfs, lesquels s’activent et déclenchent une réponse immunitaire adaptative efficace qui éradiquera l’agent infectieux et assurera une mémoire immunitaire à long terme. Récemment, l’autophagie induite par l’engagement de NOD2 a été incriminée dans le contrôle d’une infection pathogène d’une part en favorisant la dégradation de l’agent infectieux, et d’autre part en permettant la présentation de peptides pathogènes par le CMH-II. Ces deux processus sont fortement perturbés en l’absence d’une autophagie fonctionnelle [
17]. L’autophagie participe également à la présentation de peptides antigéniques sur les molécules du CMH-I, nécessaire à l’activation et à la fonction des lymphocytes T CD8 cytotoxiques [
18,
32]. Enfin, elle peut, dans certains contextes, mener à la mort cellulaire [
19]. Bien qu’une mort autophagique consécutive à une infection microbienne n’ait à ce jour jamais été décrite, un tel mécanisme pourrait conceptuellement représenter une protection contre la diffusion de pathogènes. Ainsi, il a récemment été montré que la mort cellulaire autophagique optimise fortement la présentation croisée d’antigènes par les cellules dendritiques [
20]2, ce qui pourrait participer au contrôle d’infections. L’invalidation de l’expression de gènes ATG a permis de démontrer in vivo le rôle antimicrobien joué par l’autophagie. Un tel rôle concerne aussi les plantes que l’autophagie protège du virus de la mosaïque du tabac, la drosophile, qu’elle protège de Listeria monocytogenes et du VSV, et la souris vis-à-vis d’infections par L. monocytogenes, Toxoplasma gondii, HSV-1 (virus herpes simplex de type 1) ou encore le virus de Sindbis. Les mécanismes utilisés par l’autophagie in vivo (xénophagie et/ou relais pour d’autres réponses immunes) ne sont cependant pas précisés [3]. |
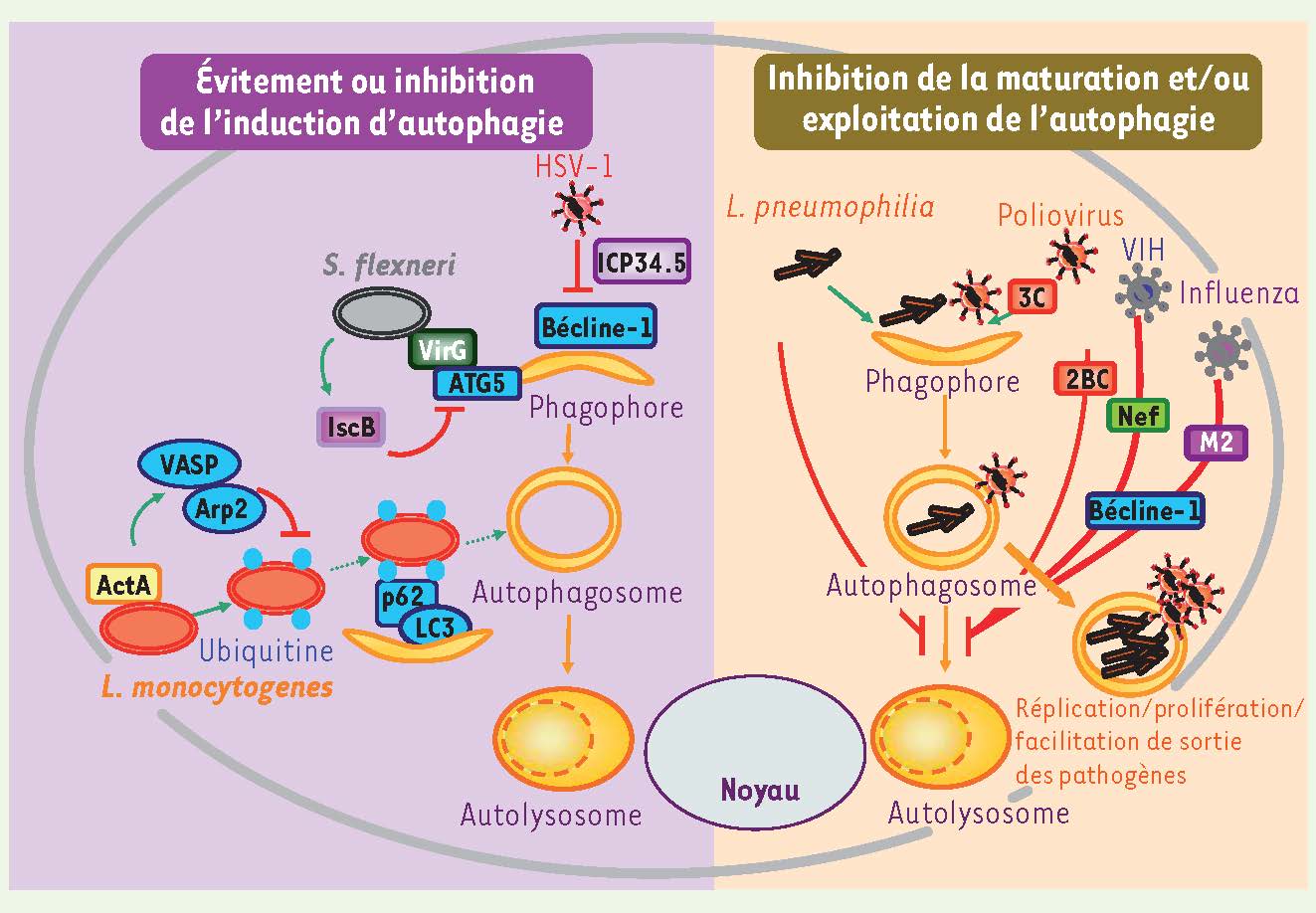
Manipulation de l’autophagie pour l’établissement d’une infection Les pathogènes intracellulaires sont au contact permanent des composants de la machinerie d’autophagie cellulaire. Il semble donc inévitable que nombre d’entre eux, capables d’éviter l’autophagie, de l’inhiber ou de l’utiliser à leur profit, aient été sélectionnés au cours de l’évolution (
Figure 4
). 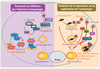 | Figure 4
Mécanismes d’échappement et de détournement de l’autophagie par des pathogènes. Les pathogènes utilisent des stratégies variées pour éviter d’être des cibles de l’autophagie. Certains d’entres eux utilisent même ce processus pour faciliter leur survie, leur prolifération/réplication et/ou leur sortie des cellules infectées. Les protéines cellulaires sont indiquées en bleu, les protéines de pathogènes par des couleurs variées. |
Stratégies d’évitement de l’adressage au processus autophagique Certains pathogènes ont développé des stratégies qui leur permettent de s’exclure activement du processus autophagique sans toutefois l’inhiber [ 3]. L’infection par Shigella flexneri mène rapidement à l’adressage des vestiges membranaires d’internalisation vers l’autophagie par une voie dépendante de p62 [
21]. De plus, la protéine de surface VirG de S. flexneri, impliquée dans la mobilité bactérienne par polymérisation de l’actine, lie ATG5, ce qui adresse la bactérie vers la voie autophagique. Afin d’échapper à ce ciblage, la bactérie sécrète le facteur IcsB qui masque le site de liaison de ATG5 sur VirG et détourne ainsi la bactérie de cette voie (
Figure 4
) [
22]. Un autre exemple est celui de Listeria monocytogenes : la protéine ActA prévient l’ubiquitinylation de la bactérie dans le cytoplasme en recrutant des protéines cellulaires à la surface bactérienne [
23]. Des bactéries dépourvues de cette activité d’ActA sont ubiquitinylées, reconnues par p62 et adressées vers la voie de dégradation autophagique (
Figure 4
). Les mécanismes d’évitement de l’autophagie sont probablement l’apanage de nombreux pathogènes, en particulier de ceux qui sont responsables d’infections chroniques persistantes : ils doivent en effet échapper à une dégradation autophagique excessive sans l’inhiber pour autant, au risque de créer des perturbations cellulaires qui leur seraient défavorables. Stratégies d’inhibition de l’induction du processus autophagique Seuls des virus ont été à ce jour clairement impliqués dans l’inhibition de la phase initiale du processus d’autophagie. L’exemple le plus démonstratif est celui de HSV-1. Le facteur de virulence de ce virus, ICP34.5, interagit physiquement avec Bécline-1 ; cette interaction prévient l’autophagie et l’annihile (
Figure 4
). ICP34.5 agit également à ce stade initial en prévenant l’activité de la PKR [ 3]. Comme pour HSV-1, les stratégies microbiennes d’inhibition de l’induction d’autophagie semblent cibler essentiellement des acteurs moléculaires de l’exécution de la machinerie autophagique [ 3]. Stratégies d’échappement à la dégradation autophagique L’inhibition de la fusion des lysosomes avec les autophagosomes est la stratégie la plus fréquemment utilisée par les micro-organismes pour contrecarrer l’autophagie. Ce n’est pas étonnant dans la mesure où la maturation autophagique est l’étape la plus dangereuse pour le pathogène puisqu’elle mène à sa dégradation, de fait irréversible. Il y a un double avantage à inhiber cette étape : prévenir la destruction et offrir une chape de protection, au sein des autophagosomes, contre d’autres facteurs antimicrobiens cytosoliques. Bactéries, virus et parasites utilisent cette stratégie via des mécanismes moléculaires encore mal connus. Par exemple, au cours de l’infection par Legionella pneumophila, l’autophagie est induite via le système de sécrétion bactérien de type IV. Cependant, cette bactérie retarde la maturation des autophagosomes, par des mécanismes toujours inconnus, ce qui permet son adaptation à des compartiments intracellulaires acides [
24]. De même des L. monocytogenes mutantes exprimant de faibles quantités de listériolysine O, une enzyme responsable de la perforation des vésicules d’internalisation qui permet l’échappement des bactéries dans le cytoplasme, restent contenues au sein de structures appelées SLAP ( spacious listeria-containing phagosomes) aux caractéristiques autophagiques. Ces compartiments ne s’acidifient pas, ce qui empêche la dégradation des bactéries et permet même leur réplication, à un niveau cependant plus faible que celui qui est observé lors de leur réplication dans le cytosol [
25]. Stratégies d’exploitation de l’autophagie au profit du pathogène L’autophagie produit de nombreux métabolites primaires, ce qui en fait une cible intéressante pour tout pathogène s’il peut en détourner l’activité pour son propre compte. Certains pathogènes bactériens peuvent utiliser l’auto-séquestration et bénéficier d’éléments nutritifs exploitables pour leur prolifération [ 3]. Ce détournement de la machinerie d’autophagie à leur profit est bien documenté dans la littérature dans le cas des virus. L’inhibition de l’autophagie se traduit par la diminution de la production de particules infectieuses dans le cas des virus de la polio, de l’immunodéficience humaine (VIH) et de l’hépatite C. Le poliovirus, via les protéines 2BC et 3C, induit un processus d’autophagie dans les cellules qu’il infecte et tire avantage de ce mécanisme pour augmenter sa virulence. Le virus pourrait induire la formation d’autophagosomes qui portent sa machinerie de réplication et/ou il utiliserait les vésicules autophagiques pour faciliter sa sortie des cellules [3]. La protéine NEF (facteur négatif) du VIH semble également être un facteur de virulence qui, en s’associant à Bécline-1, inhibe la maturation autophagique et potentialise la production de particules infectieuses dans les macrophages infectés [
26]. Le virus de l’hépatite C exploite également cette voie à son bénéfice mais via des mécanismes moléculaires encore inconnus [
27]. Enfin, un autre mode d’exploitation de l’autophagie a récemment été décrit pour le virus de la grippe, influenza. Par l’action de sa protéine M2 qui lie Bécline-1, ce virus inhibe la maturation autophagique, avec pour conséquence de compromettre la survie des cellules infectées sans pour autant modifier la production de virus infectieux [
28]. Stratégie d’adaptation aux cibles cellulaires Le VIH a développé des stratégies complexes pour contrecarrer l’autophagie. Son impact sur ce processus est différent selon la nature des cellules infectées, mais également selon qu’il infecte directement une cellule ou qu’il soit simplement en contact avec elle. La simple mise en contact de lymphocytes T CD4 humains non infectés avec des cellules exprimant la protéine d’enveloppe Env du VIH induit l’autophagie, puis la mort des lymphocytes par apoptose [
29]. Au contraire, le VIH bloque l’autophagie lorsqu’il infecte des lymphocytes T CD4, évitant ainsi d’en être une cible de dégradation [
30]. Enfin, comme nous l’avons décrit précédemment, ce virus détourne l’autophagie en inhibant l’étape de maturation dans les macrophages infectés [ 26]. Ainsi, des pathogènes qui ont élaboré des stratégies moléculaires variées pour manipuler le processsus autophagique ont été sélectionnés au cours de l’évolution. Cependant, à ce jour, la pertinence physiologique de ces stratégies n’a pas encore été démontrée in vivo. |
À l’instar de Ruy Blas [
33], ce pair d’Espagne qui dénonce la corruption de ses ministres en dressant la liste des territoires perdus, nous devons maintenant comprendre tous les caractères corruptibles de l’autophagie par les pathogènes. Quels sont les rouages moléculaires utilisés par les pathogènes pour inhiber la maturation autophagique ou pour détourner l’autophagie ? Comment des pathogènes évitent-ils d’être détectés et dirigés par des molécules d’adressage vers la voie autophagique ? Les cibles moléculaires autophagiques sont-elles uniques et/ou communes à différents pathogènes dont ceux qui sont à l’origine des infections chroniques ou persistantes ? Les processus d’adaptation microbienne à l’autophagie sont-ils également opérationnels in vivo ? Certaines de ces questions d’actualité pourraient être prochainement résolues et laissent espérer le développement de nouvelles stratégies de lutte à l’encontre d’agents infectieux. « Bon appétit, Messieurs ! » [33]. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
Le travail de notre laboratoire sur l’autophagie est soutenu par l’ANR-08-JCJC-0064-01 et le Cluster de recherche 10 infectiologie de la région Rhône-Alpes.
|
Footnotes |
1.
Xie
Z
,
Klionsky
DJ
. Autophagosome formation: core machinery and adaptations . Nat Cell Biol.
2007; ; 9 : :1102.-1109. 2.
Puyal
J
,
Ginet
V
,
Vaslin
A
, et al. Les deux visages de l’autophagie dans le système nerveux . Med Sci (Paris).
2009; ; 25 : :383.-390. 3.
Deretic
V
,
Levine
B
. Autophagy, immunity, and microbial adaptations . Cell Host Microbe.
2009; ; 5 : :527.-549. 4.
Xu
Y
,
Jagannath
C
,
Liu
XD
, et al. Toll-like receptor 4 is a sensor for autophagy associated with innate immunity . Immunity.
2007; ; 27 : :135.-144. 5.
Delgado
MA
,
Elmaoued
RA
,
Davis
AS
, et al. Toll-like receptors control autophagy . Embo J.
2008; ; 27 : :1110.-1121. 6.
Shi
CS
,
Kehrl
JH
. MyD88 and Trif target Beclin 1 to trigger autophagy in macrophages . J Biol Chem.
2008; ; 283 : :33175.-33182. 7.
Travassos
LH
,
Carneiro
LA
,
Ramjeet
M
, et al. Nod1 and Nod2 direct autophagy by recruiting ATG16L1 to the plasma membrane at the site of bacterial entry . Nat Immunol.
2010; ; 11 : :55.-62. 8.
Joubert
PE
,
Meiffren
G
,
Grégoire
IP
, et al. Autophagy induction by the pathogen receptor CD46 . Cell Host Microbe.
2009; ; 6 : :354.-366. 9.
Sumpter
R
Jr
,
Levine
B
. Autophagy and innate immunity: Triggering, targeting and tuning . Semin Cell Dev Biol.
2010; ; 21 : :699.-711. 10.
Zheng
YT
,
Shahnazari
S
,
Brech
A
, et al. The adaptor protein p62/SQSTM1 targets invading bacteria to the autophagy pathway . J Immunol.
2009; ; 183 : :5909.-5916. 11.
Shahnazari
S
,
Yen
WL
,
Birmingham
CL
, et al. A diacylglycerol-dependent signaling pathway contributes to regulation of antibacterial autophagy . Cell Host Microbe.
2010; ; 8 : :137.-146. 12.
Orvedahl
A
,
MacPherson
S
,
Sumpter
R
Jr
, et al. Autophagy protects against Sindbis virus infection of the central nervous system . Cell Host Microbe.
2010; ; 7 : :115.-127. 13.
Nakagawa
I
,
Amano
A
,
Mizushima
N
, et al. Autophagy defends cells against invading group A Streptococcus . Science.
2004; ; 306 : :1037.-1040. 14.
Orvedahl
A
,
Alexander
D
,
Tallóczy
Z
, et al. HSV-1 ICP34.5 confers neurovirulence by targeting the Beclin 1 autophagy protein . Cell Host Microbe.
2007; ; 1 : :23.-35. 15.
Dreux
M
,
Chisari
FV
. Viruses and the autophagy machinery . Cell Cycle.
2010; ; 9. 16.
Schmid
D
,
Pypaert
M
,
Munz
C
. Antigen-loading compartments for major histocompatibility complex class II molecules continuously receive input from autophagosomes . Immunity.
2007; ; 26 : :79.-92. 17.
Cooney
R
,
Baker
J
,
Brain
O
, et al. NOD2 stimulation induces autophagy in dendritic cells influencing bacterial handling and antigen presentation . Nat Med.
2010; ; 16 : :90.-97. 18.
English
L
,
Chemali
M
,
Duron
J
, et al. Autophagy enhances the presentation of endogenous viral antigens on MHC class I molecules during HSV-1 infection . Nat Immunol.
2009; ; 10 : :480.-487. 19.
McPhee
CK
,
Logan
MA
,
Freeman
MR
,
Baehrecke
EH
. Activation of autophagy during cell death requires the engulfment receptor Draper . Nature.
2010; ; 465 : :1093.-1096. 20.
Uhl
M
,
Kepp
O
,
Jusforgues-Saklani
H
, et al. Autophagy within the antigen donor cell facilitates efficient antigen cross-priming of virus-specific CD8+ T cells . Cell Death Differ.
2009; ; 16 : :991.-1005. 21.
Dupont
N
,
Lacas-Gervais
S
,
Bertout
J
, et al. Shigella phagocytic vacuolar membrane remnants participate in the cellular response to pathogen invasion and are regulated by autophagy . Cell Host Microbe.
2009; ; 6 : :137.-149. 22.
Ogawa
M
,
Yoshimori
T
,
Suzuki
T
, et al. Escape of intracellular Shigella from autophagy . Science.
2005; ; 307 : :727.-731. 23.
Yoshikawa
Y
,
Ogawa
M
,
Hain
T
, et al.
Listeria monocytogenes ActA-mediated escape from autophagic recognition . Nat Cell Biol.
2009; ; 11 : :1233.-1240. 24.
Amer
AO
,
Swanson
MS
. Autophagy is an immediate macrophage response to Legionella pneumophila . Cell Microbiol.
2005; ; 7 : :765.-778. 25.
Birmingham
CL
,
Canadien
V
,
Kaniuk
NA
, et al. Listeriolysin O allows Listeria monocytogenes replication in macrophage vacuoles . Nature.
2008; ; 451 : :350.-354. 26.
Kyei
GB
,
Dinkins
C
,
Davis
AS
, et al. Autophagy pathway intersects with HIV-1 biosynthesis and regulates viral yields in macrophages . J Cell Biol.
2009; ; 186 : :255.-268. 27.
Dreux
M
,
Gastaminza
P
,
Wieland
SF
,
Chisari
FV
. The autophagy machinery is required to initiate hepatitis C virus replication . Proc Natl Acad Sci USA.
2009; ; 106 : :14046.-14051. 28.
Gannagé
M
,
Dormann
D
,
Albrecht
R
, et al. Matrix protein 2 of influenza A virus blocks autophagosome fusion with lysosomes . Cell Host Microbe.
2009; ; 6 : :367.-380. 29.
Espert
L
,
Denizot
M
,
Grimaldi
M
, et al. Autophagie et destruction des lymphocytes T CD4 par le VIH-1 . Med Sci (Paris).
2006; ; 22 : :677.-678. 30.
Espert
L
,
Varbanov
M
,
Robert-Hebmann
V
, et al. Differential role of autophagy in CD4 T cells and macrophages during X4 and R5 HIV-1 infection . PLoS One.
2009; ; 4 : :e5787.. 31.
Le Roux
D
,
Niedergang
F
. Autophagie et micro-organismes . Med Sci (Paris).
2010; ; 26 : :534.-537. 32.
English
L
,
Chemali
M
,
Desjardins
M
. Contribution de l’autophagie dans l’apprêtement d’antigènes viraux endogènes . Med Sci (Paris).
2009; ; 25 : :906.-909. 33.
Hugo
V
. Ruy Blas . Paris: : Éditions J’ai Lu; , 2005 : :142. p. |