| |
| Med Sci (Paris). 2011 May; 27(5): 483–485. Published online 2011 May 25. doi: 10.1051/medsci/2011275013.La méthylation de l’ADN, un acteur-clé de la pluripotence Michaël Weber1* 1Université de Strasbourg/CNRS, UMR 7242, Biotechnologie et signalisation cellulaire, École supérieure de biotechnologie de Strasbourg, boulevard Sébastien Brant, 67412Illkirch, France MeSH keywords: Animaux, Différenciation cellulaire, génétique, DNA (cytosine-5-)-methyltransferase, déficit, physiologie, Méthylation de l'ADN, effets des médicaments et des substances chimiques, Embryon de mammifère, cytologie, métabolisme, Régulation de l'expression des gènes au cours du développement, Humains, Souris, Souris knockout, Cellules souches pluripotentes, Facteurs de transcription |
Au cours du développement embryonnaire, les cellules souches pluripotentes perdent leur pluripotence en se spécialisant progressivement en de nombreux types cellulaires aux fonctions variées. Ce processus est contrôlé par des facteurs de transcription qui activent des programmes génétiques spécifiques de chaque lignage cellulaire, mais également par des mécanismes épigénétiques dont des modifications post-traductionnelles des histones et la méthylation de l’ADN. L’ADN est méthylé sur le carbone 5 des cytosines, principalement dans le contexte de dinucléotides CpG, et cette modification est abondante dans le génome de tous les organismes vertébrés. La présence de cytosines méthylées dans la séquence promotrice d’un gène est généralement associée à une répression transcriptionnelle car elle inhibe directement la fixation de certains facteurs de transcription et peut également recruter des répresseurs de la famille des MBP (methyl binding proteins). Puisque les profils de méthylation sont fidèlement copiés au cours de la réplication du génome grâce à l’action de l’enzyme DNMT1 (DNA methyltransferase 1), la méthylation de l’ADN peut être considérée comme un système de mémoire épigénétique qui permet de maintenir de manière stable l’état réprimé de certains gènes dans une population cellulaire [
1]. |
Méthylation de gènes maîtres de la pluripotence au cours de la différenciation des cellules ES Plusieurs éléments suggèrent que la méthylation de l’ADN est importante pour la régulation de la pluripotence et des étapes précoces du développement embryonnaire chez les mammifères : (1) les profils de méthylation sont fortement reprogrammés au moment de la restauration de la pluripotence dans les embryons préimplantatoires et les cellules germinales primordiales [1] ; (2) les cellules pluripotentes ES (embryonic stem) murines déficientes pour les enzymes DNMT3A et DNMT3B (DNA methyltransferase 3A et 3B) qui catalysent la méthylation de novo de l’ADN perdent leur capacité de différenciation in vitro [
2] ; (3) les souris knock-out pour les gènes Dnmt3a et Dnmt3b meurent à un stade embryonnaire précoce [
3]. Pour comprendre le lien entre méthylation de l’ADN et pluripotence, il était dans un premier temps primordial d’identifier les gènes cibles de la méthylation, ce qui a été rendu possible par l’émergence de nouvelles technologies permettant de cartographier la méthylation de l’ADN à grande échelle. Plusieurs équipes de recherche ont ainsi cartographié les promoteurs cibles de la méthylation de l’ADN dans des cellules ES pluripotentes et différenciées. De manière intéressante, ces études ont montré qu’il n’y avait pas de différences de méthylation sur la plupart des gènes entre les cellules pluripotentes et différenciées. En revanche, plusieurs gènes indispensables au maintien la pluripotence comme ceux codant pour Oct4, Nanog, Gdf3, Tdgf1, Rex1 (mais pas le gène codant pour Sox2) sont des cibles de la méthylation de l’ADN au cours de la différenciation cellulaire [
4–
6], ce qui suggère que la méthylation de l’ADN participe à la répression du programme de pluripotence au cours du développement (Figure 1A).
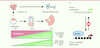 | Figure 1
La méthylation de l’ADN corrèle avec la perte de pluripotence. A. Au cours du développement embryonnaire chez la souris, la perte de pluripotence est concomitante de l’établissement des profils de méthylation de l’ADN dans les cellules en cours de différenciation. La méthylation de l’ADN est notamment dirigée vers des gènes-clés de la pluripotence, ce qui confirme les observations faites dans des modèles de différenciation de cellules ES in vitro. La méthylation de l’ADN est également ciblée vers un grand nombre de gènes méiotiques au cours de l’implantation de l’embryon. B. La méthylation des gènes de pluripotence et des gènes méiotiques dans les cellules somatiques différenciées est principalement catalysée par l’enzyme DNMT3B. Il est communément admis que cette méthylation n’est pas nécessaire pour initier la répression génique mais sert plutôt comme un mécanisme secondaire qui établit une répression stable à long terme et empêche la réactivation accidentelle. Un des défis dans les expériences de reprogrammation cellulaire qui génèrent des cellules souches pluripotentes induites (iPSC) est de surmonter efficacement cette barrière épigénétique. Les boules représentent les CpG dans un état méthylé (noir) ou non méthylé (blanc). |
|
Caractérisation des gènes cibles de la méthylation in vivo
De nombreux travaux indiquent que les cellules en culture peuvent accumuler des profils de méthylation de l’ADN anormaux. Il était donc important de confirmer in vivo les observations faites dans des systèmes de différenciation in vitro, ce qui est rendu difficile par la faible quantité de matériel biologique qui peut être collecté aux stades embryonnaires précoces. Afin de répondre à cette question, nous avons optimisé la technique de MeDIP1 (methylated DNA immunoprecipitation) pour de faibles quantités de cellules (< 10 000) et cartographié la méthylation de l’ADN de l’ensemble des promoteurs de gènes chez la souris au cours du développement péri-implantatoire, c’est-à-dire au moment où les profils de méthylation de l’ADN sont progressivement mis en place lors de l’acquisition de l’identité des lignages somatiques [
7]. Nos résultats montrent que les promoteurs de plusieurs gènes-clés de la pluripotence, comme ceux codant pour Oct4, Gdf3, Dppa3, Tcl1, Rex1, sont les cibles de la méthylation de l’ADN après l’implantation (Figure 1A), ce qui confirme les observations faites dans les modèles de cellules ES. Parmi les autres gènes méthylés durant l’implantation, on trouve un grand nombre de gènes spécifiques des cellules germinales, notamment des gènes méiotiques tels que ceux codant pour Dazl, Sycp1, Tex12, Spo11, Mei1. Ces gènes apparaissent statistiquement comme les cibles privilégiées de la méthylation de l’ADN dans l’embryon, ce qui indique qu’une des fonctions majeures de cette modification est de réprimer durablement le programme méiotique dans les cellules somatiques de l’embryon. Chez les mammifères, deux enzymes catalysent la méthylation de novo de l’ADN au cours du développement : DNMT3A et DNMT3B. Un travail récent de l’équipe de Claire Francastel a montré que DNMT3B est impliqué dans la méthylation et la répression de plusieurs gènes spécifiques des cellules germinales dans les tissus somatiques adultes chez la souris [
8]. En utilisant des souris knock-out pour chacune des enzymes, nous avons confirmé que DNMT3B plutôt que DNMT3A est la principale enzyme responsable de la méthylation des gènes méiotiques et des gènes de pluripotence dans les embryons post-implantatoires [7]. De plus, comme c’est le cas dans les tissus somatiques adultes, un certain nombre de ces gènes sont réactivés de manière aberrante dans les embryons Dnmt3b
−/−
, ce qui démontre que la méthylation de l’ADN est un facteur nécessaire pour établir une répression stable de ces gènes au cours de l’embryogenèse et empêcher leur réactivation accidentelle (Figure 1B). |
Déméthyler pour reprogrammer Ces résultats ont des implications dans le domaine de la reprogrammation cellulaire. Alors que la différenciation cellulaire est généralement considérée comme un processus unidirectionnel in vivo, il a été démontré qu’il est possible de reprogrammer in vitro des cellules différenciées en cellules pluripotentes (que l’on nomme cellules souches pluripotentes induites ou iPSC) en forçant l’expression de quelques facteurs de transcription [
9]. Cela implique la réactivation et donc la déméthylation, via des mécanismes encore mal compris, de gènes-clés de la pluripotence tels que ceux codant pour Oct4 et Nanog [
12]. Cependant il apparaît que cette reprogrammation épigénétique est très inefficace, ce qui contribue au fait que l’efficacité de reprogrammation cellulaire soit pour l’instant très faible (0,01-1 %). L’utilisation de composés pharmacologiques qui interfèrent avec la méthylation de l’ADN augmente l’efficacité de reprogrammation [
10], ce qui confirme que cette dernière constitue une barrière épigénétique qui stabilise l’identité cellulaire et empêche la reprogrammation. Comprendre quels sont les régulateurs et les gènes cibles de la méthylation de l’ADN en relation avec la perte de pluripotence apparaît donc nécessaire pour essayer d’améliorer l’efficacité de reprogrammation. De plus, outre celle des gènes associés à la pluripotence, les cellules différenciées présentent également une méthylation de certains gènes régulant d’importantes voies de différenciation. Récemment il a été montré que ces méthylations ne sont pas correctement reprogrammées lors du processus d’établissement des iPS et affectent le potentiel de différenciation de ces cellules iPSC qui gardent une mémoire épigénétique de leur origine [
11]. En résumé, il est donc fondamental d’essayer de mieux comprendre comment les profils de méthylation de l’ADN participent à établir et maintenir l’identité d’une cellule, ce qui permettra d’optimiser les technologies de reprogrammation cellulaire et facilitera leur utilisation thérapeutique. Conflit d’intérêts Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
Footnotes |
1.
Guibert
S
,
Forne
T
,
Weber
M
. Dynamic regulation of DNA methylation during mammalian development . Epigenomics.
2009; ; 1 : :81.-98. 2.
Jackson
M
,
Krassowska
A
,
Gilbert
N
, et al.
Severe global DNA hypomethylation blocks differentiation and induces histone hyperacetylation in embryonic stem cells . Mol Cell Biol.
2004; ; 24 : :8862.-8871. 3.
Okano
M
,
Bell
DW
,
Haber
DA
,
Haber
DA
,
Li
E
. DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development . Cell.
1999; ; 99 : :247.-257. 4.
Mohn
F
,
Weber
M
,
Rebhan
M
, et al.
Lineage-specific polycomb targets and de novo DNA methylation define restriction and potential of neuronal progenitors . Mol Cell.
2008; ; 30 : :755.-766. 5.
Laurent
L
,
Wong
E
,
Li
G
, et al.
Dynamic changes in the human methylome during differentiation . Genome Res.
2010; ; 20 : :320.-331. 6.
Farthing
CR
,
Ficz
G
,
Ng
RK
, et al.
Global mapping of DNA methylation in mouse promoters reveals epigenetic reprogramming of pluripotency genes . PLoS Genet.
2008; ; 4 : :e1000116.. 7.
Borgel
J
,
Guibert
S
,
Li
Y
, et al.
Targets and dynamics of promoter DNA methylation during early mouse development . Nat Genet.
2010; ; 42 : :1093.-100. 8.
Velasco
G
,
Hube
F
,
Rollin
J
, et al.
Dnmt3b recruitment through E2F6 transcriptional repressor mediates germ-line gene silencing in murine somatic tissues . Proc Natl Acad Sci USA.
2010; ; 107 : :9281.-9286. 9.
Takahashi
K
,
Yamanaka
S
. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors . cell.
2006; ; 126 : :663.-676. 10.
Huangfu
D
,
Maehr
R
,
Guo
W
, et al.
Induction of pluripotent stem cells by defined factors is greatly improved by small-molecule compounds . Nat Biotechnol.
2008; ; 26 : :795.-797. 11.
Lapasset
L
,
Milhavet
O
,
Lemaitre
J-M
. La reprogrammation vers la pluripotence peut-elle effacer la mémoire d’une vie antérieure ?
Med Sci (Paris).
2010; ; 26 : :902.-903. 12.
Coulombel
L
. Reprogrammation nucléaire d’une cellule différenciée. On efface tout et on recommence . Med Sci (Paris).
2007; ; 23 : :667.-670. 13.
Weber
M
. Profils de méthylation de l’ADN dans les cellules normales et cancéreuses . Med Sci (Paris).
2008; ; 24 : :731.-733. |