| |
| Med Sci (Paris). 2011 October; 27(10): 882–888. Published online 2011 October 21. doi: 10.1051/medsci/20112710017.Utilisation en routine du MALDI-TOF-MS pour l’identification des pathogènes en microbiologie médicale Étienne Carbonnelle1* and Xavier Nassif2 1Hôpital européen Georges Pompidou, AP-HP, Université Paris Descartes, Sorbonne Paris Cité, Faculté de médecine, Paris, France. 2Hôpital Necker-Enfants malades, AP-HP, Université Paris Descartes, Sorbonne Paris Cité, Faculté de médecine, Paris, France. |
Un impératif pour le laboratoire de microbiologie : gagner du temps pour une prise en charge optimale du patient infecté La mission première du laboratoire de microbiologie est d’assister le clinicien pour le diagnostic et la prise en charge du patient atteint de maladie infectieuse. Le laboratoire doit mettre en évidence et identifier l’agent pathogène responsable, étudier sa sensibilité aux différentes molécules antimicrobiennes, rechercher la présence de facteurs de virulence et, le cas échéant, comparer les différentes souches isolées afin d’établir un lien de clonalité notamment en cas d’épidémies. Répondre à toutes ces demandes nécessite la mise en route de différentes techniques dont certaines sont laborieuses, coûteuses et difficiles à réaliser en routine. Elles requièrent d’autre part un certain temps de manipulation, et ces délais ne sont pas toujours compatibles avec une prise en charge thérapeutique optimale du patient [
1]. Les laboratoires de microbiologie sont donc confrontés à un besoin urgent de « gagner du temps » sur le rendu des résultats, qu’ils concernent l’identification du pathogène ou l’étude de sa sensibilité aux antimicrobiens. En effet, l’identification rapide d’un agent pathogène au site du prélèvement conforte le clinicien dans son choix de débuter précocement un traitement probabiliste adapté, d’une part à la résistance naturelle du germe identifié, et d’autre part à l’écologie du service afin de réduire la morbidité et la mortalité. Cette prise en charge thérapeutique ciblée, qui sera adaptée secondairement en fonction du résultat de la sensibilité du pathogène aux antimicrobiens, permet de limiter l’utilisation empirique de molécules à large spectre et contribue à la réduction de l’émergence des résistances. Actuellement, l’identification et l’étude de la sensibilité des micro-organismes nécessitent plusieurs étapes qui reposent principalement sur la détection des caractéristiques phénotypiques du germe étudié. L’utilisation de colorations (coloration de Gram par exemple), la morphologie des colonies, l’examen au microscope, l’isolement en culture sur différents milieux, les tests biochimiques, qu’ils soient réalisés manuellement ou par des automates, sont les principes même de la classification et de l’identification des bactéries, des levures et des champignons. Ces approches, utilisées depuis de nombreuses années, permettent le rendu des résultats dans un laps de temps compris entre 24 h et 72 h, mais nécessitent, condition sine qua non, que les micro-organismes présentent un métabolisme actif, ce qui est heureusement le cas pour la grande majorité des prélèvements reçus. Ce délai peut être allongé lorsque la culture est lente et/ou difficile ou lorsque les tests phénotypiques sont peu performants, ce qui est le cas avec les mycobactéries, les germes atypiques ou les anaérobies par exemple. Dans ces derniers cas, il peut être de plusieurs jours à plusieurs semaines. En parallèle, des approches alternatives ont été développées pour les cas difficiles. L’utilisation de la biologie moléculaire, méthode de référence dans bien des cas, permet une identification bactérienne rapide et précise. Les avantages de la biologie moléculaire sont nombreux : elle s’affranchit de la culture, ce qui permet l’identification de micro-organismes non cultivables ou de croissance lente ; les résultats sont en règle générale obtenus assez rapidement et ce d’autant que la PCR en temps réel est utilisée. Malheureusement, les informations obtenues ne sont pas toujours suffisamment discriminantes pour permettre l’identification au niveau de l’espèce et l’étude d’autres gènes cibles est requise, ce qui augmente les délais de réponse. Outre les problèmes liés à son coût, cette méthodologie nécessite une grande expertise et doit être réservée aux identifications difficiles ou aux germes dont la culture est fastidieuse. |
Application de la spectrométrie de masse à l’identification de micro-organismes La spectrométrie de masse (MS), technique utilisée depuis la fin du XIXe siècle, a été appliquée dès les années 1970 à l’identification de micro-organismes [
2]. Le développement des techniques de spectrométrie de masse à ionisation douce comme le MALDI-TOF (matrix assisted laser desorption ionisation time-of-flight) ou l’ESI (electrospray ionisation) a permis l’analyse d’un large panel de biomarqueurs et l’essor de cette technologie [
3,
33]. Parallèlement, des spectromètres de masse moins complexes techniquement (mode linéaire) et moins coûteux se sont développés, facilitant leur diffusion au-delà des seuls laboratoires de recherche. L’engouement actuel pour l’utilisation du MALDI-TOF-MS en routine tient au fait que ce dernier peut être utilisé par l’ensemble des techniciens, qu’il est plus précis que les techniques phénotypiques classiques, d’un coût unitaire moindre pour chaque identification et très rapide, l’identification d’un pathogène ne nécessitant que quelques minutes. Une nouvelle approche associant la PCR à large spectre (ciblant les gènes codant pour les protéines ribosomales ou des gènes de ménage) et l’ESI (PCR-ESI-MS) se développe. Cette approche, qui s’applique directement au prélèvement, permet de s’affranchir de l’étape de culture. Elle est rapide (résultats en quelques heures), semi-quantitative et détecte un large panel de micro-organismes (bactéries, champignons et virus) [
4,
5]. Il faut cependant en évaluer la sensibilité et la spécificité. |
Principe Les applications de la spectrométrie de masse sont très vastes et concernent principalement l’identification de peptides ou de protéines, l’analyse de leur séquence en acides aminés ou encore la mise en évidence de modifications post-traductionnelles [ 33,
34]. Cette technologie largement utilisée depuis longtemps, s’implante depuis quelques années en microbiologie. Le principe général du MALDI-TOF-MS est simple : des ions de masse et de charge différentes soumis à un champ électrique se déplacent, et la distance parcourue en un temps donné est fonction du rapport masse sur charge (m/z). La première étape consiste à mélanger l’échantillon à la matrice, l’évaporation des solvants conduisant à la cristallisation de la matrice avec l’échantillon. Le mélange ainsi formé est déposé sur un support (plaque métallique). Plusieurs techniques de dépôt existent : les préparations en « couches minces » : l’échantillon est déposé sur une couche de matrice préalablement déposée sur la plaque cible pour former de larges films minces polycristallins ; les préparations en « gouttes épaisses » ou « gouttes sèches » : matrice et échantillon sont mélangés soit dans un tube puis déposés sur la plaque cible, soit directement sur la plaque cible ; les préparations en « sandwich » : l’échantillon est déposé sur un film de matrice avant d’être lui-même recouvert par une dernière couche de matrice. Une fois les échantillons déposés sur la cible, cette dernière est introduite dans le spectromètre de masse. Chaque dépôt est soumis à l’action du rayon laser UV. Le rôle de la matrice est d’absorber l’énergie provenant du laser ce qui provoque la vaporisation de l’échantillon avec formation d’ions de masses différentes. Les ions ainsi formés, généralement de charge +1 dans le cas du MALDI, vont être mis en mouvement sous l’action d’un champ électrique, et l’analyseur va les séparer en fonction de leur rapport m/z. Ils traversent ensuite un certain nombre de grilles d’extraction avant d’atteindre le « tube de vol » à l’extrémité duquel se trouve le détecteur. Les ions sont séparés selon leur temps de vol, ceux de petite taille atteignant les premiers le détecteur. Le temps de vol (time of flight) pour atteindre le détecteur est utilisé pour calculer la masse de chaque particule. La somme des ions analysés va former un spectre caractéristique de l’échantillon. Classiquement, l’axe des abscisses correspond au rapport masse sur charge (m/z) et l’axe des ordonnées à l’intensité relative du signal. Les premières applications de cette approche à l’identification de bactéries intactes remontent au milieu des années 1990 [
6]. Elles ne concernaient que quelques laboratoires de recherche et quelques espèces bactériennes. Depuis ces premiers travaux, les empreintes spectrales des principales espèces cultivables de bactéries, levures et champignons ont été obtenues. Préparation des échantillons Les paramètres expérimentaux : méthode d’extraction des protéines, concentration en NaCl, composition des tampons utilisés, conditions de culture, nature du dépôt (goutte séchée, couche mince, etc.) influencent la qualité des spectres en modifiant la cristallisation de la matrice avec l’échantillon et, par conséquent, l’efficacité d’ionisation des molécules testées. Maîtriser et standardiser ces variables permet d’optimiser l’identification et d’augmenter la reproductibilité. Le choix de la matrice utilisée est un paramètre important. Elle doit être adaptée à la nature de l’échantillon et répondre à certains critères physico-chimiques pour obtenir un bon rendement d’ionisation et un spectre de bonne résolution. Les matrices les plus utilisées sont l’acide 2,5-dihydroxybenzoïque (acide gentisique ou DHB), l’acide trans-3,5-diméthyloxy-4-hydroxycinnamique (acide sinapinique ou SA) et l’acide α-cyano-4-hydroxycinnamique (α-CHCA). À l’heure actuelle, le dépôt en goutte épaisse utilisant l’α-CHCA est largement utilisé pour les identifications de routine. En effet, la cristallisation obtenue est homogène contrairement à celle qui est obtenue avec la DHB, permet une utilisation automatique des acquisitions par le spectromètre et offre une excellente qualité de spectres. Dans son application en microbiologie, la technique du MALDI-TOF repose sur la détection de peptides ; les conditions de croissance des pathogènes jouent donc un rôle déterminant dans la variabilité et la qualité des spectres qui sont influencées de façon plus ou moins importante par les milieux de culture et le temps d’incubation [
7]. De même, la qualité du dépôt, le séchage et le mélange avec la matrice influencent la cristallisation. Le contrôle et la standardisation de ces paramètres permettent de limiter ces variations [ 6]. La nature exacte des peptides détectés est mal connue. Il s’agit essentiellement de protéines cytosoliques basiques, en quantité abondante, hydrophiles, dont les principales identifiées sont les protéines ribosomales ou les « cold shock proteins » [
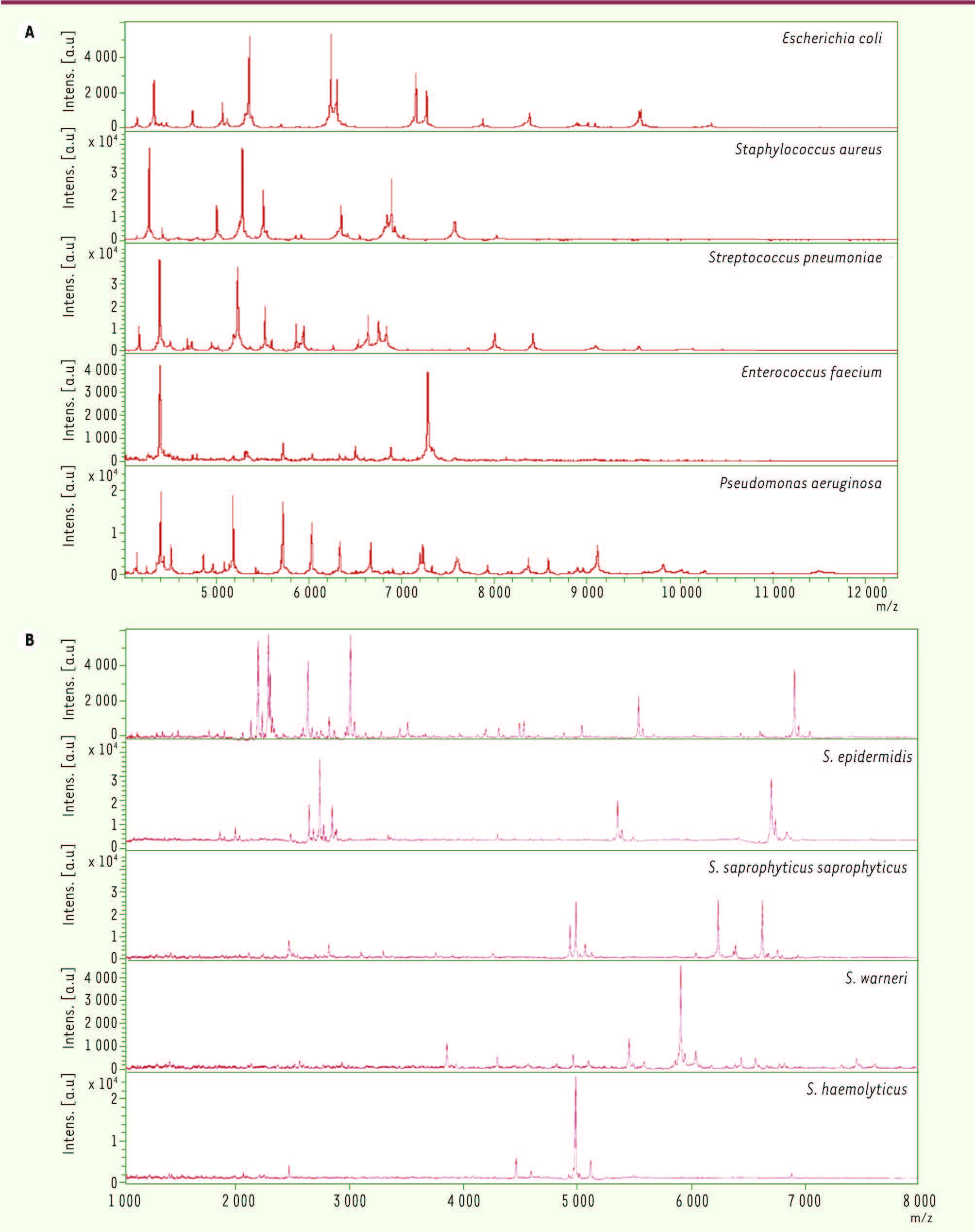
8]. Banques de données Deux types de banques de données L’identification des micro-organismes par cette technique repose sur la constatation que l’empreinte spectrale est différente d’un genre à l’autre et d’une espèce à l’autre
(Figures 1A,B)
. La réalisation des banques de données doit prendre en compte les variations de spectres afin de développer une stratégie fiable permettant une identification précise. Deux stratégies principales existent. La première consiste à créer une banque de données dans laquelle, pour une espèce donnée, l’essentiel des pics de plusieurs souches est conservé. Dans ce cas c’est la grande similitude entre le spectre de la bactérie à identifier et celui d’une souche dans la base de données qui permettra d’identifier l’espèce à tester. La seconde stratégie a pour objectif de sélectionner et de ne retenir que les pics spécifiques d’espèce, pics constants ayant une intensité relative élevée. Le spectre de la souche à tester est alors comparé aux pics spécifiques retenus dans la banque de données. Dans cette approche, le nombre de pics est plus restreint [
9]. La première stratégie impose d’avoir un très grand nombre de souches par espèce dans la banque de données à l’inverse de la seconde.
 | Figure 1.
A. Exemples de spectres. A. Empreintes spectrales obtenues à partir de colonies entières de cinq espèces bactériennes différentes, sans étape d’extraction. La matrice utilisée est l’α-CHCA. B. Empreintes spectrales obtenues à partir de colonies entières de cinq staphylocoques d’espèces différentes, sans étape d’extraction. La matrice utilisée est l’α-CHCA. |
Systèmes et bases de données disponibles Parmi les fabricants de spectromètre de masse, deux systèmes MALDI-TOF-MS complets sont disponibles à la vente, commercialisés par Bruker et Shimadzu. Chaque instrument est accompagné d’un logiciel de pilotage, d’une banque de données et d’un système expert permettant l’identification. L’identification du micro-organisme repose sur l’analyse du spectre généré par le spectromètre de masse et c’est cette analyse qui diffère en fonction des stratégies développées par les sociétés distributrices. La société Bruker a développé son propre système d’identification avec le MALDI BioTyper. Sa stratégie se concentre sur les pics ayant la plus forte intensité relative (au maximum une centaine de pics dont l’intensité relative est supérieure au bruit de fond). La société Shimadzu utilise la banque de donnée SARAMIS développée par la société AnagnosTec GmbH récemment reprise par BioMérieux. Leur approche se concentre sur les pics qui sont partagés par un nombre de souches de la même espèce (généralement 15) afin de créer une signature spectrale de référence ou « super spectre ». La société Andromas développe une base de données et un système expert compatibles avec les spectromètres de masse distribués par Bruker et Shimadzu. Le principe consiste à identifier les pics obtenus qui sont caractéristiques d’une espèce donnée. Ainsi la banque de données est constituée d’un nombre restreint de pics pour chaque espèce bactérienne et il existe pour chaque espèce bactérienne une seule ou un nombre restreint d’entrées. Cette banque de données couvre les germes habituels, mais aussi les mycobactéries, les dermatophytes ainsi que les différentes espèces d’Aspergillus et les levures [
10,
11]. Notons qu’actuellement, seule cette base de données offre la possibilité d’identifier les mycobactéries. Par ailleurs, une seule étude comparant les deux systèmes Bruker et Shimadzu en utilisation de routine étant publiée, il est difficile de tirer des conclusions sur les performances. Les identifications de l’espèce des pathogènes analysés étaient correctes dans 93,6 % et 88,3 % des échantillons pour Bruker et Shimadzu respectivement [
12]. Application en microbiologie de routine Plusieurs laboratoires de microbiologie hospitalière mais aussi d’analyse médicale se sont récemment équipés de MALDI-TOF-MS, faisant le choix d’abandonner les tests phénotypiques classiques. L’implantation de cette technologie ne semble pas poser de problème majeur. Les spectromètres de masse, grâce au logiciel de pilotage, sont simples à utiliser et leur prise en main par l’ensemble du personnel aussi bien technique que médical se fait en douceur. Le point le plus délicat sur le plan technique concerne le mélange échantillon/matrice et son dépôt sur la plaque métallique de support. Un excès d’échantillon de même qu’un mauvais dépôt peuvent altérer la qualité du spectre et rendre l’identification difficile. Une certaine précision est nécessaire afin de ne pas contaminer l’échantillon voisin. Une fois ces paramètres maîtrisés, les performances varient en fonction des espèces. Dans les études analysant les résultats obtenus en routine, le pourcentage d’identification correcte au genre des espèces les plus fréquemment isolées varie de 87 % à 99 %
(Tableau 1)
[
13–
18]. Les entérobactéries ne posent pas de problème majeur avec une identification correcte de l’espèce dans 97 % à 99 % des échantillons selon les études. Un point délicat reste l’identification des Shigella sp. dont les spectres ne sont pas présents dans toutes les bases de données [ 13,
15]. Parmi les cocci à Gram positif, les streptocoques du groupe mitis sont particulièrement difficiles à identifier et à distinguer de Streptococcus pneumoniae [ 13– 15].
Tableau I.
| Auteurs |
Échantillons |
Id à l’espèce |
Id au genre |
Identifications
difficiles |
Commentaires |
| Seng et al., 2009 [13] |
Routine (n = 1 660) |
Tous les prélèvements de routine |
83,8 % |
95,0 % |
Propionobacterium acnes
|
Dans cette étude, le MALDI-TOF est utilisé d’emblée pour l’identification. |
|
|
|
|
|
Streptococcus pneumoniae
|
|
|
|
|
|
|
Stenotrophomonas maltophilia
|
|
|
|
|
|
|
Shigella sp. |
|
|
| Blondiaux et al., 2009 [15] |
Routine (n =362) |
Tous les prélèvements de routine |
72,9 % |
87,0 % |
Streptococcus du groupe viridans |
|
|
|
|
|
|
Shigella sp. |
|
|
| van Veen et al., 2010 [
14] |
Routine (n =980) |
Tous les prélèvements de routine |
92,0 % |
98,8 % |
Streptococcus pneumoniae
|
|
|
|
|
|
|
Bactéries anaérobies |
|
|
| Bizzini et al., 2010 [
16] |
Routine (n = 1 371) |
Tous les prélèvements de routine |
93,2 % |
98,5 % |
Shigella sp. |
Une étape d’extraction permet d’augmenter le % de résultats valides de 22,9 % |
|
|
|
|
|
Streptococcus sp. |
Erreurs de taxonomie essentiellement. |
|
| Gravet et al., 2010 [
17] |
Routine (n = 10 000) |
Tous les prélèvements de routine |
nd |
98,8 % |
Corynebacterium sp. |
Dans cette étude, le MALDI-TOF est utilisé d’emblée pour l’identification. |
|
|
|
|
|
Streptococcus sp. |
|
|
| Bessède et al., 2010 [18] |
Routine (n = 1 013) |
Tous les prélèvements de routine |
97,3 % |
99,0 % |
Acinetobacter sp. |
Une étape d’extraction permet d’augmenter le % d’identification à l’espèce de 14,7 % |
|
|
|
|
|
Streptococcus pneumoniae
|
Nombre insuffisant de souches dans la banque de données pour certaines espèces |
|
| Prod’hom et al., 2010 [
24] |
Sang (n = 126) |
Hémocultures positives |
77,8 % |
78,7 % |
Streptococcus mitis group |
La présence d’une capsule peut en partie expliquer le plus faible taux d’identification pour les bactéries :S. pneumoniae, H. influenzae, K. pneumoniae
|
|
|
|
GN : 89,1 % |
GN : 89,1 % |
Staphylococcus sp. |
|
|
|
|
GP : 71,6 % |
GP : 72,9 % |
|
|
|
| La Scola et al., 2009 [
19] |
Sang (n = 599) |
Hémocultures positives |
76,0 % |
76,0 % |
Streptococcus sp. |
|
|
|
|
|
|
Échantillons polymicrobiens |
|
|
| Stevenson et al., 2009 [
20] |
Sang (n = 212) |
Hémocultures positives (179)
|
80,2 % |
80,2 % |
Streptococcus du groupe mitis
|
|
|
|
Flacons ensemencés (33) |
|
|
Propionobacterium acnes
|
|
|
| Ferroni et al., 2010 [
21] |
Sang (n= 685) |
Hémocultures positives (388)
|
89,0 % |
98,0 % |
Streptococcus pneumoniae
|
En cas d’hémoculture polymicrobienne, le germe prédominant est dans la plupart des cas identifié |
|
|
Flacons ensemencés (312) |
|
|
Streptococcus du groupe mitis
|
Méthode très rapide |
|
| Christner et al., 2010 [
22] |
Sang (n = 277) |
Hémocultures positives |
94,2 % |
95,0 % |
Cocci Gram+
|
Les difficultés d’identification résultent d’un nombre insuffisant de bactéries, plus fréquent avec les Gram+
|
|
| Ferreira et al., 2010 [
26] |
Sang (n = 300) |
Hémocultures positives |
42,6 % |
71,6 |
Streptococcus mutans
|
Pas de culture polymicrobienne |
|
|
|
GN : 83,3 % |
GN : 96,6 % |
Staphylococcus sp. |
|
|
|
|
GP : 31,8 % |
GP : 65,7 % |
Staphylococcus aureus
|
|
|
| Moussaoui et al., 2010 [
25] |
Sang (n = 532) |
Hémocultures positives |
90,0 % |
nd |
Streptococcus groupe mitis
|
|
|
|
|
GN : 91,1 % |
|
Staphylococcus sp. |
|
|
|
|
GP : 89 % |
|
|
|
|
| Ferreira et al., 2010 [
23] |
Urine (n = 220) |
Urines positives |
91,8 % |
92,7 % |
Streptococcus sp. |
Meilleurs résultats avec un inoculum> 105 CFU/ml |
|
|
|
GN : 93,6 % |
GN : |
Enterococcus sp. |
E. coli > 105 CFU/ml : 97,6 % |
|
|
|
GP : 66,6 % |
94,6 % |
Raoultella sp. |
Identifications correctes |
|
|
|
|
GP : |
|
|
|
|
|
|
66,6 % |
|
|
Résumé des principales études utilisant le MALDI-TOF en routine pour l’identification bactérienne. GN : Gram- ; GP : Gram+ ; nd : non disponible ; Id : identification. |
Une autre application de cette technologie est l’identification des bactéries directement à partir des prélèvements, soit à partir de flacons d’hémocultures détectés comme positifs [19–25], soit à partir des urines [23]. Le gain de temps est alors très important pour la mise en route d’une antibiothérapie. Concernant les hémocultures, les résultats sont obtenus avec les protocoles les plus rapides en une vingtaine de minutes après quelques étapes intermédiaires qui permettent la séparation des globules blancs des globules rouges. Les pourcentages d’identification correcte du genre varient de 72 % à 98 % selon les études
(Tableau I)
. On retrouve les mêmes difficultés d’identification que celles que l’on rencontre lorsqu’on travaille à partir des colonies. Dans le cas des hémocultures polymicrobiennes, les spectres obtenus sont plus difficiles à interpréter et, dans le meilleur des cas, le germe prédominant est identifié. Pour les urines, les résultats sont très encourageants à condition d’avoir un inoculum bactérien supérieur à 105 CFU/mL [23]. Principales difficultés rencontrées Cette technique soulève parfois des difficultés d’identification, plus souvent rencontrées avec les bactéries à Gram positif
(Tableau 1)
. Dans la plupart des cas, la qualité du dépôt est en cause. Un excès de bactéries ou de levures dans le mélange avec la matrice, un dépôt non homogène sur la cible peuvent créer des spectres de mauvaise qualité (Figure 3). Dans certains cas, il est au contraire nécessaire de faire des dépôts plus riches, par exemple en présence de souches isolées des muqueuses ( Pseudomonas aeruginosa) ou de bactéries difficiles à prélever à partir du milieu de culture ( Nocardia sp). Ceci est aussi vrai pour certaines espèces comme les Propionibacterium sp., ou l’utilisation de milieux chromogènes, conditions dans lesquelles le nombre de pics obtenus dans le spectre est plus faible, ce qui rend plus délicate l’interrogation des banques de données. Il est par ailleurs fortement déconseillé d’appliquer cette technique à des colonies obtenues autour des disques d’antibiotiques (antibiogramme en diffusion), les spectres étant plus difficiles à interpréter. Pour faciliter l’identification, les cultures obtenues en 24 h sont préférées aux cultures obtenues en 48 h. Quant à l’identification des champignons filamenteux, elle requiert la présence de spores. Certaines études proposent la réalisation d’une étape d’extraction protéique avant l’analyse afin d’augmenter la qualité des spectres. Cette étape supplémentaire peut se justifier pour certaines espèces comme les streptocoques, ou en cas d’inoculum faible notamment pour les hémocultures ou les urines [16, 18,
27]. Perspectives : détection de la résistance et des facteurs de virulence Il est envisageable, à partir des spectres obtenus, de développer de nouvelles recherches afin d’identifier des protéines spécifiques ou des marqueurs prédictifs de la présence de facteurs de virulence ou de la résistance aux antimicrobiens 1 [
35–
37]. L’identification de la leucocidine de Panton-Valentine de Staphylococcus aureus par exemple a fait l’objet de différentes recherches dont les conclusions demeurent contradictoires, soulignant plus que jamais l’importance de la standardisation des différentes acquisitions [
28,
29]. En effet, certains pics peuvent ne pas être systématiquement présents, des variations apparaissant en fonction du mode opératoire. Dans ce genre de travail, avant d’affirmer qu’un pic est lié à la présence d’un facteur de virulence, il est impératif de réaliser plusieurs dépôts et acquisitions indépendants, voire d’identifier précisément la nature du peptide d’intérêt. De même dans l’avenir, le MALDI-TOF-MS pourrait permettre l’identification des protéines spécifiques de la résistance comme par exemple les β-lactamases [
30, 35,
36]. Plusieurs études ont tenté de différencier les souches de Staphylococcus aureus résistantes à la méticilline (SARM) des souches sensibles (SASM). Les différents résultats obtenus n’ont pas permis de prédire et d’identifier avec suffisamment de précision les deux types de souches [
31,
32, 37]. |
La spectrométrie de masse de type MALDI-TOF s’impose progressivement et massivement dans les laboratoires de microbiologie. Ses atouts sont nombreux, rapidité et précision d’identification, prise en main et utilisation aisées, faible coût par échantillon [13]. Certes l’achat et l’entretien d’un tel système peuvent apparaître onéreux, mais la balance à long terme est très en faveur du MALDI-TOF. En effet, une identification rapide et précise faite directement à partir des prélèvements permet une optimisation de la prise en charge du patient. Ceci se traduit par une réduction de la prescription empirique d’anti-infectieux à large spectre dont les conséquences sont multiples [
38] : lutte contre l’émergence des résistances, diminution du coût des traitements, diminution des journées d’hospitalisation. Par ailleurs, les tests phénotypiques, les galeries d’identification et autres kits d’agglutination sont remplacés, diminuant d’autant les dépenses des laboratoires. À l’heure actuelle, il est impératif d’évaluer et de comparer les différents systèmes disponibles quant à leurs performances techniques, à la précision d’identification, aux mises à jour et à la maintenance des banques de données. Enfin, le champ d’investigation de cette technologie reste très largement ouvert permettant d’envisager dans un avenir proche de comparer les souches, d’identifier des facteurs de virulence ou encore d’étudier la résistance aux antimicrobiens. Conflit d’intérêts
E. Carbonnelle et X. Nassif déclarent avoir une participation financière dans le capital de l’entreprise Andromas.
|
Footnotes |
-
Peters
RP
,
van Agtmael
MA
,
Danner
SA
, et al.
New developments in the diagnosis of bloodstream infections . Lancet Infect Dis.
2004; ; 4 : :751.–760.
-
Anhalt
JP
,
Fenselau
C.
Identification of bacteria using mass spectrometry . Anal Chem.
1975; ; 47 : :219.–225.
-
Karas
M
,
Hillenkamp
F.
Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons . Anal Chem.
1988; ; 60 : :2299.–2301.
-
Ecker
DJ
,
Sampath
R
,
Massire
C
, et al.
Ibis T5000 : a universal biosensor approach for microbiology . Nat Rev Microbiol.
2008; ; 6 : :553.–558.
-
Ecker
DJ
,
Sampath
R
,
Li
H
, et al.
New technology for rapid molecular diagnosis of bloodstream infections . Expert Rev Mol Diagn.
2010; ; 10 : :399.–415.
-
Claydon
MA
,
Davey
SN
,
Edwards-Jones
V
,
Gordon
DB.
The rapid identification of intact microorganisms using mass spectrometry . Nat Biotechnol.
1996; ; 14 : :1584.–1586.
-
Valentine
N
,
Wunschel
S
,
Wunschel
D
, et al.
Effect of culture conditions on microorganism identification by matrix-assisted laser desorption ionization mass spectrometry . Appl Environ Microbiol.
2005; ; 71 : :58.–64.
-
Ryzhov
V
,
Fenselau
C.
Characterization of the protein subset desorbed by MALDI from whole bacterial cells . Anal Chem.
2001; ; 73 : :746.–750.
-
Carbonnelle
E
,
Beretti
JL
,
Cottyn
S
, et al.
Rapid identification of Staphylococci isolated in clinical microbiology laboratories by matrix-assisted laser desorption ionization-time of flight mass spectrometry . J Clin Microbiol.
2007; ; 45 : :2156.–2161.
-
Lotz
A
,
Ferroni
A
,
Beretti
JL
, et al.
Rapid identification of mycobacterial whole cells in solid and liquid culture media by MALDI-TOF MS . J Clin Microbiol.
2010; ; 48 : :4481.–4486.
-
Alanio
A
,
Beretti
JL
,
Dauphin
B
, et al.
MALDI-TOF Mass spectrometry for fast and accurate identification of clinically relevant Aspergillus species . Clin Microbiol Infect.
2011; ; 17 : :750.–755.
-
Cherkaoui
A
,
Hibbs
J
,
Emonet
S
, et al.
Comparison of two matrix-assisted laser desorption ionization-time of flight mass spectrometry methods with conventional phenotypic identification for routine identification of bacteria to the species level . J Clin Microbiol.
2010; ; 48 : :1169.–1175.
-
Seng
P
,
Drancourt
M
,
Gouriet
F
, et al.
Ongoing revolution in bacteriology : routine identification of bacteria by matrix-assisted laser desorption ionization time-of-flight mass spectrometry . Clin Infect Dis.
2009; ; 49 : :543.–551.
-
Van Veen
SQ
,
Claas
EC
,
Kuijper
EJ.
High-throughput identification of bacteria and yeast by matrix-assisted laser desorption ionization mass spectrometry (MALDI-TOF MS) in routine medical microbiology laboratory . J Clin Microbiol.
2010; ; 48 : :900.–907.
-
Blondiaux
N
,
Gaillot
O
,
Courcol
RJ.
MALDI-TOF mass spectrometry to identify clinical bacterial isolates : Evaluation in a teaching hospital . Pathol Biol (Paris).
2010; ; 58 : :55.–57.
-
Bizzini
A
,
Durussel
C
,
Bille
J
, et al.
Performance of matrix-assisted laser desorption ionization-time of flight mass spectrometry for identification of bacterial strains routinely isolated in a clinical microbiology laboratory . J Clin Microbiol.
2010; ; 48 : :1549.–1554.
-
Gravet
A
,
Camdessoucens-Miehe
G
,
Gessier
M
, et al.
The use in routine of mass spectrometry in a hospital microbiology laboratory . Pathol Biol (Paris).
2011; ; 59 : :19.–25.
-
Bessede
E
,
Angla-Gre
M
,
Delagarde
Y
, et al.
MALDI Biotyper, experience in the routine of a University hospital . Clin Microbiol Infect.
2011; ; 17 : :533.–538.
-
La Scola
B
,
Raoult
D.
Direct identification of bacteria in positive blood culture bottles by matrix-assisted laser desorption ionisation time-of-flight mass spectrometry . PLoS One .
2009; ; 4 : :e8041..
-
Stevenson
LG
,
Drake
SK
,
Murray
PR.
Rapid identification of bacteria in positive blood culture broths by MALDI-TOF mass spectrometry . J Clin Microbiol.
2009; ; 48 : :444.–447.
-
Ferroni
A
,
Suarez
S
,
Beretti
JL
, et al.
Real-time identification of bacteria and Candida species in positive blood culture broths by matrix-assisted laser desorption ionization-time of flight mass spectrometry . J Clin Microbiol.
2010; ; 48 : :1542.–1548.
-
Christner
M
,
Rohde
H
,
Wolters
M
, et al.
Rapid identification of bacteria from positive blood culture bottles by use of matrix-assisted laser desorption-ionization time of flight mass spectrometry fingerprinting . J Clin Microbiol.
2010; ; 48 : :1584.–1591.
-
Ferreira
L
,
Sanchez-Juanes
F
,
Gonzalez-Avila
M
, et al.
Direct identification of urinary tract pathogens from urine samples by matrix-assisted laser desorption ionization-time of flight mass spectrometry . J Clin Microbiol.
2010; ; 48 : :2110.–2115.
-
Prod’hom
G
,
Bizzini
A
,
Durussel
C
, et al.
MALDI-TOF mass spectrometry for direct bacterial identification from positive blood culture pellets . J Clin Microbiol.
2010; ; 48 : :1481.–1483.
-
Moussaoui
W
,
Jaulhac
B
,
Hoffmann
AM
, et al.
Matrix-assisted laser desorption ionization time-of-flight mass spectrometry identifies 90 % of bacteria directly from blood culture vials . Clin Microbiol Infect.
2010; ; 16 : :1631.–1638.
-
Ferreira
L
,
Sanchez-Juanes
F
,
Guerra
IP
, et al.
Microorganisms Direct Identification from blood culture by Maldi-Tof mass spectrometry . Clin Microbiol Infect.
2011; ; 17 : :546.–551.
-
Ferreira
L
,
Sanchez-Juanes
F
,
Munoz-Bellido
JL
,
Gonzalez-Buitrago
JM.
Rapid method for direct identification of bacteria in urine and blood culture samples by MALDI-TOF MS : intact cell vs. extraction method . Clin Microbiol Infect.
2011; ; 17 : :1007.–1012.
-
Bittar
F
,
Ouchenane
Z
,
Smati
F
, et al.
MALDI-TOF-MS for rapid detection of staphylococcal Panton-Valentine leukocidin . Int J Antimicrob Agents.
2009; ; 34 : :67.–70.
-
Dauwalder
O
,
Carbonnelle
E
,
Benito
Y
, et al.
Detection of Panton-Valentine toxin in Staphylococcus aureus by mass spectrometry directly from colony : time has not yet come . Int J Antimicrob Agents.
2010; ; 36 : :193.–194.
-
Camara
JE
,
Hays
FA.
Discrimination between wild-type and ampicillin-resistant Escherichia coli by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry . Anal Bioanal Chem.
2007; ; 389 : :1633.–1638.
-
Du
Z
,
Yang
R
,
Guo
Z
, et al.
Identification of Staphylococcus aureus and determination of its methicillin resistance by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry . Anal Chem.
2002; ; 74 : :5487.–5491.
-
Jackson
KA
,
Edwards-Jones
V
,
Sutton
CW
,
Fox
AJ.
Optimisation of intact cell MALDI method for fingerprinting of methicillin-resistant Staphylococcus aureus . J Microbiol Methods.
2005; ; 62 : :273.–284.
-
Gonçalves
A
,
Bertucci
F
,
Birnbaum
D
,
Borg
JP.
,
Profiling protéique SELDI-TOF et cancer du sein : applications cliniques potentielles . Med Sci (Paris).
2007; ; 23 ( suppl 1) : :23.–26.
-
Emadali
A
,
Gallagher-Gambarelli
M.
La protéomique quantitative par la méthode SILAC : technique et perspectives . Med Sci (Paris).
2009; ; 25 : :835.–842.
-
Nordmann
P.
Résistance aux carbapénèmes chez les bacilles à Gram négatif . Med Sci (Paris).
2010; ; 26 : :950.–959.
-
Barbier
F
,
Wolff
M.
Multirésistance chez Pseudomonas aeruginosa : vers l’impasse thérapeutique ?
Med Sci (Paris).
2010; ; 26 : :960.–968.
-
Dumitrescu
O
,
Dauwalder
O
,
Boisset
S
, et al.
Résistance aux antibiotiques chez Staphylococcus aureus : les points-clés en 2010 . Med Sci (Paris).
2010; ; 26 : :943.–949.
-
Trémolières
F.
Quand le miracle antibiotique vire au cauchemar . Med Sci (Paris).
2010; ; 26 : :925.–929.
|