| |
| Med Sci (Paris). 2012 April; 28(4): 372–374. Published online 2012 April 25. doi: 10.1051/medsci/2012284014.Pluripotence des cellules souches Quand l’épissage alternatif s’en mêle Mathieu Gabut1* 1University of Toronto, Terrence Donnelly centre for cellular and biomolecular research, 160 College street, room 1030, TorontoM5S 3E1, Ontario, Canada MeSH keywords: Épissage alternatif, génétique, physiologie, Animaux, Différenciation cellulaire, Facteurs de transcription Forkhead, Régulation de l'expression des gènes au cours du développement, Humains, Modèles biologiques, Cellules souches pluripotentes, métabolisme, Transcriptome |
Le contrôle de la pluripotence des cellules souches Les cellules souches embryonnaires (CSE) sont issues du blastocyste et sont capables de s’autorenouveler théoriquement indéfiniment sous forme indifférenciée. En outre, les CSE sont pluripotentes, ce qui signifie qu’elles peuvent se différencier en n’importe quel type cellulaire composant les tissus embryonnaires et que, en interaction étroite avec le placenta, elles ont la capacité de former un organisme viable et fertile. Les CSE possèdent un programme d’expression génique unique favorisant à la fois leur autorenouvellement et le maintien de leur pluripotence. Ce programme est minutieusement contrôlé à différentes étapes de l’expression des gènes et conserve le génome dans une configuration dynamique qui favorise une activation rapide des gènes impliqués dans la différenciation en réponse à des signaux développementaux. La régulation de ce programme intervient tout d’abord au niveau de la chromatine, par le biais de la méthylation de l’ADN, du remodelage des nucléosomes et de modifications post-traductionnelles des histones. De nombreuses études ont également établi qu’un nombre limité de facteurs, dont Oct4/POU5F1 (POU class 5 homeobox), Sox2 (SRY [sex determining region Y]-box 2) et Nanog, constitue un réseau de régulateurs de la transcription qui joue un rôle clé dans le maintien des propriétés des cellules souches ou dans leur réacquisition dans le modèle des cellules pluripotentes induites ou iPSC (induced pluripotent stem cells) [1]. En effet, ces facteurs de transcription possèdent une double fonction dans les CSE indifférenciées : d’une part, ils stimulent leur propre expression par le biais de boucles autorégulatrices et celle de gènes nécessaires au maintien de la pluripotence et, d’autre part, ils répriment la transcription de gènes impliqués dans les programmes de différenciation cellulaire [2]. Ce réseau contrôle également l’expression de nombreux microARN qui contribuent au maintien des CSE, et répriment, avec l’aide de protéines du complexe Polycomb, l’expression de microARN normalement exprimés au cours de la différenciation [2]. Au cours des cinq dernières années, un mécanisme supplémentaire de régulation du destin des CSE a été mis en évidence, l’épissage alternatif, dont l’importance reste encore à définir. |
L’épissage alternatif : un mécanisme clé de l’expression des gènes L’épissage alternatif est une étape cruciale de la maturation des ARNm chez les eucaryotes. Elle permet l’inclusion différentielle de séquences exoniques dans les ARNm matures et favorise ainsi la synthèse de plusieurs isoformes d’ARNm à partir d’un gène unique. L’épissage alternatif est régulé au cours du développement et permet d’accroître la diversité du transcriptome et du protéome exprimés dans chaque type cellulaire [3]. Chez l’homme, des analyses de séquençage ARN à haut débit (RNA-seq) ont montré que l’épissage alternatif concerne plus de 95 % des gènes multi-exons, et que les gènes dont l’expression est régulée au niveau transcriptionel sont essentiellement différents des gènes régulés par cette voie [4, 5]. L’épissage alternatif constitue donc un mécanisme de régulation de l’expression des gènes à part entière, au même titre que la régulation de la transcription, par exemple. Les technologies des puces à ADN, et plus récemment du RNA-seq, ont permis l’identification de nombreuses isoformes d’ARNm dont l’épissage alternatif est régulé de façon différente entre les CSE et des populations de cellules différenciées qui dérivent de ces cellules souches. Cependant, à l’exception de quelques événements d’épissage alternatif qui affectent l’activité de facteurs de transcription importants pour le maintien de la pluripotence, tels que Oct4, Tcf3 (transcription factor 3), Sall4 (Sal[Drosophila]-like 4) et Nanog [6, 7], la fonction de la quasi-totalité de ces isoformes reste inconnue à ce jour. |
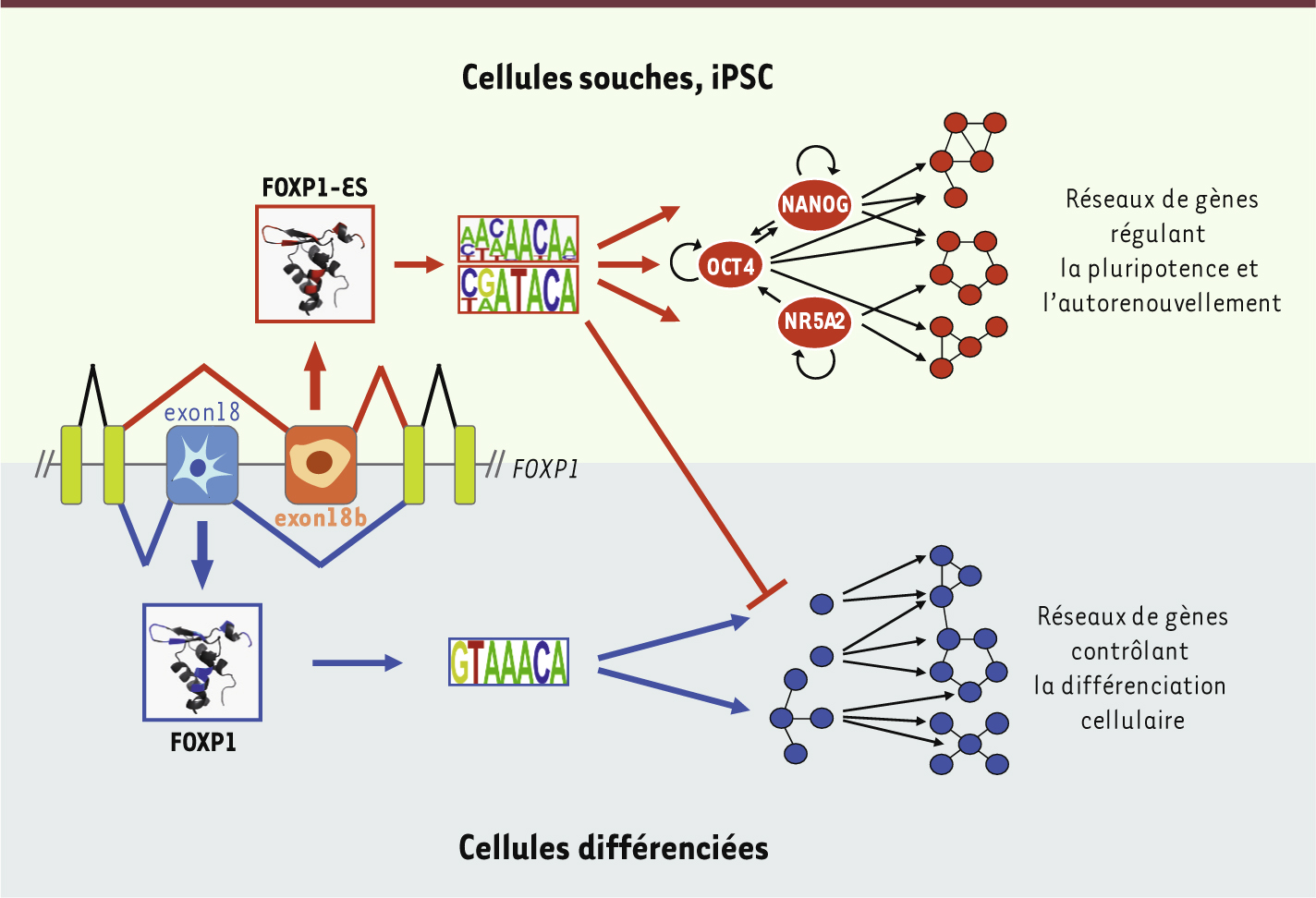
L’épissage des ARNm du gène FOXP1 : un interrupteur de la pluripotence Afin de déterminer la contribution de l’épissage alternatif dans la régulation des CSE humaines, nous avons utilisé des puces à ADN qui permettent de détecter simultanément plus de 5 000 événements d’épissage. Cette approche a identifié plus de 240 exons dont l’épissage est différentiellement régulé entre les CSE pluripotentes et leurs dérivés au cours de leur différenciation en endoderme, mésoderme, et ectoderme [8]. Un événement d’épissage a retenu notre attention, car il implique un exon non annoté dans le gène FOXP1 codant pour un facteur de transcription de la famille Forkhead. Nous avons démontré que cet exon 18b est spécifiquement inclus dans les CSE humaines (FOXP1-ES) et progressivement remplacé au cours de la différenciation cellulaire par l’exon 18 qui, lui, est détecté dans la quasi-totalité des tissus adultes (Figure 1). De façon remarquable, le profil d’épissage alternatif de l’exon 18b est conservé au cours de l’évolution, en particulier dans les CSE murines, mais également dans le gène ancestral FoxP chez les invertébrés [9], ce qui suggère qu’il joue un rôle important au cours du développement. En effet, les exons 18 et 18b codent pour une partie du domaine de liaison à l’ADN des isoformes FOXP1 et FOXP1-ES, respectivement, et modifient la spécificité d’interaction à l’ADN de ces deux isoformes. Une analyse, réalisée grâce à des puces ADN de type PBM (protein binding microarray) représentant toutes les combinaisons possibles de séquences de huit nucléotides, a confirmé que la protéine FOXP1 reconnaît un motif GTAAACA, alors que FOXP1-ES s’associe préférentiellement à des motifs CGATACA et des motifs riches en A/C (Figure 1), ce qui suggère que ces deux isoformes contrôlent des programmes génétiques distincts. L’utilisation de siARN (small interfering) dirigés spécifiquement contre les exons 18 et 18b, couplée à une analyse par RNA-seq, a révélé que chaque isoforme de FOXP1 régule des réseaux de gènes différents dans les CSE humaines. En particulier, FOXP1-ES réprime l’expression de nombreux gènes impliqués dans la différenciation cellulaire et stimule l’expression de OCT4, NANOG et NR5A2 (nuclear receptor subfamily 5, group A, member 2), trois facteurs de transcription clés pour les propriétés de pluripotence.
 | Figure 1. Modèle de la régulation de l’épissage et de la fonction des isoformes FOXP1-ES et FOXP1 dans les cellules souches pluripotentes ou différenciées, respectivement. Les exons 16 à 20 du gène FOXP1 sont représentés. La spécificité de liaison à l’ADN de FOXP1 et FOXP1-ES est indiquée sous forme de séquences consensus dont la taille des lettres représente la fréquence observée des différents nucléotides à chaque position dans les motifs ADN. |
Enfin, nous avons démontré que l’isoforme Foxp1-ES, mais pas Foxp1, est requise pour le maintien de la pluripotence et de l’autorenouvellement des CSE murines, ainsi que pour la reprogrammation de fibroblastes embryonnaires murins en cellules iPSC. L’ensemble de ces données démontre qu’un événement d’épissage alternatif dans le gène FOXP1 se positionne en amont du réseau de facteurs de transcription qui contrôle les programmes génétiques spécifiques des CSE, et se comporte comme un interrupteur qui régule le maintien des propriétés des cellules souches ou leur différenciation. |
L’épissage alternatif : un régulateur clé du transcriptome et du devenir des cellules souches ? Les études réalisées au cours des trois dernières années grâce au RNA-seq ont permis de mettre en évidence que les cellules pluripotentes, et en particulier les CSE, possèdent un transcriptome très diversifié, et expriment de nombreux ARN qui diffèrent des modèles de transcrits ou d’ADNc actuellement établis [10]. Ces résultats impliquent également que la diversité du protéome des CSE et la fonction de la vaste majorité de ces ARN restent encore à définir. En effet, les rares exemples d’événements d’épissage alternatif décrits pour des facteurs de transcription tels que Nanog, Oct4, Tcf3 et Foxp1 démontrent clairement que des variations subtiles de domaines fonctionnels jouent un rôle déterminant pour reprogrammer la fonction de ces gènes, indépendamment de la régulation de leur transcription. Ces exemples soulignent l’importance d’étudier la diversité du transcriptome des CSE d’un point de vue non seulement descriptif, mais aussi fonctionnel, et suggèrent que les modèles actuellement établis de réseaux de gènes assurant le contrôle des capacités de pluripotence et d’autorenouvellement des CSE vont être profondément remaniés. Ces études suscitent également un intérêt important pour identifier les mécanismes qui définissent la diversité du protéome des CSE et contribuent à maintenir l’identité pluripotente des cellules souches. Enfin, elles soulignent l’importance de caractériser les facteurs qui régulent le transcriptome en réponse à des cascades de signalisation lors de la différenciation cellulaire. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Cette étude a été réalisée avec l’appui des instituts de recherche en santé du Canada (IRSC) et le soutien du Pr Benjamin Blencowe de l’Université de Toronto. Je tiens à remercier les Drs Johann Soret et Éric Lécuyer pour leurs commentaires sur le manuscrit.
|
1. Yamanaka S, Blau HM. Nuclear reprogramming to a pluripotent state by three approaches . Nature. 2010; ; 465 : :704.–712. 2. Young RA. Control of the embryonic stem cell state . Cell. 2011; ; 144 : :940.–954. 3. Nilsen TW, Graveley BR. Expansion of the eukaryotic proteome by alternative splicing . Nature. 2010; ; 463 : :457.–463. 4. Pan Q, Shai O, Lee LJ, et al. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing . Nat Genet. 2008; ; 40 : :1413.–1415. 5. Wang ET, Sandberg R, Luo S, et al. Alternative isoform regulation in human tissue transcriptomes . Nature. 2008; ; 456 : :470.–476. 6. Cheong CY, Lufkin T. Alternative splicing in self-renewal of embryonic stem cells . Stem Cells Int. 2011; ; 2011 : :560261.. 7. Das S, Jena S, Levasseur DN. Alternative splicing produces Nanog protein variants with different capacities for self-renewal and pluripotency in embryonic stem cells . J Biol Chem. 2011; ; 286 : :42690.–42703. 8. Gabut M, Samavarchi-Tehrani P, Wang X, et al. An alternative splicing switch regulates embryonic stem cell pluripotency and reprogramming . Cell. 2011; ; 147 : :132.–146. 9. Santos ME, Athanasiadis A, Leitao AB, et al. Alternative splicing and gene duplication in the evolution of the FoxP gene subfamily . Mol Biol Evol. 2011; ; 28 : :237.–247. 10. Kolle G, Shepherd JL, Gardiner B, et al. Deep-transcriptome and ribonome sequencing redefines the molecular networks of pluripotency and the extracellular space in human embryonic stem cells . Genome Res. 2011; ; 21 : :2014.–2025. |