| |
| Med Sci (Paris). 2012 May; 28(5): 490–496. Published online 2012 May 30. doi: 10.1051/medsci/2012285013.Petits ARN non codants et spermatogenèse Yannick Romero,1# Pierre Calvel,1# and Serge Nef1* 1Département de médecine génétique et développement, faculté de médecine, université de Genève, 1, rue Michel-Servet, 1211Genève 4, Suisse |
La spermatogenèse est le processus biologique par lequel des cellules souches diploïdes, les spermatogonies, se différencient en gamètes haploïdes, les spermatozoïdes. Ce processus complexe est divisé en trois phases. Au cours de la 1re phase, dite mitotique, les spermatogonies se renouvellent et prolifèrent activement avant leur entrée dans la 2e phase de méiose. Cette seconde phase permet le brassage aléatoire de l’information génétique (processus de recombinaison) dans les spermatocytes, ce qui produit des cellules haploïdes génétiquement uniques, les spermatides rondes. Finalement, la 3e phase de spermiogenèse permet aux spermatides rondes de se différencier en spermatozoïdes, qui seront ensuite libérés dans le fluide testiculaire au cours de la spermiation. L’entièreté de ce processus est sous le contrôle des cellules de Sertoli qui forment avec les cellules germinales un ensemble cellulaire extrêmement intriqué et complexe, l’épithélium séminifère (Figure 1). Par leurs fonctions nourricières de support et de protection contre les pathogènes, les cellules de Sertoli permettent un développement approprié des cellules germinales au cours de la spermatogenèse. L’une des singularités de la spermatogenèse est que les étapes qui la composent s’effectuent sous l’action de multiples facteurs protéiques souvent exprimés de manière alternée et transitoire. Ces schémas d’expressions complexes, qui se déroulent au sein d’un même lignage cellulaire, impliquent ainsi l’existence de régulations transcriptionnelles ou traductionnelles permettant l’enchaînement approprié d’événements cellulaires majeurs tels que la recombinaison homologue, la condensation nucléaire ou la formation du flagelle.
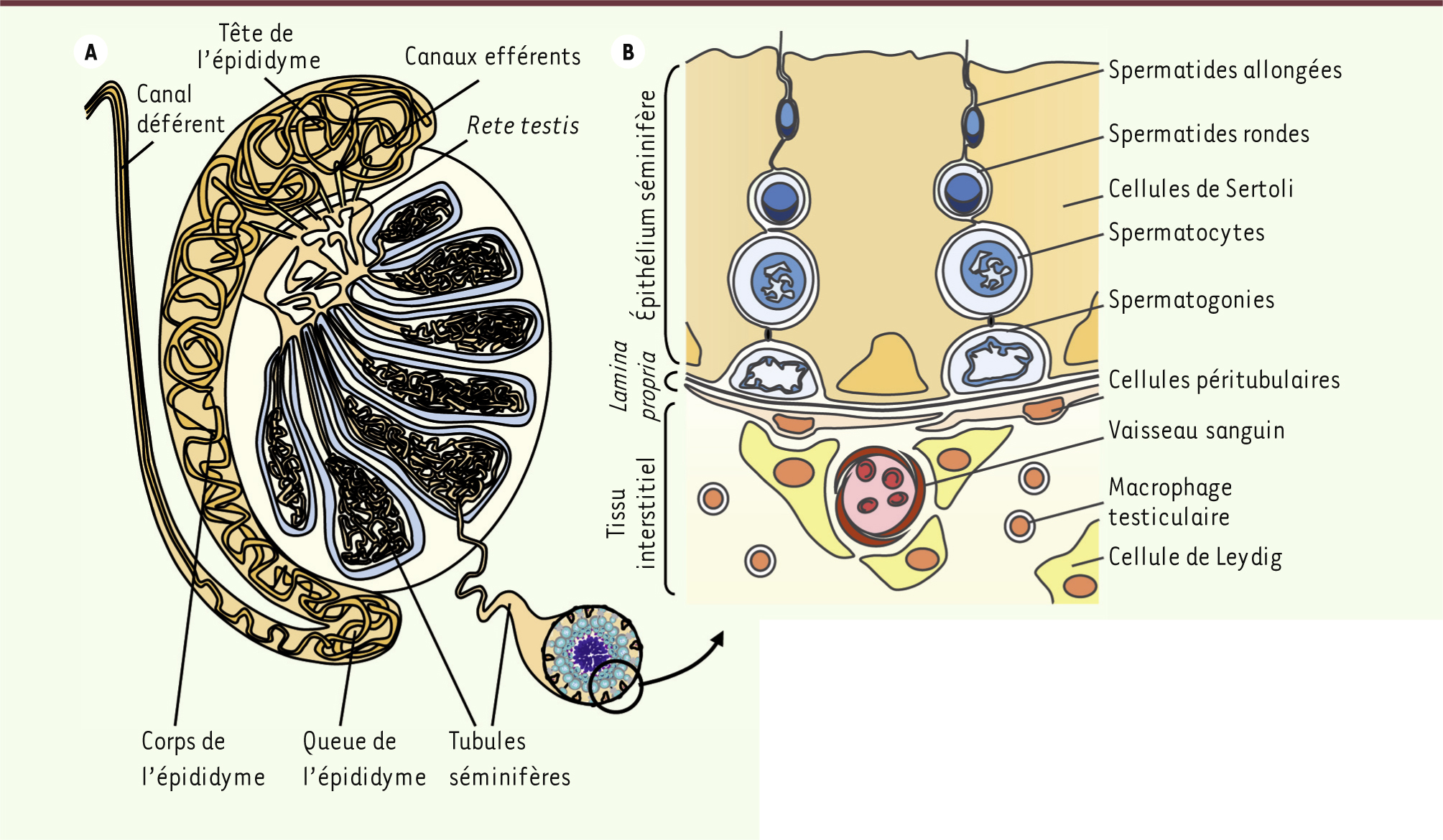
 | Figure 1.
Structure du testicule et de l’épithélium séminifère. A. Description schématique générale du testicule, de l’épididyme et du canal déférent. B. Description de l’organisation de l’épithélium séminifère et des relations entre les cellules de Sertoli et les cellules germinales. |
Dans ce contexte, un nouvel échelon de régulations post-transcriptionnelles a été récemment identifié, en l’objet des petits ARN non codants. Le but de cette revue est non seulement de décrire les différentes classes de petits ARN non codants présents dans les cellules germinales et somatiques du testicule, mais également de détailler leur mode d’action et leur impact sur la spermatogenèse et la fonction testiculaire en général. |
Classification et description des petits ARN non codants Les ARN non codants, comme leur nom l’indique, sont des transcrits qui ne codent pas pour une protéine. La famille des ARN non codants comprend historiquement les ARN de transfert (ARNt), les ARN ribosomiques (ARNr), les petits ARN nucléaires (snRNA pour small nuclear RNA) et les petits ARN nucléolaires (snoRNA pour small nucleolar RNA), chacune de ces catégories s’avérant essentielle au processus de traduction dans toutes les cellules d’un organisme donné. La famille des ARN non codants s’est agrandie depuis une quinzaine d’années avec l’identification progressive de nouvelles classes d’ARN non codants de taille inférieure à 40 nucléotides, telles que les microARN (miARN), les ARN interagissant avec la protéine PIWI (P-element induced wimpy testis piARN pour PIWI-interacting RNA) et les petits ARN interférents endogènes (endo-siARN). Ces petits ARN non codants, qui font l’objet de cette revue, remplissent de multiples fonctions dont celles de l’inhibition post-transcriptionnelle des gènes et de la répression des rétrotransposons. Ils modulent des processus biologiques essentiels à un organisme tels que la prolifération, la différenciation et l’apoptose. Une dérégulation de l’expression des miARN est ainsi souvent à la source de multiples pathologies telles que les cancers [1], le diabète [2] ou les troubles mentaux [3]. |
Les microARN : biogenèse, mode d’action et expression testiculaire Les miARN (ou miR) sont des petits ARN non codants de 20 à 22 nucléotides transcrits à partir du génome nucléaire, et qui régulent l’expression de gènes en se liant à la région 3’UTR (untranslated region) d’ARN messagers cibles. Les miARN sont conservés au cours de l’évolution, et leurs séquences génomiques correspondantes représentent environ 1 à 2 % du génome (pour revue voir [4]). En 2011, la dernière version de miRBase répertorie 1 492 miARN chez l’humain, 782 chez la souris et 417 chez le rat1. Chez les mammifères, les séquences qui transcrivent les miARN sont principalement localisées dans les introns ou plus rarement dans les exons de gènes codant pour une protéine. Dans cette configuration, les miARN s’expriment sous le contrôle du promoteur du gène codant pour les protéines - dans lequel ils se trouvent - et sont générés à partir du préARN messager, ce qui suggère une coordination entre l’expression des miARN et l’expression de l’ARN messager dans lequel ils résident [5]. Dans le cas d’une localisation intergénique, le gène du miARN possède son propre promoteur et s’exprime de manière indépendante. Les miARN sont transcrits sous forme de précurseurs, puis clivés successivement par plusieurs RNases telles que DROSHA et DICER1 pour donner naissance à des miARN matures simple brin de 20 à 22 nucléotides (Figure 2A) [35]. Ces miARN matures s’associent à un complexe protéique appelé RISC (RNA induced silencing complex) et agissent au niveau post-transcriptionnel comme régulateurs négatifs de l’expression, en affectant la traduction et/ou en promouvant la dégradation des ARNm cibles (pour revue voir [6, 7]).
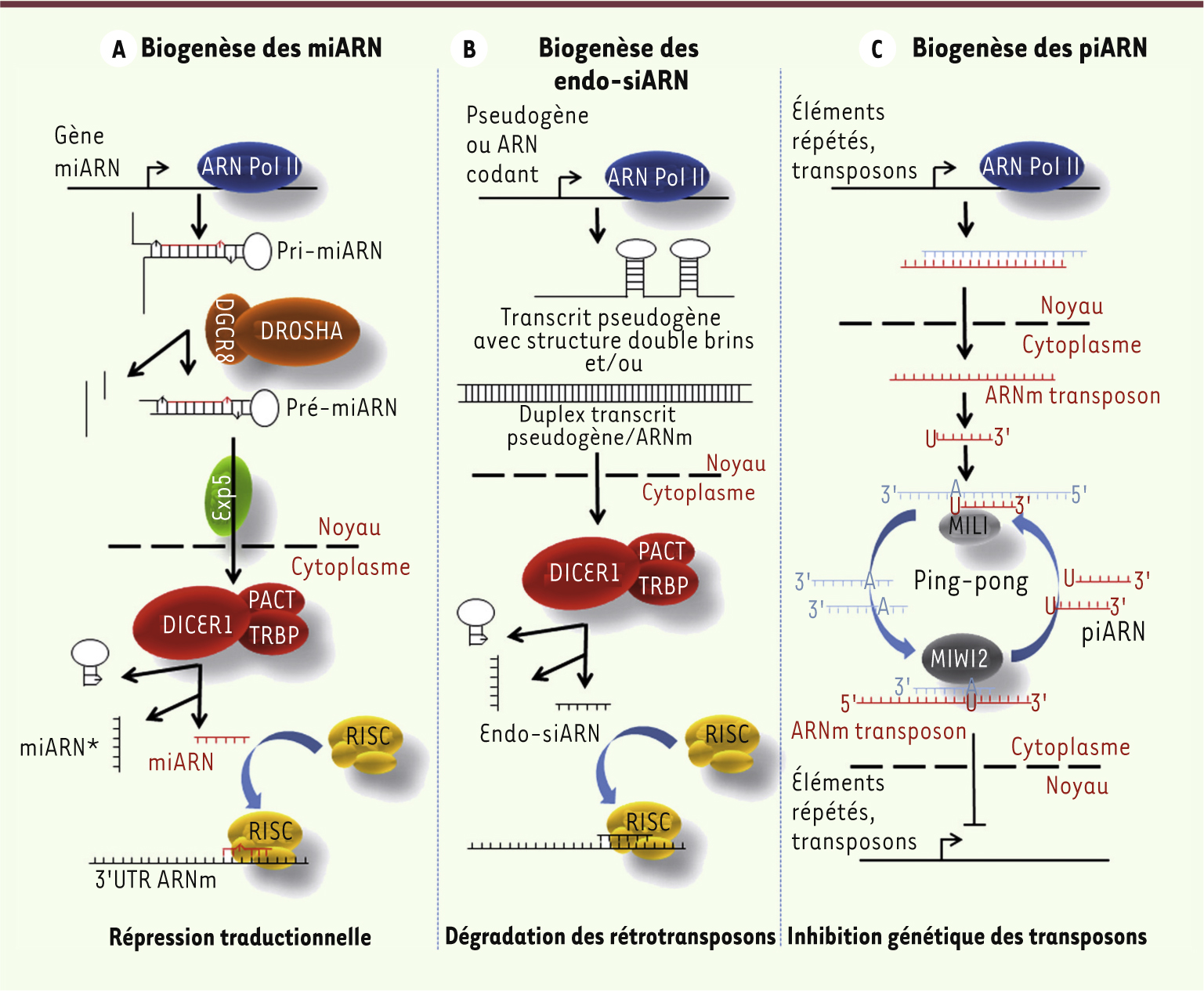
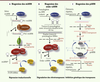 | Figure 2.
Voies de biogenèse des petits ARN non codants. A. Biogenèse des microARN. Chez les mammifères, les miARN sont transcrits en un long ARN primaire (pri-miARN) dont la taille peut varier d’une centaine à plusieurs milliers de nucléotides. Ces structures sont ensuite clivées par un complexe enzymatique composé d’une RNase de type III DROSHA et d’une protéine liant l’ARN double brin DGCR8 qui donne naissance à un miARN précurseur (pré-miARN) [ 33]. À ce stade, les pré-miARN sont exportés dans le cytoplasme par l’exportine-5 (Exp5) puis clivés par DICER1, une autre RNase de type III, pour donner naissance à un duplex miARN/miARN* de 20 à 22 nucléotides, en association avec les protéines TRBP ( TAR RNA binding protein) et PACT ( PKR activating protein) nécessaires à l’assemblage de ce complexe [ 34]. Les brins du duplex sont ensuite déroulés et le microARN mature simple brin s’associe à un complexe protéique appelé RISC ( RNA induced silencing complex) composé entre autres de protéines de la famille Argonaute (Ago). Ce complexe RISC chargé du miRNA mature « scanne » puis s’hybride de manière imparfaite avec la région 3’UTR d’un ARN messager cible, et agit au niveau post-transcriptionnel comme régulateur négatif de l’expression. Cette régulation post-transcriptionnelle s’effectue au stade de la synthèse protéique en affectant la traduction (pour revue voir [ 6]) et/ou en promouvant la dégradation des ARNm cibles [ 7]. B. Biogenèse des endo-siARN. Les précurseurs des siARN endogènes sont des transcrits composés de longues structures en tête d’épingle ou d’ARN double brin dérivés de transcrits hybrides sens/anti-sens provenant du même locus (cis-endo-siARN) ou de deux locus différents (trans-endo-siARN). À partir de ces précurseurs sont produits les petits endo-siARN, un processus dépendant du clivage par DICER1 mais pas de DGCR8 [ 12]. C. Biogenèse des piARN. Les piARN prépachytènes sont générés grâce au cycle « ping-pong » qui requiert un réservoir de piARN primaires (en rouge) déjà présents dans la cellule, dont la genèse reste encore un mécanisme inconnu. Ils sont ensuite chargés sur la protéine MILI qui va produire un piARN secondaire (en bleu), clivé ensuite par le complexe MIWI2 pour donner naissance aux piARN terminaux impliqués dans la régulation et la dégradation d’éléments transposables délétères (pour revue voir [ 8]). |
De multiples prédictions bio-informatiques suggèrent que les miARN modulent l’expression de près de 60 % des gènes de mammifères (pour revue voir [4]). Ceci n’est en soi pas vraiment surprenant puisque chaque miARN régule simultanément l’expression de plusieurs ARN messagers. Inversement, un ARN messager peut être la cible de multiples miARN. La présence de petits ARN non codants (miARN, endo-siARN et piARN) ainsi que celle des protéines nécessaires à leur biogenèse et fonction (incluant DROSHA, DICER1, AGO2 [argonaute 2 ou EIF2C2], PIWIL1 [piwi-like homolog 1 chez l’homme]/MIWI, PIWIL4/MIWI2 et PIWIL2/MILI) ont été décrites dans les différentes sous-populations de cellules germinales, telles que les spermatocytes pachytènes, les spermatides rondes et allongées et les cellules de Sertoli [8, 9]. Jusqu’à présent, le nombre de miARN identifiés dans le testicule est évalué à 141. Parmi ceux-ci, 35 % sont préférentiellement exprimés dans le testicule tandis que 5 % y sont exclusivement présents [10]. Récemment, le décryptage de l’ensemble des miARN exprimés dans les cellules germinales à l’aide de techniques de séquençage à grande échelle a permis d’identifier de nouveaux miARN spécifiquement exprimés dans le testicule prépubère [11]. Il est clair que l’émergence du séquençage à haut débit permettra, dans un futur proche, d’identifier l’ensemble des petits ARN non codants (dont les miARN) exprimés dans les différentes populations germinales et somatiques du testicule. |
siARN endogènes : récents acteurs du contrôle post-transcriptionnel Les siARN endogènes (endo-siARN) mammifères sont des petits ARN non codants de 21 nucléotides, transcrits à partir d’éléments transposables et/ou de pseudogènes (Figure 2B). Cette nouvelle classe de petits ARN non codants a été décrite dans les ovocytes [12], les cellules souches embryonnaires [13] et récemment dans les cellules germinales mâles [9]. Les données détaillant le rôle et l’importance de ces endo-siARN dans la régulation de la spermatogenèse sont toujours parcellaires, même si une étude récente a montré l’expression d’au moins 73 endo-siARN dans le testicule de souris, dont certains sont capables d’induire la dégradation d’ARNm cibles in vitro [9]. |
piARN : gardiens de l’intégrité génétique de la lignée germinale mâle Environ 30 à 40 % du génome des mammifères sont composés d’éléments transposables [14]. Différents mécanismes existent dans les cellules germinales pour contrôler l’expression de ces rétrotransposons. Ils incluent notamment la répression de la transcription par méthylation de l’ADN [15] et la répression par ARN interférence [16]. Dans ce cadre, une autre classe de petits ARN non codants de 26 à 32 nucléotides de longueur a été découverte il y a quelques années dans la lignée germinale [17, 18]. Ces petits ARN, appelés piARN, s’associent avec les protéines PIWI pour former des complexes ribonucléoprotéiques impliqués notamment dans la répression des rétrotransposons au sein de la lignée germinale mâle. Concrètement, il existe deux sous-populations de piARN produites dans les cellules germinales mâles, indépendamment de DICER1 et DGCR8 (DiGeorge syndrome critical region gene 8) : les piARN prépachytènes et les piARN pachytènes. Les piARN prépachytènes sont transcrits dans les prospermatogonies pendant l’embryogenèse à partir d’éléments transposables (Figure 2C). Quant aux piARN pachytènes, ils proviennent de longs regroupements génomiques de piARN dont le processus de production est encore inconnu. Ces derniers sont supposés être chargés sur le complexe MILI/MIWI, mais leur fonction à la fin de la méiose et durant la spermiogenèse n’est pas encore connue (pour revue voir [8]). À noter que le nombre de piRNA est particulièrement impressionnant puisqu’il a été évalué à environ 40 000 selon la base de données piRNAbank2. Hormis leur action régulatrice sur l’expression des éléments transposables du génome, le rôle des piARN durant la spermatogenèse chez le mammifère n’a pas encore été directement révélé. Des études menées in vivo sur les conséquences de mutations des gènes de la voie de biosynthèse des piARN confirment cependant leur importance : on observe par exemple chez les souris mutantes Mili ou Miwi2 un arrêt méiotique de la spermatogenèse, ainsi qu’une forte augmentation de l’expression des rétrotransposons. D’autre part, les spermatides rondes dans les testicules de souris mutantes pour le gène Miwi sont incapables de se différencier en spermatozoïdes [19]. |
Étude fonctionnelle des petits ARN non codants lors de la spermatogenèse Le premier exemple de régulation traductionnelle par les miARN dans les cellules germinales a été publié il y a quelques années. Il concerne l’action de miR-122a sur l’ARNm de la protéine de transition 2 (TNP2), l’un des effecteurs clefs de la condensation chromatinienne lors de la spermiogenèse [20]. Plus récemment, l’équipe de Brinster a pu démontrer que miR-21, un miARN fortement exprimé dans les cellules souches spermatogoniales, était impliqué dans la régulation de l’expression de ETV5 (ets variant gene 5), un facteur de transcription essentiel au maintien de la niche spermatogoniale via le facteur de croissance GDNF (glial cell derived neurotrophic factor) [21]. Par ailleurs, une étude très récente portant sur miR-17-92 prouve le rôle de ce miARN dans le contrôle de la différenciation des spermatogonies, puisqu’il interviendrait dans l’inhibition de gènes proméiotiques tels que le proto-oncogène c-kit, ou Stat3 (signal transducer and activator of transcription 3), cette répression étant levée sous l’effet de l’acide rétinoïque [22]. Enfin, d’autres miARN tels que miR-469 [23] ou miR-184 [24] ont également fait l’objet d’études décrivant leurs rôles cruciaux lors des différentes étapes de la spermatogenèse. Hormis leur importance lors du déroulement de la spermatogenèse normale, les miARN pourraient être impliqués dans certains aspects pathologiques de la spermatogenèse. Novotny et al. ont ainsi montré en comparant des banques d’expression de testicules normaux et cancéreux, que l’ARNm du facteur de transcription E2F1, un facteur à la fois pro-oncogénique et suppresseur de tumeur, était régulé au niveau traductionnel par miR-17-92[25]. D’autres miARN tels que miR-322 et miR-323 ont été proposés comme favorisant la tumorigenèse des cellules germinales, se comportant donc comme des oncogènes potentiels [26]. Par ailleurs, des éléments ayant trait à la localisation des miARN dans le génome permettent également de nouvelles hypothèses sur le rôle de ces derniers lors de la spermatogenèse. Une étude a ainsi rapporté que 86 % des miARN présents sur le chromosome X échappent à son inactivation lors de la méiose. Ceci laisse supposer que les miARN pourraient jouer un rôle dans l’inactivation elle-même, ou qu’ils sont essentiels au contrôle post-transcriptionnel des ARNm autosomiques durant la phase tardive de la méiose et la phase postméiotique précoce [27]. Globalement, ces quelques données montrent que les régulations post-transcriptionnelles et traductionnelles opérées via les miARN interviennent à des étapes de la spermatogenèse aussi importantes que l’autorenouvellement et la prolifération des spermatogonies, le maintien des cellules méiotiques ou la condensation du noyau dans les spermatides (pour revue [28]). Toutefois, le nombre important des petits ARN non codants dans les génomes de mammifères et leurs modes d’action extrêmement pléiotropes sont incompatibles avec une étude fonctionnelle classique, gène par gène. Disséquer précisément les réseaux régulateurs impliquant ces petits ARN est donc une tâche ardue, car elle doit tenir compte de leur nombre comme de la multiplicité des régulations qu’ils exercent. De ce fait, il n’est pas surprenant que l’invalidation individuelle de certains de ces petits ARN chez la souris se soit révélée relativement peu informative, car elle se traduit souvent par une absence de phénotype. Une autre approche plus globale, et aussi plus fructueuse, consiste à invalider les gènes codant pour les protéines nécessaires à la biosynthèse des petits ARN non codants comme DROSHA, DGCR8 ou DICER1, en jouant sur le niveau d’implication de chacun dans cette biosynthèse. Par exemple, les inactivations de DROSHA ou DGCR8 permettent d’étudier les conséquences de la seule perte des miARN dans un type cellulaire donné, tandis que l’absence de DICER1 permet d’évaluer les effets de l’absence cumulative des miARN et des endo-siARN. |
DICER1, petits ARN non codants et cellules de Sertoli Le rôle des miARN et des endo-siARN durant le développement testiculaire et la spermatogenèse chez les mammifères est étudié depuis quelques années grâce au système Cre-lox, qui permet la délétion spécifique in vivo de gènes par l’emploi d’une recombinase Cre couplée à un promoteur transcriptionnel actif dans un type cellulaire donné. Par exemple, l’ablation de Dicer1 dans les cellules de Sertoli immatures grâce à une recombinase couplée au promoteur de l’AMH (anti-mullerian hormone) entraîne des défauts majeurs dans le déroulement de la spermatogenèse (Figure 3A–C). En effet, cette délétion de Dicer1 affecte drastiquement la survie et la fonction de soutien des cellules de Sertoli, puisque les souris mutantes sont stériles et que le nombre des cellules de Sertoli et des cellules germinales est dramatiquement réduit [29, 30]. Sur un plan moléculaire, plusieurs gènes essentiels au maintien de la niche spermatogoniale et à l’engagement de la spermatogenèse tels que Gdnf, Kitl(codant pour le ligand de c-kit, ou stem cell factor), Man2a2 (mannosidase, alpha, class 2A), Gata1 (globin transcription factor 1), Dhh (desert hedgehog), SerpinA5 (ou protein C inhibitor), Wt1 (Wilms tumor 1), et Sox9 ([sex determining region y]-box 9) sont sous exprimés par les cellules de Sertoli mutantes en comparaison des cellules contrôles [29, 30].
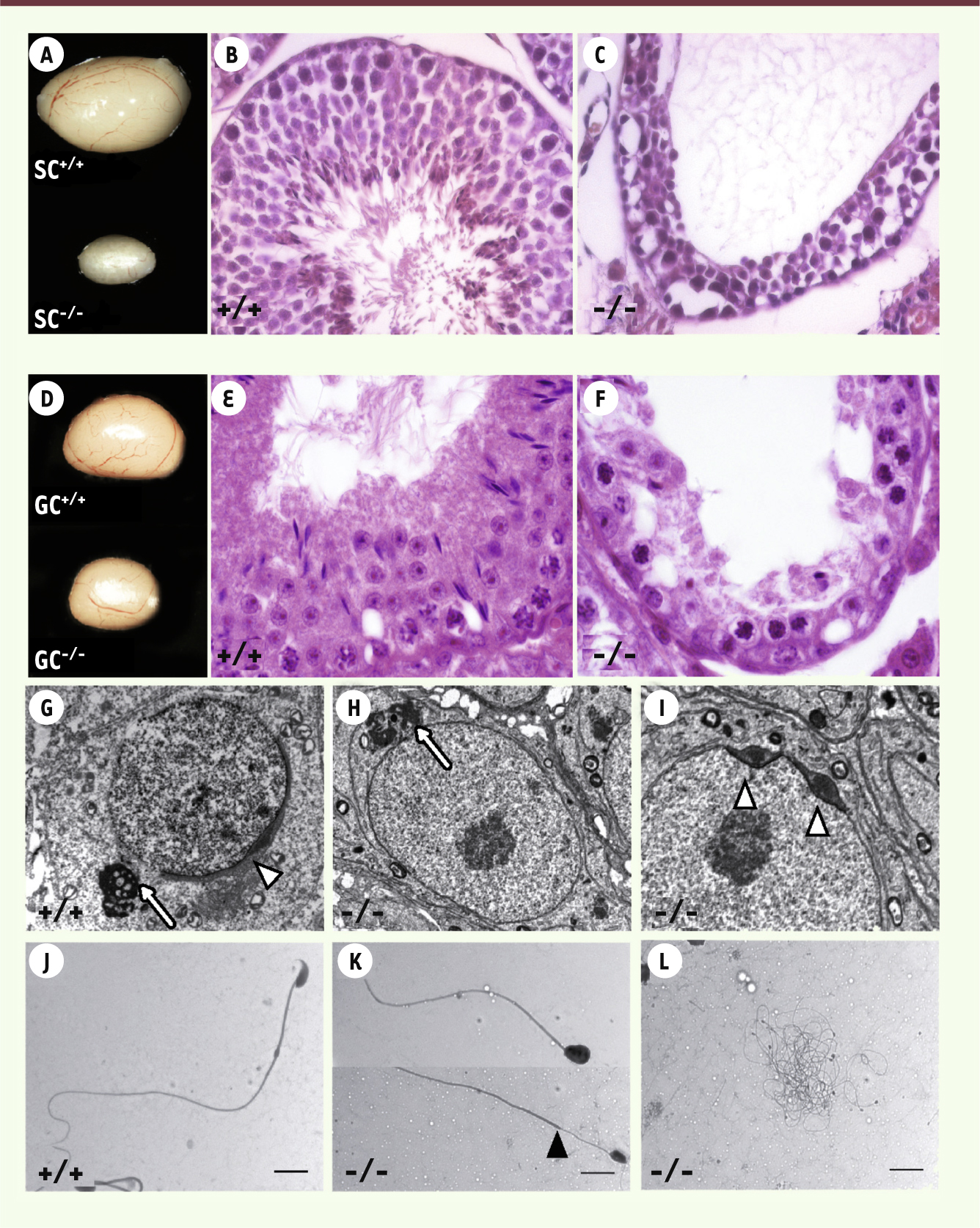
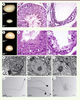 | Figure 3.
Infertilité complète due à l’invalidation génétique de Dicer1 dans la lignée germinale ou les cellules de Sertoli chez la souris. L’invalidation génétique de Dicer1 dans les cellules de Sertoli (SC, A-C) et les cellules germinales (GC, D-L) entraîne une réduction de la taille globale du testicule (A et D) et une absence quasi complète de spermatozoïdes matures et de spermatides allongées (comparer B avec C et E avec F). Des images de microscopie électronique de spermatides rondes contrôles (GC +/+, G) et mutantes (GC -/-, H et I) indiquent que le corps chromatoïde est normalement formé et localisé à proximité du noyau (flèche en H), alors que l’acrosome est fragmenté chez les mutants (têtes de flèches en G et I). Contrairement aux contrôles (J), les spermatozoïdes mutants (K et L) exhibent de multiples défauts touchant la morphologie de la tête, la pièce intermédiaire (K) et une réduction de la taille globale pour certains (L). La tête de flèche noire (K) indique la localisation ectopique de la pièce intermédiaire. Échelle en J et L : 10 µm (modifié selon [ 30, 32]). |
|
DICER1, petits ARN non codants et cellules germinales Afin de clarifier le rôle des miARN et des endo-siARN lors des différentes étapes du développement des cellules germinales, plusieurs modèles transgéniques murins ont été créés afin d’induire une délétion spécifique de Dicer1 dans les spermatogonies [31, 32]. Le premier modèle implique une recombinase Cre sous le contrôle du promoteur de l’hélicase MVH (mouse VASA homolog), actif dans les cellules germinales dès la naissance (Ddx4-Cre), tandis que le second modèle met en jeu le promoteur de la neurogénine 3, actif 5 à 7 jours post-partum (Ngn3-Cre). Dans les deux cas, les résultats indiquent que Dicer1 est absolument requis pour une spermatogenèse adulte normale puisque sa déplétion entraîne une accumulation de défauts au cours de la méiose et de la spermiogenèse conduisant à une absence de spermatozoïdes fonctionnels et à une infertilité complète (Figure 3D–L). De manière intéressante, la comparaison des deux lignées de souris mutantes révèle une gradation dans la sévérité du phénotype reproducteur, puisque la méiose de la première vague de spermatogenèse semble plus sévèrement atteinte dans les souris où l’inactivation de Dicer1 est précoce. Cette différence due au décalage dans l’expression de ces deux recombinases Cre suggère qu’un certain nombre de miARN produits et stockés dans les gonocytes et les spermatogonies pourraient jouer un rôle plus tardif lors de l’initiation et la progression de la méiose. Ces résultats démontrent ainsi que la biogenèse des endo-siARN et des miARN est essentielle au développement des cellules germinales mâles, particulièrement lors des phases méiotique et postméiotique. Toutefois, le degré de contribution de ces deux classes de petits ARN, endo-siARN et miARN, reste à évaluer. Une étude récente donne un premier élément de réponse : dans des souris mutantes pour Drosha - l’endonucléase requise pour la biosynthèse des miARN mais pas des endo-siARN -, le phénotype testiculaire observé, bien que similaire à celui des animaux mutants pour Dicer1, s’avère nettement moins sévère, ce qui confirme l’importance des endo-siARN dans la régulation de la spermatogenèse [9]. |
Perspectives et conclusion Malgré l’accumulation de données expérimentales et l’émergence de modèles fonctionnels, notre compréhension des rôles complexes joués par les différentes classes de petits ARN non codants lors de la spermatogenèse reste toujours partielle. Les développements récents du séquençage à haut débit ont permis d’identifier une pléthore de petits ARN non codants dans les lignées germinales et somatiques. De même, l’analyse des mutants de la voie de biogenèse de ces petits ARN chez la souris a souligné leur importance à de multiples étapes de la spermatogenèse. Il reste toutefois un grand pas à franchir en parvenant à assembler ces données en systèmes de régulation intégrés, censés mieux représenter l’extrême complexité des interactions existant entre les petits ARN non codants et leurs cibles. Cette approche holistique se fera, d’une part, grâce aux outils prédictifs et de modélisation proposés par les progrès de la bioinformatique et, d’autre part, grâce aux récentes améliorations dans le criblage fonctionnel à haut débit des petits ARN [4]. Cette stratégie pourrait se révéler redoutablement efficace pour caractériser les mécanismes moléculaires impliquant ces petits ARN dans la biologie de la reproduction. La résultante serait une meilleure compréhension de la fonction testiculaire et pourrait constituer un nouveau champ d’investigation afin de déchiffrer les aspects cliniques et l’étiologie de l’infertilité masculine humaine. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1. Myatt SS, Wang J, Monteiro LJ, et al. Definition of microRNAs that repress expression of the tumor suppressor gene FOXO1 in endometrial cancer . Cancer Res. 2010; ; 70 : :367.–377. 2. Pandey AK, Agarwal P, Kaur K, Datta M. MicroRNAs in diabetes: tiny players in big disease . Cell Physiol Biochem. 2009; ; 23 : :221.–232. 3. Xu Y, Li F, Zhang B, et al. MicroRNAs and target site screening reveals a pre-microRNA-30e variant associated with schizophrenia . Schizophr Res. 2010; ; 119 : :219.–227. 4. Serva A, Claas C, Starkuviene V. A potential of microRNAs for high-content screening . J Nucleic Acids. 2011; ; 2011 : :870903.. 5. Saini HK, Griffiths-Jones S, Enright AJ. Genomic analysis of human microRNA transcripts . Proc Natl Acad Sci USA. 2007; ; 104 : :17719.–17724. 6. Filipowicz W, Bhattacharyya SN, Sonenberg N. Mechanisms of post-transcriptional regulation by microRNAs : are the answers in sight ? Nat Rev Genet. 2008; ; 9 : :102.–114. 7. Guo H, Ingolia NT, Weissman JS, Bartel DP. Mammalian microRNAs predominantly act to decrease target mRNA levels . Nature. 2010; ; 466 : :835.–840. 8. Meikar O, Da Ros M, Korhonen H, Kotaja N. Chromatoid body and small RNAs in male germ cells . Reproduction. 2011; ; 142 : :195.–209. 9. Song R, Hennig GW, Wu Q, et al. Male germ cells express abundant endogenous siRNAs . Proc Natl Acad Sci USA. 2011; ; 108 : :13159.–13164. 10. Ro S, Park C, Sanders KM, et al. Cloning and expression profiling of testis-expressed microRNAs . Dev Biol. 2007; ; 311 : :592.–602. 11. Buchold GM, Coarfa C, Kim J, et al. Analysis of microRNA expression in the prepubertal testis . PLoS One. 2010; ; 5 : :e15317.. 12. Watanabe T, Totoki Y, Toyoda A, et al. Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes . Nature. 2008; ; 453 : :539.–543. 13. Babiarz JE, Ruby JG, Wang Y, et al. Mouse ES cells express endogenous shRNAs, siRNAs, and other microprocessor-independent, Dicer-dependent small RNAs . Genes Dev. 2008; ; 22 : :2773.–2785. 14. Mandal PK, Kazazian HH Jr. SnapShot: vertebrate transposons . Cell. 2008; ; 135 : :192.–e1. 15. Bourc’his D, Bestor TH. Meiotic catastrophe and retrotransposon reactivation in male germ cells lacking Dnmt3L . Nature. 2004; ; 431 : :96.–99. 16. Siomi MC, Kuramochi-Miyagawa S. RNA silencing in germlines: exquisite collaboration of Argonaute proteins with small RNAs for germline survival . Curr Opin Cell Biol. 2009; ; 21 : :426.–434. 17. Vagin VV, Sigova A, Li C, et al. A distinct small RNA pathway silences selfish genetic elements in the germline . Science. 2006; ; 313 : :320.–324. 18. Aravin A, Gaidatzis D, Pfeffer S, et al. A novel class of small RNAs bind to MILI protein in mouse testes . Nature. 2006; ; 442 : :203.–207. 19. Deng W, Lin H. Miwi, a murine homolog of piwi, encodes a cytoplasmic protein essential for spermatogenesis . Dev Cell. 2002; ; 2 : :819.–830. 20. Chen C, Ouyang W, Grigura V, et al. ERM is required for transcriptional control of the spermatogonial stem cell niche . Nature. 2005; ; 436 : :1030.–1034. 21. Niu Z, Goodyear SM, Rao S, et al. MicroRNA-21 regulates the self-renewal of mouse spermatogonial stem cells . Proc Natl Acad Sci USA. 2011; ; 108 : :12740.–12745. 22. Tong MH, Mitchell DA, McGowan SD, et al. Two miRNAs clusters, Mir-17–92 (Mirc1) and Mir-106b–25 (Mirc3), are involved in the regulation of spermatogonial differentiation in mice . Biol Reprod. 2011; ; 86 : :72.. 23. Dai L, Tsai-Morris CH, Sato H, et al. Testis-specific miRNA-469 up-regulated in gonadotropin-regulated testicular RNA helicase (GRTH/DDX25)-null mice silences transition protein 2 and protamine 2 messages at sites within coding region : implications of its role in germ cell develpoment . J Biol Chem. 2011; ; 286 : :44306.–44318. 24. Wu J, Bao J, Wang L, et al. MicroRNA-184 downregulates nuclear receptor corepressor 2 in mouse spermatogenesis . BMC Dev Biol. 2011; ; 11 : :64.. 25. Novotny GW, Sonne SB, Nielsen JE, et al. Translational repression of E2F1 mRNA in carcinoma in situ and normal testis correlates with expression of the miR-17–92 cluster . Cell Death Differ. 2007; ; 14 : :879.–882. 26. Voorhoeve PM, le Sage C, Schrier M, et al. A genetic screen implicates miRNA-372 and miRNA-373 as oncogenes in testicular germ cell tumors . Cell. 2006; ; 124 : :1169.–1181. 27. Song R, Ro S, Michaels JD, et al. Many X-linked microRNAs escape meiotic sex chromosome inactivation . Nat Genet. 2009; ; 41 : :488.–493. 28. McIver SC, Roman SD, Nixon B, McLaughlin EA. miRNA and mammalian male germ cells . Hum Reprod Update. 2012; ; 18 : :44.–59. 29. Papaioannou MD, Lagarrigue M, Vejnar CE, et al. Loss of Dicer in Sertoli cells has a major impact on the testicular proteome of mice . Mol Cell Proteomics. 2011; ; 10 : :M900587MCP200.. 30. Papaioannou MD, Pitetti JL, Ro S, et al. Sertoli cell Dicer is essential for spermatogenesis in mice . Dev Biol. 2009; ; 326 : :250.–259. 31. Korhonen HM, Meikar O, Yadav RP, et al. Dicer is required for haploid male germ cell differentiation in mice . PLoS One. 2011; ; 6 : :e24821.. 32. Romero Y, Meikar O, Papaioannou MD, et al. Dicer1 depletion in male germ cells leads to infertility due to cumulative meiotic and spermiogenic defects . PLoS One. 2011; ; 6 : :e25241.. 33. Han J, Lee Y, Yeom KH, et al. Molecular basis for the recognition of primary microRNAs by the Drosha-DGCR8 complex . Cell. 2006; ; 125 : :887.–901. 34. Kok KH, Ng MH, Ching YP, Jin DY. Human TRBP and PACT directly interact with each other and associate with dicer to facilitate the production of small interfering RNA . J Biol Chem. 2007; ; 282 : :17649.–17657. 35. Dunoyer P. La bataille du silence : mécanisme et inhibition du RNA silencing au cours des interactions plante/virus . Med Sci (Paris). 2009; ; 25 : :505.–511. |