| |
| Med Sci (Paris). 2012 May; 28(5): 526–530. Published online 2012 May 30. doi: 10.1051/medsci/2012285018.Le modèle de l’inactivation du chromosome X chez la souris Lucie Delaroche,1,2,3** Pauline Demailly,1,2 Katia Ancelin,3 and Catherine Patrat1,2,3* 1Faculté de médecine, université Paris Diderot, Paris, France 2Service de biologie de la reproduction, Hôpital Bichat, Assistance publique-hôpitaux de Paris (AP-HP), 46, rue Henri Huchard, 75018Paris, France 3Equipe épigenèse et développement des mammifères, unité de génétique et biologie du développement, Inserm U934 CNRS UMR 3215, Institut Curie, Paris, France |
L’inactivation du chromosome X (ICX) constitue un modèle intéressant pour l’étude des modifications épigénétiques intervenant après la fécondation, au cours du développement préimplantatoire chez les mammifères. Ce mécanisme dit de « compensation de dose » permet d’égaliser la quantité de produits codés par les gènes du chromosome X chez la femelle XX et chez le mâle XY, via l’inactivation d’un des deux chromosomes X dans l’embryon femelle [
1]. Un des deux chromosomes X passe, durant le développement embryonnaire précoce, d’un état euchromatique actif à un état hétérochromatique inactif, connu sous le nom de corpuscule de Barr. La mise en place de ce mécanisme a été particulièrement bien étudiée chez la souris où le processus d’inactivation du chromosome X se divise en deux phases au cours du développement embryonnaire. La première phase dite « soumise à empreinte » a lieu au cours du développement embryonnaire préimplantatoire et aboutit à l’inactivation du chromosome X d’origine paternelle (Xp) exclusivement. Cette inactivation est maintenue dans les cellules extraembryonnaires. Dans cette revue, nous nous intéresserons en particulier aux mécanismes moléculaires mis en place durant cette étape. Puis, nous aborderons plus brièvement la deuxième phase d’inactivation dite « inactivation aléatoire » qui aboutit à l’inactivation, soit du Xp, soit du chromosome X d’origine maternelle (Xm). Cette seconde vague est mise en place dans les cellules de l’embryon propre, à l’exclusion des cellules des annexes extraembryonnaires [1–
3]. |
Inactivation du chromosome X soumise à empreinte Initiation de l’inactivation de l’X après la fécondation Le déclenchement de l’inactivation de l’X est contrôlé par le centre d’inactivation du chromosome X (Xic) où se trouve le gène Xist. Au moment de l’activation du génome embryonnaire 1 au stade 2 cellules, seul l’allèle paternel de Xist est exprimé, et code pour un ARN non codant qui s’accumule en cis sur le chromosome X paternel à partir du stade 4 cellules. Les processus permettant à l’ARN Xist de s’accumuler en cis sur le chromosome X ne sont pas encore bien compris. L’organisation nucléaire, et notamment la présence des protéines se liant aux régions S/MAR ( scaffold/matrix attachment regions) de l’ADN comme hnRNPU ( heterogeneous nuclear ribonucleoprotein U), SAF-A ( scaffold attachment factor A) ou SATB1 ( special AT-rich binding protein 1) [
4], seraient impliquées dans ce processus. En réponse à l’accumulation de Xist en cis sur le Xp, une série de changements chromatiniens s’enchaîne [3]. Citons notamment la perte de l’acétylation de la lysine 9 (K9) et de la méthylation de la lysine 4 (K4) de l’histone H3 (H3) dès le stade 8 cellules, marques qui sont normalement associées à une chromatine dite permissive pour la transcription. Ensuite, au stade morula, les protéines du groupe Polycomb 2 (comme EED [embryonic ectoderm development] et EZH2 [enhancer of zeste homolog 2]) s’accumulent sur le Xp, entraînant l’hyperméthylation de la lysine 27 (K27me3) de l’histone H3. Ces changements sont associés à une chromatine réfractaire à l’expression génique. Plus tardivement, d’autres marques liées à la répression transcriptionnelle sont aussi ajoutées : par exemple, au stade blastocyste, la lysine 9 (K9) de l’histone H3 est méthylée et le variant d’histone macroH2A apparaît sur le chromosome Xp. Ainsi, au moment de l’implantation, au stade blastocyste, plus de 90 % des cellules du trophectoderme (extraembryonnaire) ont acquis de façon stable ces diverses marques hétérochromatiques, et les gènes du chromosome X paternel sont devenus silencieux dans leur grande majorité. Il semble également que le Xp est traité différemment dans ses régions géniques et non géniques (LINE et SINE2) au stade embryonnaire précoce [
5]. Au stade embryonnaire 2 cellules, le chromosome X paternel est transcriptionnellement actif. En effet, la majorité des gènes liés à l’X sont exprimés de façon biallélique [
6] au moment de l’activation du génome embryonnaire. À ce stade, les séquences répétées du Xp formeraient un compartiment silencieux, proche du nucléole, et ceci de façon indépendante de Xist. Ce compartiment silencieux serait caractérisé par l’exclusion de Cot13 et de l’ARN polymérase II, formant ainsi la première marque de transformation du Xp après la fécondation. On ne sait pas en revanche si les éléments répétés sont transmis déjà sous une forme inactive par le spermatozoïde, ou si leur silence s’installe après la fécondation mais précède le silence transcriptionnel génique. On peut imaginer que la contribution des éléments répétés serait de faciliter la progression de l’ARN Xist sur le Xp et ainsi l’extinction génique, comme cela a été démontré dans des modèles de cellules souches embryonnaires in vitro [
7]. Les premiers signes de silence transcriptionnel de novo sont observés dès le stade 4 cellules, par l’exclusion de l’ARN polymérase II au niveau du Xp et l’absence de transcrits primaires dès le stade 8 cellules [
8]. La fenêtre de début de l’inactivation varie cependant selon les gènes, allant du stade 8 cellules jusqu’au stade blastocyste [4]. Si la majorité des gènes sont silencieux à ce stade, certains échappent à ce processus, soit partiellement, soit totalement. Nature de la (ou des) marque(s) dictant le choix du chromosome X paternel au cours du développement murin Jusqu’à ce jour, la nature de l’empreinte parentale mise en jeu dans l’inactivation soumise à empreinte n’est pas connue, mais il semblerait qu’un mécanisme empêchant l’activation de l’allèle maternel de Xist et/ou une prédilection pour l’activation de l’allèle paternel de Xist soit mis en jeu. Résistance de l’allèle maternel à l’inactivation La létalité précoce d’embryons disomiques pour le X maternel (possédant deux Xm) ou héritant d’une délétion paternelle de Xist, probablement due à une déficience de compensation de dose, plaide pour une résistance du chromosome maternel à l’inactivation [
9–
11]. De même, des transgènes autosomiques portant la région Xic peuvent reproduire l’expression de Xist lorsqu’ils sont transmis à partir de la lignée germinale mâle alors qu’ils ne peuvent le faire lorsqu’ils sont transmis à partir de la lignée germinale femelle [ 8]. Cette empreinte maternelle, qui serait mise en place durant l’ovogenèse [
12], pourrait se situer au niveau du gène Xist ou agir par l’intermédiaire de Tsix, répresseur antisens de Xist de 40 kb environ [
13]. Elle est également indépendante de la méthylation de novo qui survient durant la croissance ovocytaire [
14]. Une autre hypothèse implique la protéine RNF12 (ring finger protein 12) : en effet, son absence dans des embryons femelles empêche toute mise en place de l’inactivation de l’X soumise à empreinte en raison d’un défaut d’expression de l’allèle paternel de Xist [
15]. RNF12 serait un élément déterminant pour l’expression de Xist en trans, même si le rôle exact de cette protéine n’est pas bien compris. De manière plus générale, un contenu riche en ARN et en protéines d’origine maternelle est hérité après la fécondation de l’ovocyte (voir note 1) et pourrait donc être impliqué, au moins pendant les premières divisions mitotiques, dans l’expression de l’allèle paternel Xist. En effet, des expériences de clonage somatique ont démontré que ce gène pouvait être activé à partir des deux copies d’une cellule d’origine maternelle ou bien à partir de la copie unique d’une cellule mâle (XY) [
16]. Ceci démontre que les activateurs de Xist sont présents par défaut dès la fécondation. Prédisposition de l’Xp à l’inactivation : état de l’X au cours de la spermatogenèse À l’inverse, le Xp pourrait être également prédisposé à l’inactivation du fait d’une marque ou d’une mémoire épigénétique associée à une constitution chromatinienne particulière qu’il porterait depuis son passage dans la lignée germinale mâle et qui ne serait exprimée qu’après la fécondation.
-
Inactivation des chromosomes X et Y dans le spermatocyte primaire
Au cours de la spermatogenèse, et plus précisément au stade pachytène du spermatocyte primaire, les chromosomes X et Y s’apparient grâce à leurs régions pseudoautosomiques pour former la vésicule sexuelle, ce qui engendre leur inactivation aussi appelée MSCI (meiotic sex chromosome inactivation). Ce processus est indépendant de Xist (Figure 1). On observe alors en hybridation in situ grâce à des sondes d’acides nucléiques fluorescentes (RNA-FISH) l’absence de signaux de Cot-1 (qui démontre l’absence de transcrits répétés naissants, voir note 3) [
17] et, en immunofluorescence, l’exclusion de la forme phosphorylée (active) de l’ARN polymérase II [17]. Ces deux observations montrent de façon concordante une absence de transcription de ces chromosomes, comme en témoigne aussi le silence transcriptionnel de la quasi totalité des 360 gènes liés à l’X étudiés en microarray [17]. On note aussi la présence de certaines modifications d’histones caractéristiques d’une chromatine transcriptionnellement inactive, comme l’hypoacétylation des histones H3 et H4 ou la méthylation sur H3K9. Cet état correspond à la formation de la vésicule sexuelle. Il semble cependant qu’une partie du chromosome X échappe à cette inactivation puisque seulement 10 des 77 microARN liés à l’X étudiés sont soumis à la MSCI [
18]. Ces régions qui échappent à l’inactivation transcriptionnelle coïncident probablement avec la persistance de régions actives du génome, comme en atteste la mise en évidence de la forme active de la lysine 4 de l’histone 3 (H3K4me3) dans les spermatocytes aux stades pachytène et diplotène, l’acétylation des histones H3 et H4, la sous-méthylation des lysines 9 des histones H3, ou l’apparition de variants d’histone (H3K4me, H3.3, H4A.Z) [18,
19,
22,
23]. Ces microARN pourraient avoir un rôle clé, soit dans le processus de la MSCI, soit dans la régulation post-transcriptionelle des ARNm autosomiques dans les stages postméiotiques de la spermatogenèse. Au stade spermatocyte secondaire et dans la spermatide pendant les premières étapes de la spermiogenèse, le Xp reste majoritairement non transcrit malgré la dissolution de ce corps XY. S.H. Namekawa a en effet observé que 87 % des gènes liés à l’X ne sont pas exprimés au stade spermatide contrairement à la majorité des gènes autosomiques [17]. Cela souligne la persistance de l’état inactif du chromosome X qui se matérialise alors sous forme d’une structure chromatinienne remodelée appelée PMSC (post meiotic sex chromatin).
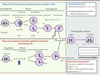 | Figure 1.
Inactivation des chromosomes sexuels au cours de la gamétogenèse et de l’embryogenèse précoce dans le sexe féminin. Au stade pachytène du spermatocyte primaire, les chromosomes X et Y s’apparient grâce à leurs régions pseudo-autosomiques pour former la vésicule sexuelle, ce qui engendre leur inactivation appelée MSCI (meiotic sex chromosome inactivation). L’état inactif du Xp (X paternel) persiste au stade spermatide sous forme d’une structure chromatinienne appelée PMSC (post meiotic sex chromatin). Il existe ensuite une réactivation du Xp dans les cellules postméiotiques de la lignée germinale mâle avant la fécondation de sorte que le Xp est transmis dans un état actif. Dans l’embryon de sexe féminin, l’inactivation du chromosome X soumise à empreinte intervient secondairement à l’accumulation de Xist sur le Xp, entraînant son extinction transcriptionnelle dès le stade 4 cellules. La nature de l’empreinte à l’origine de l’inactivation du Xp est inconnue. Il semble que le chromosome X maternel (Xm) soit porteur d’une marque apposée au cours de l’ovogenèse, empêchant l’inactivation du Xm aux stades précoces du développement. La protéine RNF12 d’origine maternelle pourrait agir en trans et activer l’expression de Xist à partir de l’allèle paternel. Le Xp conserverait aussi des traits chromatiniens d’hétérochromatine, expliquant l’inactivation de l’allèle paternel. |
-
Réactivation du chromosome Xp dans les cellules postméiotiques
Il existe ensuite une réactivation du chromosome Xp dans les cellules postméiotiques de la lignée germinale mâle avant la fécondation [19–
21]. En effet, la forme active de l’ARN polymérase II regagne le chromosome X au stade spermatide ronde, mettant un terme à son extinction transcriptionnelle [19]. Dans les spermatides rondes, une proportion significative de gènes liés à l’X, exprimés dans le testicule, échappent à la répression et deviennent actifs. Selon Mueller et al. [
24], 33 familles de gènes en copies multiples, représentant environ 273 gènes liés à l’X, sont exprimées dans les cellules postméiotiques à un niveau similaire à celui qui est observé dans les autosomes. Ainsi, en fin de spermiogenèse, certains gènes liés à l’X jusqu’alors silencieux peuvent être réexprimés comme le montre l’élévation significative en RT-PCR (reverse transcriptase polymerase chain reaction) des taux d’ARNm des gènes liés à l’X Ube1x (ubiquitin-like modifier activating enzyme 1) et Mhr6a au stade spermatide ronde par rapport au stade spermatocyte primaire [
20], ou la réactivation mosaïque du gène Ddx3x (DEAD box polypeptide 3, X-linked) observée par RNA FISH [
25]. D’autres gènes liés à l’X peuvent être transcrits de novo (exemple Sly [sycp3-like Y-linked]) [
26]. Selon P.J. Wang, la majorité des gènes liés à l’X sont effectivement réactivés à différents degrés aux stades postméiotiques, mais sans corrélation entre leur position sur le chromosome et le locus Xist [21]. On peut donc facilement imaginer que les modifications des histones déposées durant la spermiogenèse soient associées aux gènes actifs spécifiquement exprimés dans le testicule. Une étude récente plaide pour cette hypothèse : une nouvelle modification des histones, la crotonylation des lysines (Kcr) [
34] (→), est associée à un petit groupe de gènes liés à l’X et au Y qui échappent à l’inactivation dans les spermatides [
27]. Il se pourrait cependant que certaines séquences régulatrices de Xist conservent les caractéristiques chromatiniennes qui prévalent au début de la spermiogenèse (chromatine inactive) et jouent un rôle ultérieurement au cours du développement embryonnaire, expliquant l’inactivation du chromosome X paternel [
28].
(→) Voir l’article de E. Montellier et al., page 485 de ce numéro
|
Inactivation aléatoire du chromosome X Au stade tardif de maturation du blastocyste, le chromosome X paternel est réactivé au jour 4,5 dans les cellules de la masse cellulaire interne4. Cette réactivation se manifeste d’abord par la perte de l’ARN Xist qui recouvre le chromosome inactivé, puis par la perte des protéines du groupe Polycomb (EED/EZH2), suivie de la disparition des modifications des histones caractéristiques de l’état inactif (telles que les triméthylations de H3K27) et, au final, de la réactivation des gènes liés au chromosome X. Une étude récente suggère même que la réactivation de certains gènes et des séquences répétées surviendrait avant la disparition de Xist et de la marque d’hétérochromatine H3K27me2/3 sur le Xp [
29]. Cette réactivation pourrait être liée à la répression de Xist par les facteurs de pluripotence Oct4 (octamer-binding transcription factor 4) et Nanog dans les cellules préépiblastiques [
30]. Ces facteurs agiraient directement en réprimant Xist par leur liaison sur le premier intron, et indirectement en contrôlant des activateurs de Xist, comme RNF12 ou l’antisens Tsix [
31,
32,
35]. Aux jours 4,5-5 de gestation, les deux chromosomes X deviennent actifs. Cependant, dans l’embryon, ce statut est transitoire et ne dure que le temps de un à deux cycles cellulaires [3]. L’inactivation aléatoire d’un chromosome X survient immédiatement après, et représente un mécanisme essentiel à la survie de la souris femelle. Au jour 6,5, toutes les cellules embryonnaires ont inactivé un chromosome X au hasard, ce qui aboutit à l’expression monoallélique de la majorité des gènes qui lui sont liés. L’inactivation mise en place est extrêmement stable dans l’embryon, probablement à cause de la méthylation de l’ADN. Elle est ensuite transmise de façon clonale aux cellules filles au cours du développement, sauf au niveau des cellules germinales primordiales où le chromosome X inactivé est réactivé. Ce processus de réactivation est plus passif et plus lent que celui qui survient dans la masse cellulaire interne du blastocyste. Autant les données sont claires dans l’ovocyte où l’on observe une réactivation du chromosome Xm au cours de l’ovogenèse, autant celles dans le spermatozoïde sont plus incertaines : la transmission du Xp dans un état inactif ou non a fait l’objet de nombreuses controverses, même si actuellement la théorie de l’Xp transmis sous une forme préinactive est remise en cause. |
Le traitement différentiel des deux chromosomes X au cours de la fécondation et du développement embryonnaire précoce et une telle dynamique d’inactivation soulignent la plasticité des marques épigénétiques au cours de l’embryogenèse précoce. D’autre part, si le modèle murin a été particulièrement bien étudié, il semblerait que différentes stratégies soient utilisées par différents mammifères pour mettre en place ce processus [
33]. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Lyon
MF
. Gene action in the X-chromosome of the mouse (Mus musculus L.) . Nature.
1961; ; 190 : :372.–373. 2.
Mak
W
,
Nesterova
TB
,
de Napoles
M
, et al.
Reactivation of the paternal X chromosome in early mouse embryos . Science.
2004; ; 303 : :666.–669. 3.
Okamoto
I
,
Otte
AP
,
Allis
CD
, et al.
Epigenetic dynamics of imprinted X inactivation during mouse early development . Science.
2004; ; 303 : :644.–649. 4.
Tattermusch
A
.
Brockdorv
N
. A scaffold for X chromosome inactivation.
Hum Genet.
2011; ; 130 : :247.–253. 5.
Namekawa
SH
,
Payer
B
,
Huynh
KD
, et al.
Two-step imprinted X inactivation: repeat versus genic silencing in the mouse . Mol Cell Biol.
2010; ; 30 : :3187.–3205. 6.
Patrat
C
,
Okamoto
I
,
Diabangouaya
P
, et al.
Dynamic changes in paternal X-chromosome activity during imprinted X-chromosome inactivation in mice . Proc Natl Acad Sci USA.
2009; ; 106 : :5198.–5203. 7.
Chow
JC
,
Ciaudo
C
,
Fazzari
MJ
, et al.
LINE-1 activity in facultative heterochromatin formation during X chromosome inactivation . Cell.
2010; ; 11 : :956.–969. 8.
Okamoto
I
,
Arnaud
D
,
Le Baccon
P
, et al.
Evidence for de novo imprinted X chromosome inactivation independent of meiotic inactivation in mice . Nature.
2005; ; 438 : :369.–373. 9.
Lyon
M
,
Rastan
. Parental source of chromosome imprinting and its relevance for X chromosome inactivation.
Differentiation.
1984; ; 26 : :63.–67. 10.
Kay
GF
,
Barton
SC
,
Surani
MA
,
Rastan
S
. Imprinting and X chromosome counting mechanisms determine Xist expression in early mouse development . Cell.
1994; ; 77 : :639.–650. 11.
Marahrens
Y
,
Panning
B
,
Dausman
J
, et al.
Xist-deficient mice are defective in dosage compensation but not spermatogenesis . Genes Dev.
1997; ; 11 : :156.–166. 12.
Tada
T
,
Obata
Y
,
Tada
M
, et al.
Imprint switching for non-random X-chromosome inactivation during mouse oocyte growth . Development.
2000; ; 27 : :3101.–3105. 13.
Lee
JF
. Disruption of imprinted X inactivation by parent-of-origin effects at Tsix . Cell.
2000; ; 29 : :17.–27. 14.
Chiba
H
,
Hirasawa
R
,
Kaneda
M
, et al.
De novo DNA methylation independent establishment of maternal imprint on X chromosome in mouse oocytes . Genesis.
2008; ; 46 : :768.–774. 15.
Shin
J
,
Bossenz
M
,
Chung
Y
, et al.
Maternal Rnf12/RLIM is required for imprinted X-chromosome inactivation in mice . Nature.
2010; ; 21 : :977.–981. 16.
Inoue
K
,
Kohda
T
,
Sugimoto
M
, et al.
Impeding Xist expression from the active X chromosome improves mouse somatic cell nuclear transfer . Science.
2010; ; 330 : :96.–99. 17.
Namekawa
SH
,
Park
PJ
,
Zhang
LF
, et al.
Postmeiotic sex chromatin in the male germline of mice . Curr Biol.
2006; ; 16 : :660.–667. 18.
Song
R
,
Ro
S
,
Michaels
JD
, et al.
Many X-linked microRNAs escape meiotic sex chromosome inactivation . Nat Genet.
2009; ; 41 : :488.–493. 19.
Khalil
AM
,
Boyar
FZ
,
Driscoll
DJ
. Dynamic histone modifications mark sex chromosome inactivation and reactivation during mammalian spermatogenesis . Proc Natl Acad Sci USA.
2004; ; 101 : :16583.–16587. 20.
Hendriksen
PJ
,
Hoogerbrugge
JW
,
Themmen
AP
, et al.
Postmeiotic transcription of X and Y chromosomal genes during spermatogenesis in the mouse . Dev Biol.
1995; ; 170 : :730.–733. 21.
Wang
PJ
,
Page
DC
,
McCarrey
JR
. Differential expression of sex-linked and autosomal germ-cell-specific genes during spermatogenesis in the mouse . Hum Mol Genet.
2005; ; 14 : :2911.–2918. 22.
Greaves
IK
,
Rangasamy
D
,
Devoy
M
, et al.
The X and Y chromosomes assemble into H2A.Z, containing facultative heterochromatin, following meiosis . Mol Cell Biol.
2006; ; 26 : :5394.–5405. 23.
Khalil
AM
,
Driscoll
DJ
. Histone H3 lysine 4 dimethylation is enriched on the inactive sex chromosomes in male meiosis but absent on the inactive X in female somatic cells . Cytogenet Genome Res.
2006; ; 112 : :11.–15. 24.
Mueller
JL
,
Mahadevaiah
SK
,
Park
PJ
, et al.
The mouse X chromosome is enriched for multi-copy testis genes exhibiting post-meiotic expression . Nat Genet.
2008; ; 40 : :794.–799. 25.
Turner
JM
,
Mahadevaiah
SK
,
Ellis
PJ
, et al.
Pachytene asynapsis drives meiotic sex chromosome inactivation and leads to substantial postmeiotic repression in spermatids . Dev Cell.
2006; ; 10 : :521.–529. 26.
Touré
A
,
Clemente
EJ
,
Ellis
P
, et al.
Identification of novel Y chromosome encoded transcripts by testis transcriptome analysis of mice with deletions of the Y chromosome long arm . Genome Biol.
2005; ; 6 : :R102.. 27.
Tan
M
,
Luo
H
,
Lee
S
, et al.
Identification of 67 histone marks and histone lysine crotonylation as a new type of histone modification . Cell.
2011; ; 146 : :1016.–1028. 28.
Hammoud
SS
,
Nix
DA
,
Zhang
H
, et al.
Distinctive chromatin in human sperm packages genes for embryo development . Nature.
2009; ; 23 : :473.–478. 29.
Williams
LH
,
Kalantry
S
,
Starmer
J
,
Magnuson
T
. Transcription precedes loss of Xist coating and depletion of H3K27me3 during X-chromosome reprogramming in the mouse inner cell mass . Development.
2011; ; 138 : :2049.–2057. 30.
Navarro
P
,
Chambers
I
,
Karwacki-Neisius
V
, et al.
Molecular coupling of Xist regulation and pluripotency . Science.
2008; ; 321 : :1693.–1695. 31.
Barakat
TS
,
Gunhanlar
N
,
Pardo
CG
, et al.
RNF12 activates Xist and is essential for X chromosome inactivation . PLoS Genet.
2011; ; 27 : :e1002001.. 32.
Navarro
P
,
Oldfield
A
,
Legoupi
J
, et al.
Molecular coupling of Tsix regulation and pluripotency . Nature.
2010; ; 468 : :457.–460. 33.
Okamoto
I
,
Patrat
C
,
Thépot
D
, et al.
Eutherian mammals use different strategies to initiate X inactivation during development . Nature.
2011; ; 472 : :370.–374. 34.
Montellier
E
,
Rousseaux
S
,
Khochbin
S
. Feux croisés sur le nucléosome : bases moléculaires de la compaction du génome mâle haploïde . Med Sci (Paris).
2012; ; 28 : :485.–489. 35.
Rougeulle
C
. Inactivation du chromosome X : quand les facteurs de pluripotence s’en mêlent . Med Sci (Paris).
2009; ; 25 : :234.–235. |