| |
| Med Sci (Paris). 2012 May; 28(5): 547–550. Published online 2012 May 30. doi: 10.1051/medsci/2012285022.Chroniques génomiques Des signatures sans valeur ? Bertrand Jordan1* 1CoReBio PACA, case 901, parc scientifique de Luminy, 13288Marseille Cedex 9, France MeSH keywords: Animaux, Protocoles de polychimiothérapie antinéoplasique, usage thérapeutique, Biomarqueurs pharmacologiques, analyse, métabolisme, Marqueurs biologiques tumoraux, génétique, Interprétation statistique de données, Femelle, Analyse de profil d'expression de gènes, statistiques et données numériques, utilisation, Humains, Souris, Tumeurs, diagnostic, traitement médicamenteux, mortalité, Valeur prédictive des tests, Pronostic, Aliénation sociale, Troubles du comportement social, Analyse de survie |
La saga des signatures d’expression Dès la fin des années 1990, l’apparition des microarrays et la disponibilité d’informations de plus en plus précises sur les gènes humains ont offert la possibilité de mesurer le niveau d’expression de nombreux gènes dans des échantillons biologiques – et suscité l’espoir d’en tirer des informations cliniques utiles, notamment en cancérologie. Si l’on se limite au cancer du sein, près de cinquante articles sont parus qui présentent des « signatures d’expression », c’est-à-dire qui montrent que la mesure du niveau d’expression d’un jeu de gènes dans un échantillon tumoral permet d’évaluer le pronostic de la malade et, dans certains cas, de prévoir le niveau d’efficacité d’une chimiothérapie. La
Figure 1
montre l’un des exemples les plus connus, celui de la « signature d’Amsterdam », publiée en 2002, [
1] qui porte sur 70 gènes et classe les malades en deux catégories dont le pronostic est très nettement différent (voir [
16]) (→).
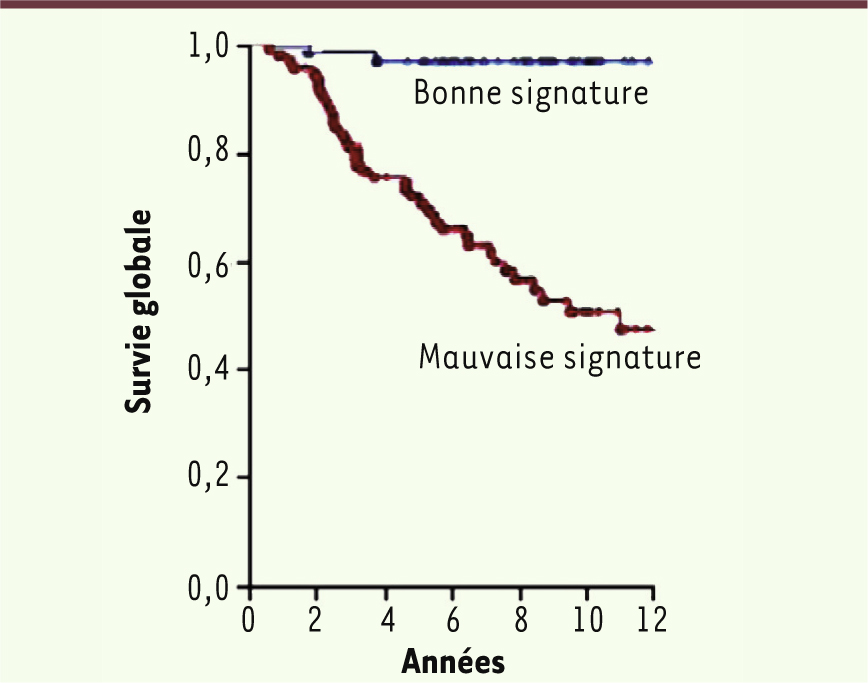
 | Figure 1
Courbes de survie de femmes présentant un cancer du sein sans envahissement ganglionnaire. Les courbes présentent soit une « bonne » (en bleu), soit une « mauvaise » (en rouge) signature d’expression selon l’étude de Van de Vijver
et al. [ 1]. Extrait partiel et remanié de la figure 2 de cet article. |
(→)Voir m/s hors série n°1, mars 2012
On se souvient que c’est à partir de ces résultats qu’a été fondée l’entreprise Agendia, qui commercialise depuis 2004 un test appelé MammaPrint [16]. De nombreuses autres signatures, portant sur le même cancer ou sur d’autres localisations et comportant un nombre de gènes très variable, d’une dizaine à un millier, ont été publiées et ont, dans quelques cas, donné naissance à des tests commercialisés. |
L’ère du soupçon… et son épilogue ? Mais ces travaux ont fait l’objet de nombreuses critiques [
2–
4]. Au début, il s’est surtout agi de relever les imperfections techniques. Cela concernait notamment la caractérisation des « sondes » présentes sur le microarray, surtout à l’époque où il s’agissait de clones d’ADNc dont l’entretien et la mise en œuvre sont délicats, le passage aux oligonucléotides ayant considérablement simplifié les choses. On a aussi mis en cause la reproductibilité des hybridations, la précision des mesures d’intensité ou le fait que, durant les premières années, les mesures n’étaient pas systématiquement faites en double ou en triple1 comme S. Michiels et C.Hill le discutaient dans ces colonnes en 2008 [
17]. Ces problèmes furent peu à peu résolus, grâce notamment au travail du consortium microarray quality control [
5] et au départ de la fabrication des microarrays vers les entreprises, mieux à même de maîtriser la qualité d’un processus de fabrication complexe que les laboratoires universitaires. Mais ce sont alors les méthodes d’analyse des données qui furent mises en cause. Ces études souffrent en effet du « fléau de la dimension » : un important déséquilibre entre le nombre de données (des dizaines de milliers de niveaux d’expression) et le nombre d’« observations » (une ou deux centaines d’échantillons). Comme les mesures sont affectées d’un « bruit » assez élevé (avec des fluctuations d’au moins 20 ou 30 %, beaucoup plus pour les faibles valeurs), il s’avère que l’on peut facilement élaborer des classifications qui semblent corrélées aux données cliniques, mais qui reposent en fait sur les variations aléatoires des signaux. Cela apparaît clairement lorsque l’on teste la signature sur un nouveau jeu d’échantillons totalement indépendants : on constate souvent qu’elle a perdu toute valeur prédictive. Encore faut-il pratiquer ce test essentiel sur des échantillons réellement indépendants2 – ce dont beaucoup des premières études se sont dispensées… Une fois ces questions levées, encore faut-il apprécier si, finalement, cette signature techniquement et statistiquement valide apporte un réel plus clinique, c’est-à-dire aboutit à une meilleure survie, à une diminution des effets secondaires (en évitant des chimiothérapies inutiles) et/ou à une diminution des coûts. C’est là le but de grands essais cliniques encore en cours, comme le programme MINDACT [
6] qui porte sur 6 000 patientes et vise à examiner l’apport clinique du test MammaPrint. |
Un réexamen grâce aux données disponibles sur Internet Mais la saga ne semble pas terminée, du moins si l’on en croit un très intéressant article paru récemment dans une revue relativement obscure (nous reviendrons sur ce point) : son titre affirme que « la plupart des signatures choisies au hasard sont significativement corrélées à la survie à la suite d’un cancer du sein » [
7] – en d’autres termes, que les jeux de gènes définis après moult études par différentes équipes ne sont pas plus informatifs qu’un assortiment aléatoire ! Des publications avaient déjà mis en cause l’unicité d’un tel choix [
8] – mais de là à prendre les gènes au hasard, le saut est notable… Voyons donc comment ont procédé les auteurs, biologistes et bioinformaticiens à l’Université libre de Bruxelles. Ils ont en fait effectué leur étude en utilisant des données d’expression disponibles sur Internet, et ont, très logiquement, choisi celles qui sous-tendent l’article de Van de Vijver déjà cité [1]. Rappelons qu’il s’agit d’échantillons tumoraux congelés provenant de 295 malades suivis en moyenne 8 ans après le traitement, et que les profils d’expression avaient été déterminés, en collaboration avec Rosetta Inpharmatics (Kirkland, Wash, États-Unis), à l’aide de microarrays comportant des oligonucléotides longs mesurant l’expression de près de 25 000 gènes3,. L’ensemble des résultats d’hybridation a été téléchargé à partir du site de Rosetta, de même que les données cliniques disponibles pour chaque patiente. Les auteurs ont choisi comme paramètre principal la survie globale. Ils peuvent alors repérer, au sein de cet ensemble, le jeu de gènes correspondant à une signature publiée, et examiner, avec les valeurs d’expression répertoriées, si cette signature est en effet significativement corrélée avec la survie. La description que je donne ici est naturellement un peu simplifiée : il a fallu régler de délicats problèmes d’annotation (parle-t-on bien des mêmes gènes dans les données de Van de Vijver en 2002 et dans la signature X en 2010 ?), et utiliser une technique de classification unique et robuste pour toutes les signatures – mais tout ceci est bien décrit dans l’article et dans ses suppléments [7]. |
La défaite sociale chez la souris ? Une des premières signatures testées par les auteurs avait été publiée en 2007 dans la revue Cell, et concerne « la résilience vis-à-vis de la défaite sociale » chez la souris [
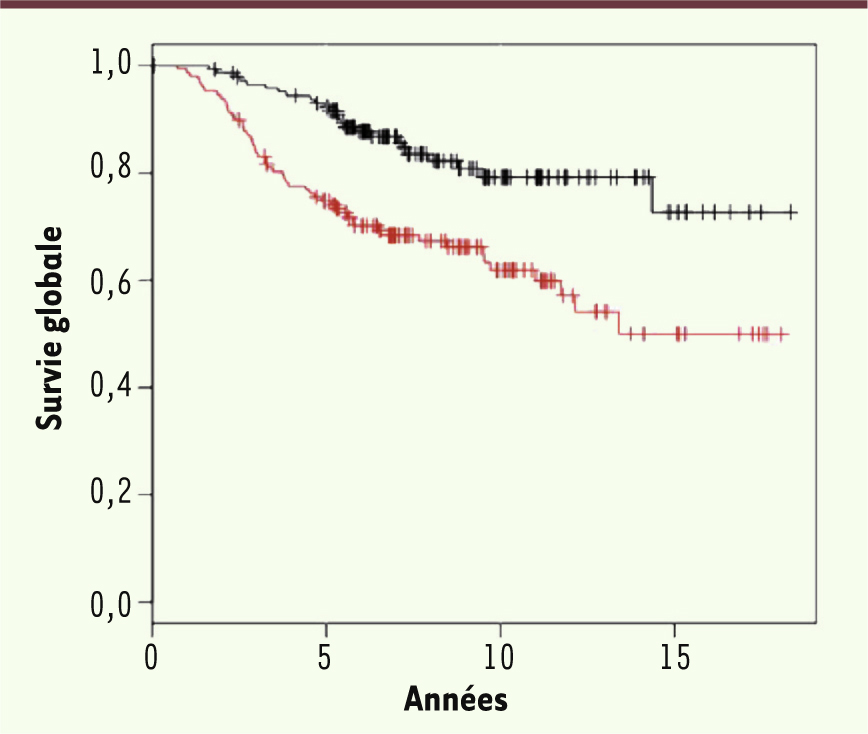
9]. Ce sujet peut prêter à sourire, mais il s’agit d’un travail tout à fait sérieux montrant que, après une série d’épreuves traumatisantes, des souris génétiquement identiques peuvent rester durablement déprimées ou au contraire récupérer rapidement leurs compétences sociales. Et cet état de sensibilité ou de résistance se traduit, dans leur cerveau, par des profils d’expression nettement distincts permettant l’élaboration d’une signature. Celle-ci n’a évidemment rien à voir avec la survie après un cancer du sein. Néanmoins, en l’utilisant avec les données de Van de Vijver (avec les gènes humains correspondant à ceux de la signature chez la souris) on obtient un classement assez convaincant des malades (Figure 2) !
 | Figure 2
Classement des patientes grâce à la signature d’expression de la défaite sociale chez la souris [ 7, 9]. En rouge : patientes dont le spectre d’expression correspond à la catégorie « sensible ». Le risque relatif de décès pour cette catégorie s’élève à 2,4 avec une valeur de p de 0,00014 – la classification est donc tout à fait significative statistiquement. Graphique remanié extrait de la figure 1 de [ 9] (© licence Creative Commons).
|
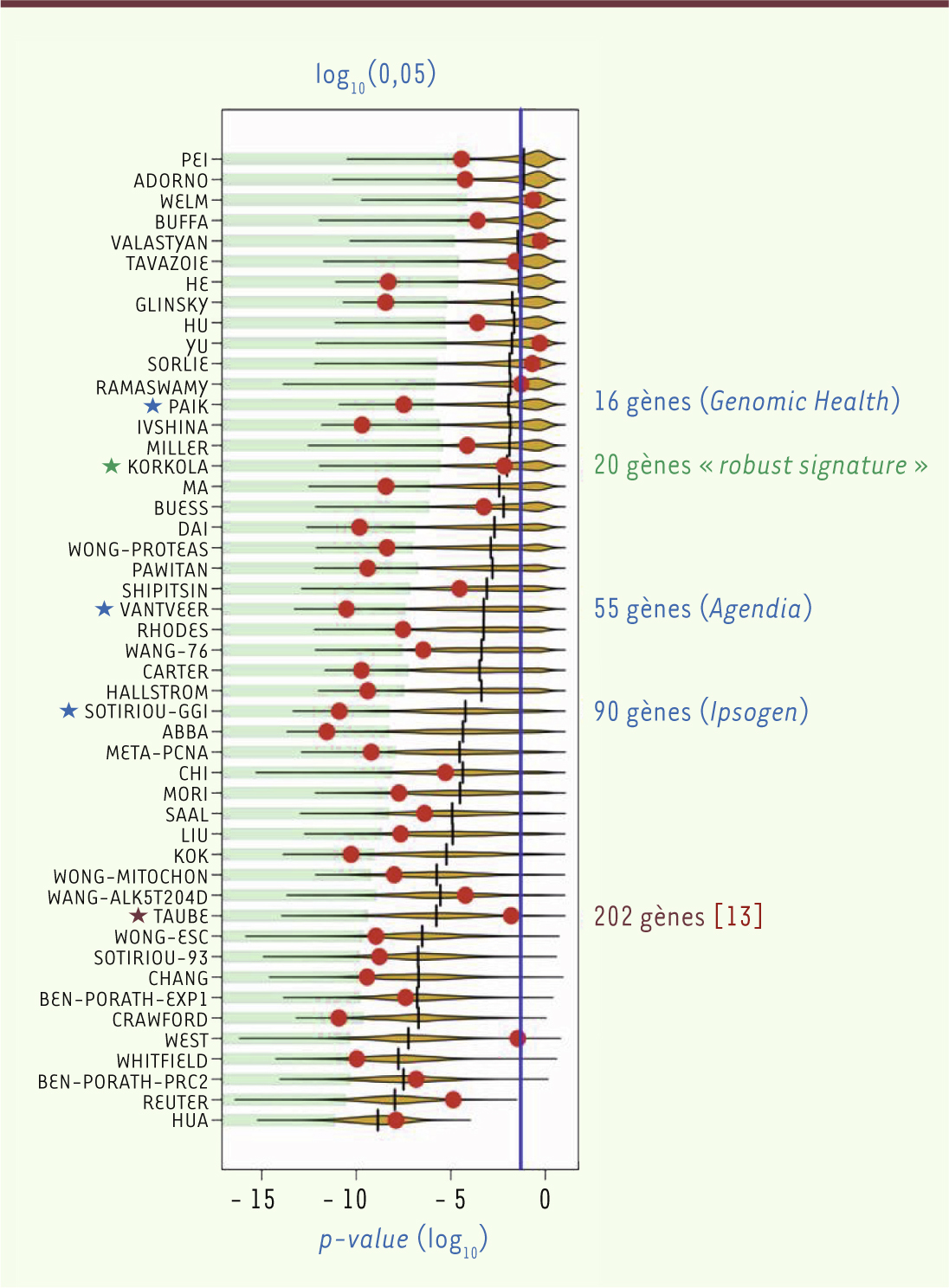
Ces résultats surprenants et même choquants ont été généralisés par les auteurs de manière systématique. Ils ont collationné les 47 signatures d’expression publiées4 relatives au cancer du sein (portant sur un nombre de gènes qui va de deux à plus de mille), et ont pour chacune testé mille signatures de même taille mais dont les gènes sont pris au hasard. Et ils ont ensuite examiné la valeur prédictive (pour la survie après cancer du sein) de chacun des profils obtenus. Les résultats sont présentés dans la
Figure 3
, dont le message est que « la plupart des signatures publiées ne sont pas significativement meilleures que des signatures de même taille prises au hasard ». Sur cette figure, les signatures sont rangées par ordre croissant de taille, la première comportant deux gènes, la dernière 1160. La barre verticale bleue correspond à la valeur de p = 0,05, souvent prise comme seuil de validité statistique. Dans chaque cas, les mille signatures au hasard correspondent au fuseau jaune (l’épaisseur du fuseau représentant l’effectif pour chaque valeur de p), leur médiane est figurée par un trait vertical noir. Enfin, la valeur obtenue en utilisant la signature publiée est signalée par le point rouge. Une bonne signature devrait donc être figurée par un point rouge situé très à gauche, loin sur la « queue » du fuseau des signatures au hasard. On voit – c’est rassurant – que c’est bien le cas pour la signature de Paik et al. [
10] correspondant au test Oncotype de l’entreprise Genomic Health, pour celle de Van de Vijver et al. [1] (repérée par « VANTVEER ») dont découle le test MammaPrint (Agendia) et pour le Genomic Index de Sotiriou et al. [
11] (Genomic Index, Ipsogen). Les auteurs ont donc un peu forcé leur message, mais il est vrai que beaucoup de signatures ne sont pas, ou guère, meilleures que la médiane des signatures au hasard, comme celle de Korkola et al. pourtant dite « robuste » dans le titre même de l’article [
12], et que certaines, comme celle de Taube et al. [
13] récemment parue dans le Proceedings of the National Academia of Sciences of USA et portant sur 202 gènes, sont bien pires.
 | Figure 3
Test de 47 signatures publiées (voir le texte). Extrait remanié de la figure 2 de l’article [ 7] (© Licence Creative Commons)
|
La suite de l’article montre comment ces résultats assez surprenants s’expliquent par le caractère dominant d’une signature de prolifération peu spécifique, impliquant de très nombreux gènes (près de la moitié de ceux examinés), et par le fait que la tendance à la prolifération a en effet une valeur prédictive par rapport à l’évolution d’un cancer. C’est un résumé schématique des conclusions de ce très intéressant article, mais comme le cadre d’une chronique est déjà presque dépassé, venons-en aux conclusions… sans oublier de mentionner un tout récent « papier » émanant de quelques-uns des meilleurs auteurs du domaine et montrant que l’on peut réduire à trois le nombre de gènes nécessaire à une bonne classification des cancers du sein [
14]. |
Les statistiques, décidément… On aurait pu penser que les « génomistes » avaient compris l’importance d’un traitement statistique rigoureux des masses considérables de données accumulées grâce aux récentes avancées technologiques : il semble bien que le processus ne soit pas encore terminé. Pour les « balayages du génome » (GWAS), l’analyse semble sous contrôle, et on est loin du Far West des années 1990 où des conclusions contradictoires étaient tirées à partir de l’analyse de quelques centaines de marqueurs sur quelques dizaines de patients et témoins. Pour les profils d’expression, la validité statistique des corrélations entre une signature d’expression et un paramètre clinique est maintenant assurée, on ne teste plus un classificateur sur des patients dont certains ont servi à son établissement, mais la qualité du classificateur par rapport à d’autres classificateurs possibles n’est en général pas évaluée [8]. L’article de Venet et al. [7] ne remet pas en cause la validité clinique des signatures publiées : elles sont en effet, pour la plupart, significativement corrélées avec la survie globale – mais beaucoup ne sont pas mieux corrélées que bien des signatures de même taille choisies au hasard. Cela signifie que l’on ne peut pas en tirer – comme c’est souvent fait – des hypothèses sur les mécanismes biologiques en dissertant sur la nature et la fonction présumée des gènes constituant la signature, puisqu’un autre choix aurait pu être tout aussi performant. Il ne suffit donc pas de démontrer une corrélation entre la signature et le paramètre biologique, il faut aussi que cette corrélation soit bien meilleure que celle observée avec la majorité des signatures au hasard de même taille. Comme on le voit sur la
Figure 3
, si le nombre de gènes est élevé, une signature doit avoir une valeur de p de l’ordre de 10−10 ou 10−11… et non de 5.10−2, pour que l’on puisse fonder une hypothèse biologique sur l’identité des gènes qui la constituent. |
La résistance de l’ establishment
Pour terminer, force est de noter une considérable résistance du milieu à la remise en cause d’une approche largement pratiquée, donnant lieu à des publications prestigieuses et à des applications commerciales prometteuses. Comme le raconte [
15] Vincent Detours, auteur senior de l’article [7], il a fallu quatre années et six refus de publication pour que ce travail paraisse finalement dans une revue d’audience assez limitée… et pendant ce temps d’autres signatures d’expression aussi peu valides ont continué à paraître dans les journaux les plus prestigieux. On peut certes discuter tel ou tel point du travail rapporté dans cette chronique, et nuancer certaines assertions des auteurs5, mais il semble évident que cette étude aurait mérité une publication rapide, un grand retentissement – et aurait dû déclencher un examen beaucoup plus précis des données avancées par de nombreux auteurs, et surtout de leurs conclusions. Que cela n’ait pas eu lieu montre une regrettable résistance des « grandes » revues à la remise en cause de ce qui est presque devenu un paradigme. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. |
Footnotes |
1.
Van de Vijver
MJ
,
He
YD
,
van’t Veer
LJ
, et al.
A gene-expression signature as a predictor of survival in breast cancer . N Engl J Med.
2002; ; 347 : :1999.–2009. 2.
Tan
PK
,
Downey
TJ
,
Spitznagel
EL
Jr
. Evaluation of gene expression measurements from commercial microarray platforms . Nucleic Acids Res.
2003; ; 31 : :5676.–5684. 3.
Jordan
BR
. How consistent are gene expression chip platforms?
BioEssays.
2004; ; 26 : :1236.–1242. 4.
Jordan
B
. Coup de tabac sur les puces . Med Sci (Paris).
2004; ; 20 : :487.–490. 5.
MAQC Consortium . The MicroArray Quality Control (MAQC) project shows inter- and intraplatform reproducibility of gene expression measurements . Nat Biotechnol.
2006; ; 24 : :1151.–1161. 6.
Cardoso
F
,
Piccart-Gebhart
M
,
Van’t Veer
L
,
Rutgers
E
. TRANSBIG Consortium. The MINDACT trial: the first prospective clinical validation of a genomic tool . Mol Oncol.
2007; ; 1 : :246.–251. 7.
Venet
D
,
Dumont
JE
,
Detours
V
. Most random gene expression signatures are significantly associated with breast cancer outcome . PLoS Comput Biol.
2011; ; 7 : :e 1002240.. 8.
Ein-Dor
L
,
Zuk
O
,
Domany
E
. Thousands of samples are needed to generate a robust gene list for predicting outcome in cancer . Proc Natl Acad Sci USA.
2006; ; 103 : :5923.–5928. 9.
Krishnan
V
,
Han
MH
,
Graham
DL
, et al.
Molecular adaptations underlying susceptibility and resistance to social defeat in brain reward regions . Cell.
2007; ; 131 : :391.–404. 10.
Paik
S
,
Shak
S
,
Tang
G
, et al.
A multigene assay to predict recurrence of tamoxifen-treated, node-negative breast cancer . N Engl J Med.
2004; ; 351 : :2817.–2826. 11.
Sotiriou
C
,
Wirapati
P
,
Loi
S
, et al.
Gene expression profiling in breast cancer: understanding the molecular basis of histologic grade to improve prognosis . J Natl Cancer Inst.
2006; ; 98 : :262.–272. 12.
Korkola
JE
,
Blaveri
E
,
DeVries
, et al.
Identification of a robust gene signature that predicts breast cancer outcome in independent data sets . BMC Cancer.
2007; ; 7 : :61.. 13.
Taube
JH
,
Herschkowitz
JI
,
Komurov
K
, et al.
Core epithelial-to-mesenchymal transition interactome gene-expression signature is associated with claudin-low and metaplastic breast cancer subtypes . Proc Natl Acad Sci USA.
2010; ; 107 : :15449.–15454. 14.
Haibe-Kains
B
,
Desmedt
C
,
Loi
S
, et al.
A three-gene model to robustly identify breast cancer molecular subtypes . J Natl Cancer Inst.
2012; ; 104 : :311.–325. 16.
Bertucci
F
,
Birnbaum
D
. Génomique et recherche clinique en cancérologie mammaire . Med Sci (Paris).
2012 ; ( Suppl 1) : :14.–18. 17.
Michiels
S
,
Hill
C
. Défis statistiques posés par les biopuces : autant d’espoir que de faux positifs ?
Med Sci (Paris).
2008; ; 24 : :317.–319
|