| |
| Med Sci (Paris). 2012 October; 28(10): 819–821. Published online 2012 October 12. doi: 10.1051/medsci/20122810007.Une micropuce de diagnostic hybride pour la détection d’ARN non codants Konstantinia Skreka1 and Mathieu Rederstorff1,2* 1Innsbruck biocenter, département de génomique et RNomique, faculté de médecine, Innrain, 6020Innsbruck, Autriche 2Université de Lorraine, Biopôle, CNRS UMR 7214 AREMS (ARN-RNP, structure-fonction-maturation, enzymologie moléculaire et structurale), 9, avenue de la forêt de Haye, Vandoeuvre-lès-Nancy, 54506, France MeSH keywords: Techniques de chimie combinatoire, utilisation, ADN, analyse, Séquençage nucléotidique à haut débit, méthodes, Humains, Analyse sur microréseau, Modèles biologiques, ARN non traduit, génétique, RT-PCR |
Malgré l’importance majeure des ARN messagers (ARNm), les transcriptomes eucaryotes sont essentiellement constitués d’ARN non messagers ou ARN non codants (ARNnc) pour des protéines. Ces derniers sont impliqués dans des processus biologiques variés mettant en jeu des mécanismes moléculaires et cellulaires multiples. Récemment, les ARNnc ont même été décrits comme d’excellents marqueurs diagnostiques pour de nombreuses pathologies, notamment en cancérologie [
1]. |
Séquençage à haut débit ou micropuces : le débat fait rage Pour la réalisation d’une analyse transcriptomique, les approches basées sur le séquençage à haut débit tendent à remplacer progressivement les approches basées sur l’utilisation de micropuces. En effet, ces dernières nécessitent la connaissance a priori des séquences à étudier. Elles sont opaques à l’identification de nouvelles molécules, telles que des nouveaux variants issus de l’épissage alternatif d’un préARNm ou des nouveaux petits ARNnc, contrairement aux approches par séquençage dites « ouvertes ». Les approches par séquençage sont, en revanche, encore peu utilisées en diagnostic [
2,
3]. Récemment, plusieurs études ont été entreprises avec comme objectif la comparaison de l’efficacité, de la sensibilité et de la reproductibilité de ces deux approches majeures de transcriptomique. L’analyse d’échantillons biologiques ou d’une population synthétique contrôlée d’ARN a permis de comparer les résultats obtenus par séquençage ou micropuce. Bien que les deux approches apparaissent très reproductibles et spécifiques, les approches basées sur l’utilisation de puces demeurent sensiblement plus précises en termes de quantification absolue d’espèces très peu abondantes [
4,
5]. Malheureusement, les conditions optimisées d’analyse par micropuce d’ARNm ne peuvent pas s’appliquer à l’étude des ARNnc. En effet, la très grande hétérogénéité de ces derniers en termes de taille, d’abondance et de structures secondaires rend leur étude par ce type d’approches particulièrement complexe, en raison de l’impossibilité de produire des sondes adaptées à l’ensemble des molécules. D’autre part, les gradients de température utilisés pour permettre l’hybridation, dans un premier temps des plus grands ARN sur leurs sondes, puis ensuite celle des plus petits ARN, occasionnent de nombreuses hybridations non spécifiques et un bruit de fond parfois très important [
6]. Les puces dédiées à l’analyse des seuls microARN (miARN) sont en revanche utilisées en routine dans de nombreux laboratoires. Ces puces sont généralement composées d’oligonucléotides de type LNA (locked nucleic acids) (Figure 1), qui correspondent à des analogues d’ARN thermostables présentant une affinité extrême pour leurs acides nucléiques complémentaires. Ainsi, l’utilisation d’oligonucléotides comportant des résidus de type LNA dans leur séquence permet d’obtenir, même pour des tailles réduites, des températures de fusion et d’hybridation spécifique élevées.
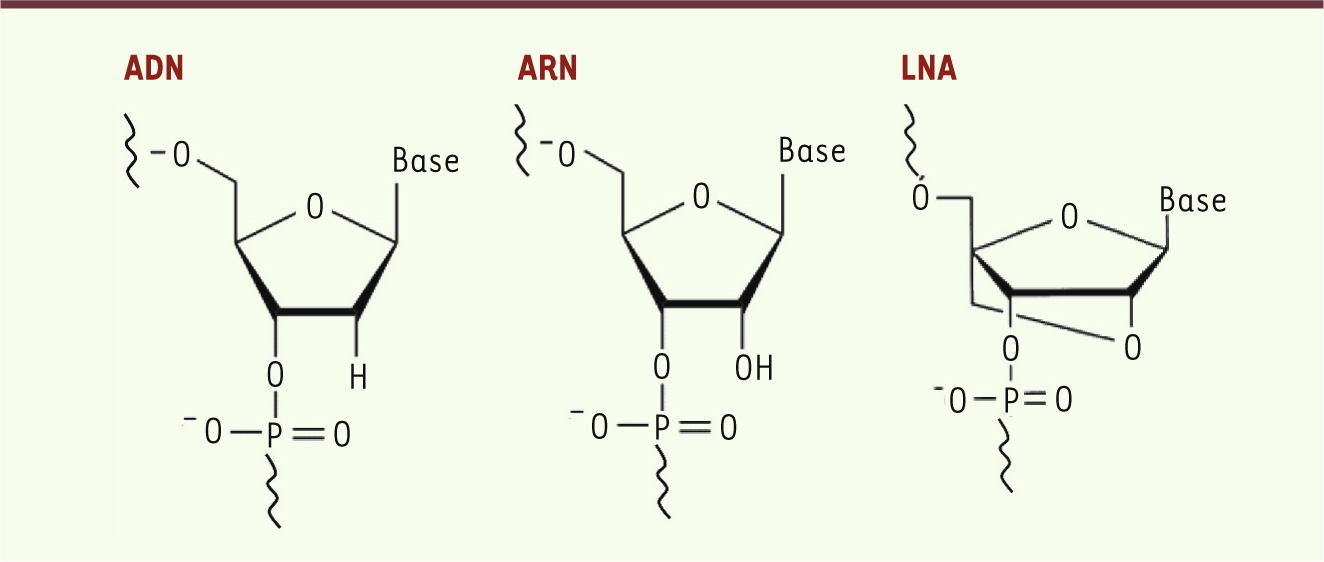
 | Figure 1.
Structure de nucléotides de type ADN, ARN et LNA. Un nucléotide LNA (locked nucleic acids) est un analogue synthétique stable d’ARN, dont la conformation 3’endo est stabilisée par un pont méthylène entre l’atome d’oxygène en position 2’ et l’atome de carbone 4’. De ce fait, la température de fusion d’un oligonucléotide comportant des résidus LNA dans sa séquence sera bien plus élevée que celle d’un oligonucléotide ADN ou ARN de séquence identique. |
Pour l’étude des miARN, des collections d’oligonucléotides LNA, prêtes à être fixées sur puces ou déjà associées à leur support, sont disponibles dans le commerce et offrent une spécificité d’hybridation et de discrimination importante des miARN, malgré la taille réduite des séquences utilisées. L’utilisation de micropuces entièrement basées sur ce type de technologie LNA n’est toutefois pas envisageable pour des approches de grande envergure en raison de leur coût prohibitif. À l’inverse, les puces à ADN, qui sont plus accessibles financièrement, ne présentent pas quant à elles la sensibilité indispensable à la détection spécifique de très petites séquences. Ainsi, il n’existe pas d’approche globale permettant l’analyse simultanée d’un transcriptome non codant très hétérogène de nature. |
Une micropuce hybride pour l’analyse simultanée d’une population hétérogène d’ARN Pour palier ces difficultés, nous avons développé une approche innovante consistant à combiner sur les micropuces des oligonucléotides courts de type LNA et des oligonucléotides classiques ADN plus longs, dédiés respectivement à l’hybridation (dans les mêmes conditions expérimentales, notamment de température) de très petits ARN (miARN) et d’ARNnc plus longs et plus structurés (Figure 2) [
7]. La seule contrainte de cette approche consiste ainsi à générer des oligonucléotides (LNA et DNA) présentant des températures de fusion identiques, de façon à pouvoir les utiliser au cours de la même expérience.
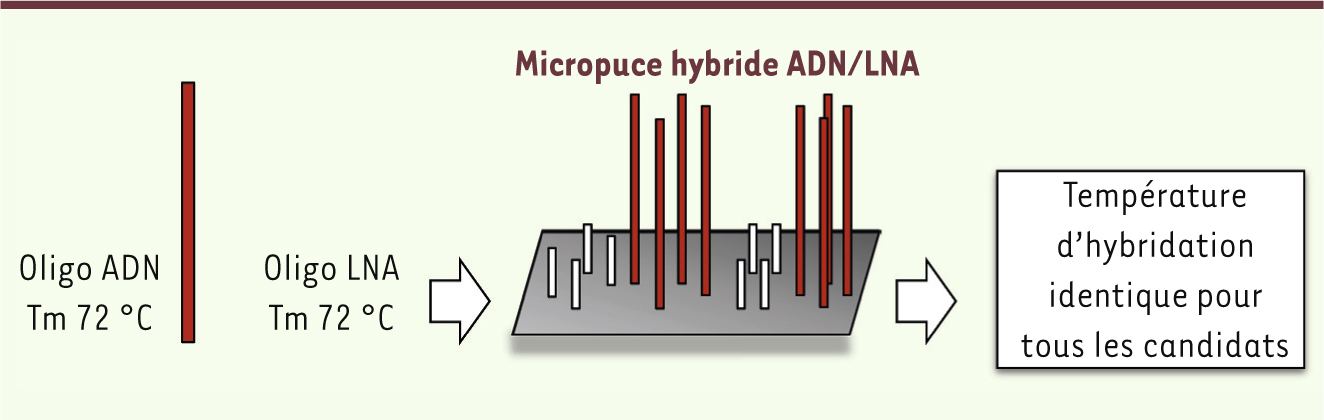
 | Figure 2.
Principe de la puce hybride ADN/LNA. L’utilisation combinée sur le support d’oligonucléotides longs, de structure ADN, et d’oligonucléotides plus courts, de structure LNA, présentant la même température de fusion, permet l’analyse simultanée d’une population très hétérogène d’ARNnc [ 7]. Tm : température moyenne. |
Pour valider notre méthode, nous avons utilisé la puce hybride pour l’analyse simultanée de miARN et de longs ARNnc structurés (ARN de transfert [ARNt], petits ARN nucléolaires [snoARN]) à partir d’ARN total de cerveau murin. Puis, nous avons testé le système pour analyser l’expression différentielle de différents ARNnc entre des cellules souches embryonnaires de souris et des cellules neuronales murines différenciées. La méthode a permis de confirmer l’expression différentielle de miARN entre les cellules souches embryonnaires et les cellules de cerveau adulte (comme décrit dans la littérature), mais également de détecter celle de snoARN. C’est une observation intéressante parce qu’elle suggère l’implication possible de ces snoARN dans les processus de différenciation cellulaire. Les données obtenues avec la puce hybride ont ensuite été validées par northern-blotting et RT-PCR (reverse-transcriptase polymerase chain reaction) quantitative. L’incorporation progressive de marqueurs correspondant à des ARNnc dans les outils diagnostiques en clinique devrait permettre d’affiner le pronostic et l’évolution de nombreuses pathologies. Plus le nombre de biomarqueurs identifiés de ce type sera important, plus les diagnostics gagneront en précision. Ce type d’analyse pourrait, à terme, être réalisé en ayant recours à des puces hybrides ADN/LNA, permettant aussi bien l’analyse de très grands ARN structurés (ARN BC200 et maladie d’Alzheimer) que de très petits ARN (miARN et cancer), simultanément et à un moindre coût, ce d’autant que les quantités de matériel nécessaires pour ce type d’approche sont compatibles avec les références actuelles en diagnostic clinique. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Rederstorff
M.
Une approche originale de sélection de nouveaux ARN non codants . Med Sci (Paris).
2011; ; 27 : :343.–345. 2.
Jordan
B.
Chroniques génomiques : séquençage de nouvelle génération : déjà en clinique ?
Med Sci (Paris).
2011; ; 27 : :1127.–1130. 3.
Jordan
B.
Is there a niche for DNA microarrays in molecular diagnostics?
Expert Rev Mol Diagn.
2010; ; 10 : :875.–882. 4.
Marioni
JC
,
Mason
CE
,
Mane
SM
, et al.
RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays . Genome Res.
2008; ; 18 : :1509.–1517. 5.
Willenbrock
H
,
Salomon
J
,
Sokilde
R
, et al.
Quantitative miRNA expression analysis: comparing microarrays with next-generation sequencing . RNA.
2009; ; 15 : :2028.–2034. 6.
Hutzinger
R
,
Mrazek
J
,
Vorwerk
S
,
Huttenhofer
A.
NcRNA-microchip analysis: A novel approach to identify differential expression of noncoding RNAs . RNA Biol.
2010; ; 7 : :586.–595. 7.
Skreka
K
,
Zywicki
M
,
Karbiener
M
, et al.
Expression profiling of a heterogeneous population of ncRNAs employing a mixed DNA/LNA microarray . J Nucleic Acids.
2012; ; 2012 : :283560.. |