| |
| Med Sci (Paris). 2012 October; 28(10): 893–896. Published online 2012 October 12. doi: 10.1051/medsci/20122810020.Chroniques génomiques Rare is frequent Bertrand Jordan1* 1CoReBio PACA, case 901, parc scientifique de Luminy, 13288Marseille Cedex 9, France MeSH keywords: Dosage génique, physiologie, Fréquence d'allèle, Variation génétique, Génome humain, génétique, Humains, Polymorphisme de nucléotide simple, Spécificité d'espèce |
Paraphrasant des expressions apparemment paradoxales comme Less is more ou Small is beautiful, le titre de cette chronique renvoie, en fait, aux variations repérées dans notre ADN par les nouvelles approches de séquençage à grande vitesse [
1], à l’irritante question de « l’héritabilité manquante » [
2] et, au-delà, à l’histoire démographique très particulière de notre espèce. Ces résultats ont déjà été rapidement évoqués dans une Brève, mais ils sont suffisamment importants pour que j'y revienne dans cette chronique (→) [
3].
(→) Voir m/s n° 8-9, août-septembre 2012, page 721
|
La diversité vue au niveau des snip L’analyse de la diversité génétique humaine a fait un grand bond en avant depuis que l’emploi de puces à ADN a autorisé l’étude de centaines de milliers de polymorphismes dans le génome de milliers d’individus [
4]. On a pu ainsi repérer l’appartenance de personnes à des groupes d’ascendance liés à l’histoire et à la géographie de l’humanité (groupes qui ne constituent néanmoins pas des « races » [
5]), et aussi reprendre sur des bases enfin solides l’analyse des déterminants génétiques de maladies complexes. Les très nombreux « balayages du génome » (GWAS pour genome-wide association studies) pratiqués ces dernières années se sont néanmoins révélés décevants [2]. En effet, les gènes ainsi identifiés, bien que statistiquement significatifs et confirmés par des études indépendantes, ne rendent compte que d’une petite partie de l’héritabilité des affections en cause. La majeure partie de cette héritabilité (parfois jusqu’à 80 ou 90 %) reste inexpliquée, ce qui rend d’ailleurs toute prédiction à la Navigenics
1 plus qu’aléatoire [
6]. Une explication avancée depuis quelque temps repose sur le fait que, par construction, ces études ne s’intéressent qu’aux polymorphismes fréquents, c’est-à-dire ceux pour lesquels l’allèle mineur est présent chez au moins 5 % de la population. Précisons un peu ce point. Les puces à ADN les plus perfectionnées ne peuvent comporter que quelques millions de segments d’ADN différents à leur surface (ce qui est déjà une performance assez extraordinaire). Du coup, elles n’analysent dans l’ADN qui leur est appliqué qu’un nombre limité de locus, disons un million pour fixer les idées. Or il y a potentiellement trois milliards de points de variation possibles dans un génome haploïde humain. Les puces ciblent donc les polymorphismes les plus fréquents, les snip repérés par de nombreuses études et pour lesquels on s’est fixé arbitrairement une limite de fréquence de 5 % pour l’allèle mineur (l’allèle majeur étant alors présent à 95 % dans la population puisque ces locus sont quasiment tous bialléliques). On pouvait espérer que, comme formulé dans l’hypothèse common disease/ common variant [
7], la majeure partie du déterminisme génétique des maladies fréquentes serait portée par de tels variants. Le résultat des milliers d’analyses GWAS menées à ce jour montre clairement que ce n’est pas le cas. Il ne reste donc plus qu’à répertorier les variations dans l’ensemble du génome, c’est-à-dire à le séquencer intégralement. La logique du raisonnement est impeccable, mais séquencer (sans erreurs, bien sûr) quelques milliards de nucléotides chez quelques milliers de personnes, ce n’est pas une mince affaire ! |
SNV, SNP, qualité de la séquence et nombre de variants C’est pourtant ce qu’ont réalisé les auteurs de deux articles parus au début de l’été dans la revue Science [
8,
9] et accompagnés d’un commentaire détaillé [
10]. Pour des raisons évidentes, il ne s’agit pas (encore…) de séquence intégrale, mais néanmoins des exomes [
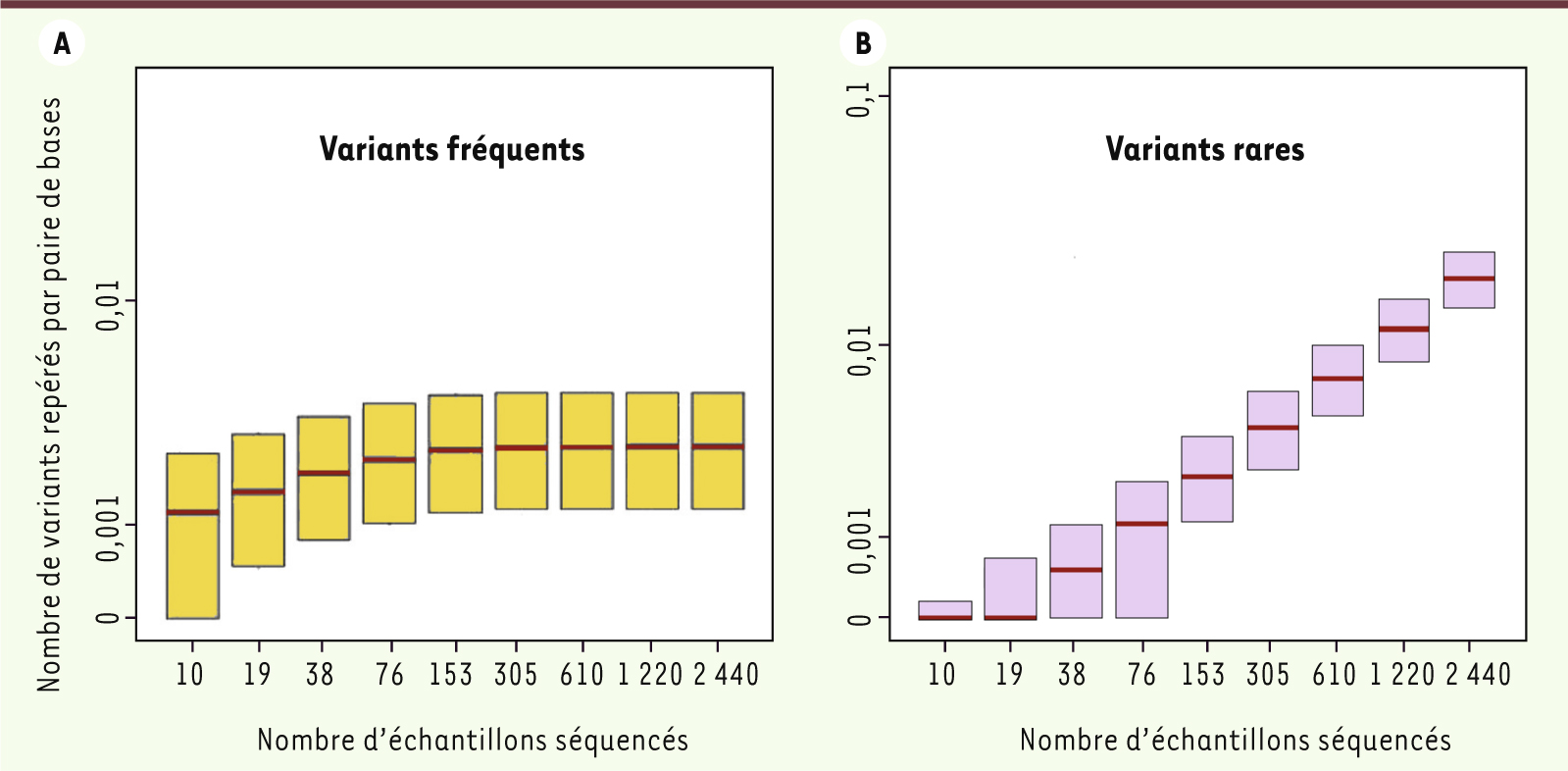
11] complets de plus de deux mille personnes dans un cas [8], de 202 gènes choisis chez quatorze mille individus dans l’autre [9]. Et le message qui ressort de ces résultats, conformément au titre de cette chronique, c’est l’extrême abondance des variations rares dans notre ADN : au moins un variant toutes les vingt bases lorsqu’on les totalise sur 14 000 échantillons. Voyons de plus près les données obtenues par ces deux équipes, avant de décrire leur interprétation. Le premier projet [8], vraiment de grande ampleur, a lu en tout 63 térabases (63 000 gibabases) d’ADN. En fait, l’exome entier, totalisant 22 mégabases, a été séquencé chez 2 440 personnes à une redondance moyenne de 111. Au total, plus de 500 000 variants sont repérés au sein des 15 600 gènes examinés dans cet échantillon de population. Un point de terminologie : les variants sont désignés, dans cet article [8] comme dans celui de Nelson et al. [9], par l’acronyme SNV (pour single nucleotide variant), le terme de snip ou SNP (single nucleotide polymorphism) étant réservé à ceux dont l’allèle mineur a une fréquence supérieure à 5 %. Les snip (ou SNP) sont donc aussi des SNV, l’inverse n’étant pas nécessairement vrai. Le deuxième article [9] s’est limité à 202 gènes considérés comme des cibles de médicaments, mais les a lus chez plus de 14 000 individus à une redondance moyenne de 27. Dans les deux cas, la qualité des données est évidemment essentielle : il ne s’agit pas de prendre des erreurs de séquence pour des SNV ! Les auteurs présentent sur ce point des vérifications assez convaincantes qui donnent à penser que la très grande majorité des variations observées est bien réelle. Le premier projet répertorie donc plus de 500 000 SNV, dont la plupart sont rares (fréquence de l’allèle mineur inférieure à 0,5 %). En fait, plus de la moitié de ceux-ci ne sont vus qu’une seule fois parmi les presque 2 500 personnes étudiées. Et la majorité de ces SNV sont non synonymes, altèrent la protéine codée par le gène, aboutissant souvent (d’après les méthodes de prédiction appliquées) à son inactivation. En d’autres termes, chaque personne porte entre 30 et 100 substitutions aboutissant à une perte de fonction, dont une vingtaine à l’état homozygote [10]. Les données ciblées sur 202 gènes [9] vont dans le même sens, découvrant un très grand nombre de mutations rares dont beaucoup altèrent la structure et vraisemblablement la fonction de la protéine correspondante. Notons que, logiquement, plus on séquence, plus on trouve de variations : c’est ce que montre la
Figure 1
qui indique le nombre total de SNV trouvé par base de l’ADN en fonction du nombre de séquences effectuées. On voit que l’effectif des snip (SNP ou SNV fréquents) plafonne rapidement, ce qui est logique : puisque l’allèle mineur est présent à une fréquence d’au moins 5 %, on doit tous les répertorier en lisant une ou deux centaines de séquences. Par contre le nombre de SNV rares augmente linéairement avec le nombre de séquences lues. Selon l’article de Nelson et al. [9], qui porte sur le plus grand nombre de personnes, en lisant un million d’ADN on trouverait un SNV tous les deux nucléotides.
 | Figure 1.
Nombre de variants fréquents et rares dans l’exome humain. Le nombre total de variants fréquents (A) et rares (B) trouvés par séquençage des exomes est exprimé par nucléotide d’ADN et représenté en fonction du nombre d’échantillons séquencés (chacun provenant d’un individu différent). On voit que le total des variants fréquents (allèle mineur à plus de 5 %) plafonne très vite, alors que celui des variants rares (allèle mineur à moins de 0,5 %) continue d’augmenter (graphique redessiné à partir de la figure 12 du supplément de l’article de Tennessen et al. [ 8]. Le trait rouge indique la médiane des valeurs observées). |
Le premier article, qui a séparé les données obtenues selon l’ascendance (européenne ou afro-américaine) des personnes concernées2, indique aussi que les mutations rares diffèrent le plus souvent selon les populations - ce qui est logique dans la mesure où elles sont d’apparition récente (voir plus loin). |
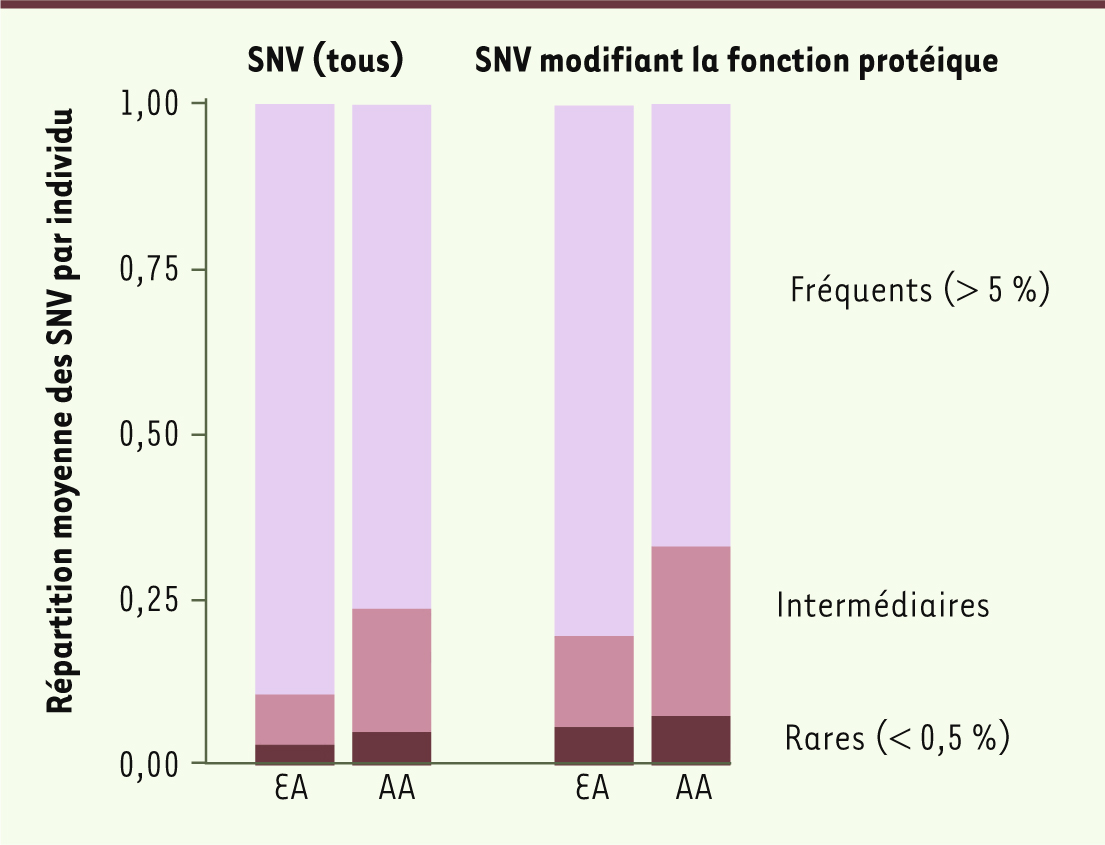
Une nouvelle vision de notre diversité génétique Les résultats que présentent ces deux articles ne sont pas totalement inattendus - mais ces données solides et portant sur un nombre élevé d’individus n’en présentent pas moins un grand intérêt. Elles ne remettent pas en cause la grande homogénéité de notre espèce par rapport à pratiquement toutes les autres, témoignage de notre origine récente [5], mais nous donnent une vision bien plus précise de l’état actuel de notre population. Levons tout de suite un malentendu possible : ces mutations rares sont abondantes au niveau de la population - mais relativement peu nombreuses chez chaque individu, dont le génotype est dominé par les snip, comme le montre la
Figure 2
. Notons aussi que le nombre de variations observées dans ces travaux ne remet pas en cause la valeur précédemment admise du taux de mutations dans l’ADN humain, qui reste estimé à environ 10- 8 par nucléotide et par génération.
 | Figure 2.
Répartition de SNV trouvés par individu. Les variations sont différenciées en rares (rouge grenat), fréquentes (mauve) et intermédiaires (rose), et distinguées selon l’ascendance des personnes étudiées (EA : Européens-Américains, AA : Afro-Américains). À gauche, l’ensemble des SNV, à droite les SNV induisant une altération significative de la protéine concernée. On constate que la proportion des variations de fréquence intermédiaire est plus importante chez les Afro-Américains, ce qui correspond à la plus grande ancienneté de cette population (redessiné à partir de la figure 4C de [ 8]). |
La surprise vient plutôt de la forte proportion de mutations « significatives », celles qui non seulement modifient la séquence de la protéine codée par le gène en cause mais, d’après les différentes méthodes de prédiction employées, aboutissent à son inactivation : cela concerne environ 20 % des SNV, et une vingtaine de ceux-ci sont présents à l’état homozygote chez chaque individu. |
Une espèce vraiment très singulière La clé de ces très nombreuses variations rares, de surcroît souvent délétères, est à chercher dans notre histoire. Contrairement aux autres espèces, la nôtre a connu une expansion démographique à la fois considérable et très récente : quelques dizaines de milliers d’individus il y a 100 000 ans, quelques millions il y a 10 000 ans, un milliard en 1800 et bientôt 10 milliards. Les auteurs, et notamment Tennessen et al. [8], ont pu estimer d’après leurs résultats la date de début de l’expansion accélérée à – 5000 ans. Du coup la sélection (sélection négative que les auteurs désignent souvent par purifying selection) n’a guère eu le temps d’agir et d’éliminer les mutations apparues au sein de cette population en augmentation très rapide, même lorsqu’elles ont un effet néfaste. Les polymorphismes fréquents, les snip, sont généralement plus anciens et leurs effets délétères sont faibles, comme le montrent les résultats des balayages du génome, avec des risques relatifs liés au « mauvais » allèle qui dépassent rarement 1,2 ou 1,3 [2]. L’héritabilité manquante réside donc bien, en grande partie tout au moins, dans les variants rares. Mais il ne va pas être facile de la pister. Tout d’abord, le fait que de nombreuses personnes portent des mutations apparemment délétères à l’état homozygote sans présenter de pathologie visible montre bien que les redondances fonctionnelles au sein de notre génome sont nombreuses : la prédiction du phénotype à partir du génotype n’est vraiment pas de tout repos. D’autre part, ces variants rares sont généralement différents selon les populations3, contrairement aux snip : c’est logique si l’on considère qu’ils sont apparus récemment, mais cela signifie que les nouvelles études d’association génétique devront être conduites à l’intérieur de populations très bien définies. Et enfin, pour des raisons statistiques tenant à la rareté même de ces variations, la capacité d’études GWAS - fondées sur la séquence - à détecter des associations est faible, même si l’on suppose que l’allèle rare et délétère induit un risque relatif de 5, ce qui est très élevé. En tout cas, ces données très riches montrent combien Cavalli-Sforza et ses collaborateurs avaient raison de militer, dès le début des années 1990, en faveur d’un programme destiné à répertorier la diversité génétique humaine, le human genome diversity project [
12]. Très critiqué pour ses possibles implications commerciales, suspecté de pouvoir alimenter un nouveau « racisme scientifique », et vu avec méfiance par beaucoup des populations qu’il se proposait d’étudier, ce projet n’a guère été financé et n’a pas connu l’ampleur souhaitée par son promoteur. Ce n’est peut-être que moindre mal : la technologie a tellement évolué dans l’intervalle que ces études peuvent aujourd’hui être menées à un niveau de détail inimaginable dans la décennie 1990. Les résultats des deux articles présentés ici montrent, à l’évidence, tout l’intérêt de tels travaux. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Jordan
B.
Le boom des séquenceurs nouvelle génération . Med Sci (Paris).
2010; ; 26 : :325.–327. 2.
Jordan
B.
À la recherche de l’héritabilité perdue . Med Sci (Paris).
2010; ; 26 : :541.–543. 3.
Jordan
B.
On a retrouvé l’héritabilité perdue… . Med Sci (Paris).
2012; ; 28 : :721.. 4.
Jordan
B.
Génotypage tous azimuts . Med Sci (Paris).
2007; ; 23 : :772.. 5.
Jordan
B.
L’humanité au pluriel. La génétique et la question des races. , Paris: : Seuil; , 2008
6.
Jordan
B.
Les tests génétiques grand public en « caméra cachée » . Med Sci (Paris).
2011; ; 27 : :103.–106. 7.
Reich
DE
,
Lander
ES.
On the allelic spectrum of human disease . Trends Genet.
2001; ; 17 : :502.–510. 8.
Tennessen
JA
,
Bigham
AW
,
O’Connor
TD
, et al.
Evolution and functional impact of rare coding variation from deep sequencing of human exomes . Science.
2012; ; 337 : :64.–69. 9.
Nelson
MR
,
Wegmann
D
,
Ehm
MG
, et al.
An abundance of rare functional variants in 202 drug target genes sequenced in 14, 002 people . Science.
2012; ; 337 : :100.–104. 10.
Casals
F
,
Bertranpetit
J.
Human genetic variation, shared and private . Science.
2012; ; 337 : :39.–40. 11.
Jordan
B.
Du bon usage des exomes . Med Sci (Paris).
2010; ; 26 : :1111.–1114. 12.
Cavalli-Sforza
LL.
The Human Genome Diversity Project: past, present and future . Nat Rev Genet.
2005; ; 6 : :333.–340. |