| |
| Med Sci (Paris). 2012 November; 28(11): 926–928. Published online 2012 November 12. doi: 10.1051/medsci/20122811008.Structure d’une nanomachine bactérienne
L’aiguille du système de sécrétion de type 3 Antoine Loquet,1* Birgit Habenstein,1 Jean-Philippe Demers,1 Stefan Becker,1 and Adam Lange1 1Max Planck Institute for biophysical chemistry, Am Fassberg 11, 37077Göttingen, Allemagne MeSH keywords: Animaux, Protéines bactériennes, composition chimique, ultrastructure, Systèmes bactériens de sécrétion, physiologie, Toxines bactériennes, métabolisme, Membrane cellulaire, Cryomicroscopie électronique, Cristallographie aux rayons X, Endotoxines, Cellules eucaryotes, Bactéries à Gram négatif, Modèles moléculaires, Résonance magnétique nucléaire biomoléculaire, méthodes, Conformation des protéines, Sous-unités de protéines, Salmonella typhimurium, Relation structure-activité |
Les bactéries utilisent des mécanismes d’infection extrêmement sophistiqués afin d’attaquer les cellules hôtes. L’une des armes les plus dévastatrices des bactéries à gram négatif est le système de sécrétion de type 3 (T3SS), une nanomachine qui injecte les toxines dans la cellule eucaryote au moyen d’une seringue moléculaire, appelée « l’aiguille du T3SS ». Notre équipe a développé de nouvelles méthodes utilisant la résonance magnétique nucléaire (RMN) du solide pour étudier ce type de nanomachines. Ces travaux, publiés dans la revue Nature [
1], ont dévoilé la première structure à résolution atomique d’une aiguille du T3SS, celle de la bactérie Salmonella typhimurium, notamment impliquée dans les fièvres typhoïdes et les gastroentérites. |
L’arme fatale des bactéries : le système de sécrétion de type 3 (T3SS) De nombreuses bactéries pathogènes utilisent le T3SS afin d’infecter les cellules eucaryotes, ces cellules hôtes pouvant être humaines, animales ou végétales. Ces espèces bactériennes pathogènes comprennent Salmonella, Shigella, Yersinia et Pseudomonas, ainsi que les souches d’Escherichia, telles que l’enterohemorrhagic E. coli (EHEC) qui a causé d’importants problèmes de santé publique en Europe en 2011. L’action du T3SS est associée à des maladies variées incluant les intoxications par l’eau et la nourriture, la peste, le choléra, la fièvre typhoïde et les pneumopathies. Le T3SS est une nanomachine comprenant deux parties principales [
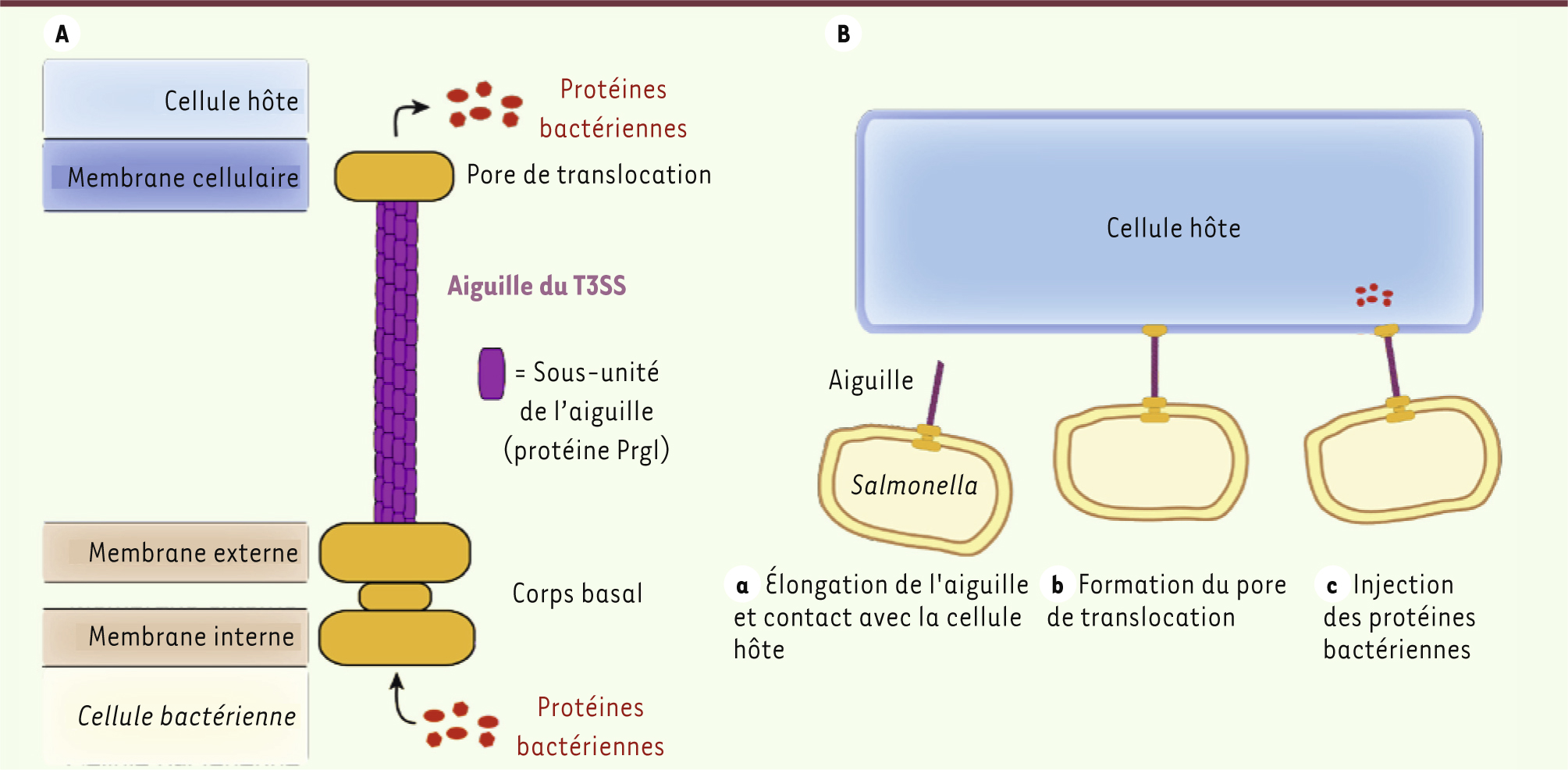
2] (Figure 1A). Le corps basal s’étend du cytoplasme de la bactérie et traverse ses deux membranes, formant un pore d’export. L’aiguille est la partie extracellulaire du T3SS : il s’agit d’un assemblage supramoléculaire de 100 à 200 exemplaires d’une même sous-unité protéique, par exemple PrgI pour Salmonella typhimurium. L’aiguille s’allonge au fur et à mesure que des sous-unités protéiques sont sécrétées, pour finalement former un tube creux d’environ 50-100 nanomètres de longueur. Une fois le contact avec la cellule hôte établi, un pore de translocation est formé au bout de l’aiguille et ancré dans la membrane cellulaire afin d’injecter les protéines bactériennes, qui seront transportées de la bactérie vers la cellule à travers l’intérieur de l’aiguille (Figure 1B).
 | Figure 1.
Organisation structurale et rôle de l’aiguille du T3SS. A. L’aiguille est formée d’un tube creux d’une longueur d’environ 50-100 nanomètres assemblé par 100-200 sous-unités protéiques (PrgI pour S. typhimurium). B. L’aiguille joue un rôle primordial durant l’infection en injectant les protéines bactériennes dans la cellule hôte. |
|
L’aiguille du T3SS : un véritable défi technologique Bien que la structure de la sous-unité monomérique ait pu être obtenue par cristallographie des rayons X et par la RMN en solution, la détermination de la structure de l’aiguille du T3SS constitue un véritable défi technologique. En effet, ces techniques biophysiques conventionnelles ne sont pas applicables à ce type d’assemblage. En raison de son architecture tubulaire, l’aiguille demeure résistante à toute tentative de cristallisation. L’aiguille est également une entité insoluble de grande taille moléculaire qui ne peut donc pas être étudiée par la RMN en solution. D’importants progrès ont été réalisés en cryomicroscopie électronique [
3], une technique permettant d’obtenir l’enveloppe 3D de ce type d’assemblages. Un modèle structural de l’aiguille a été récemment publié [
4] où la structure de la sous-unité monomérique obtenue par cristallographie a été insérée par docking moléculaire dans l’enveloppe 3D obtenue par cryomicroscopie. Ce type de reconstruction n’est cependant pas exempt d’erreurs, car (1) l’enveloppe est obtenue à une résolution modérée, environ 7 Å, insuffisante pour distinguer chaque acide aminé individuellement, et (2) cette approche ne tient pas compte des possibles changements de conformation de la sous-unité protéique entre la forme monomérique et la forme assemblée en aiguille. |
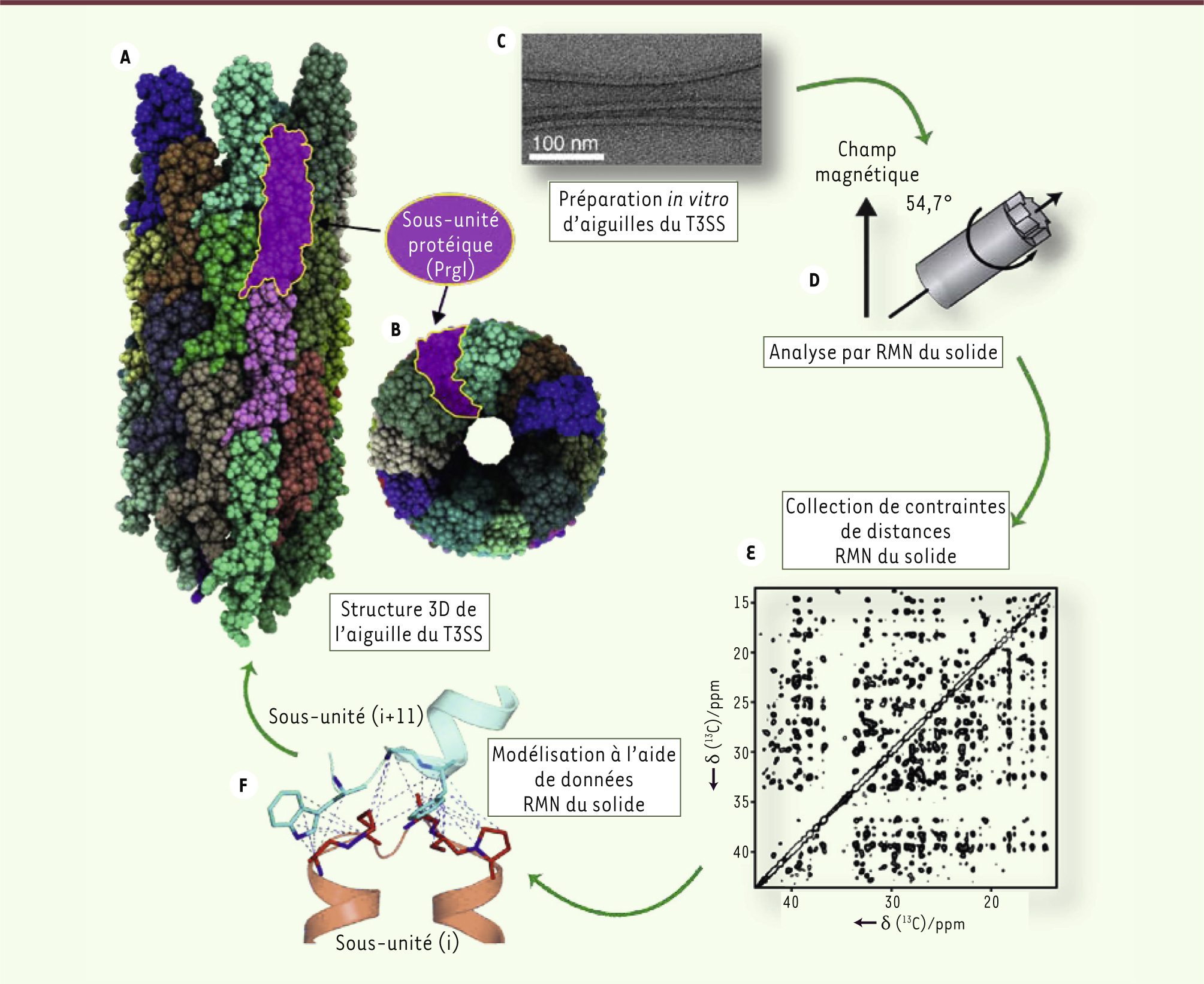
Une nouvelle approche méthodologique basée sur la RMN du solide Pour arriver à la structure atomique de l’aiguille, notre équipe a utilisé une approche basée sur la RMN du solide. Cette technique spectroscopique permet d’étudier la structure de protéines solides [
5] telles que les microcristaux de protéines [
6], les fibrilles amyloïdes [
7], les protéines membranaires [
8] ou les protéines de grande taille [
9]. L’échantillon est produit avec un marquage isotopique au carbone-13 (13C) et azote-15 (15N), puis introduit dans un rotor (Figure 2) tournant à plusieurs milliers de tours par seconde avec un angle particulier (54,7°) appelé « l’angle magique ». Cette rotation rapide compense l’absence de réorientation moléculaire en phase solide et permet d’observer à haute résolution un signal par atome de carbone ou d’azote. Notre équipe a développé une méthode exploitant la dilution des isotopes de 13C [
10] qui améliore la résolution de ces signaux. Ainsi, les spectres de l’aiguille du T3SS constituent, à l’heure actuelle, la référence en termes de qualité spectroscopique pour la RMN du solide. Dans une méthode complémentaire [7], des sous-unités sélectivement marquées au 13C ont été mélangées afin de déterminer l’interface supramoléculaire entre deux sous-unités. Nous avons combiné ces deux méthodes dans l’étude récemment publiée dans le journal Nature [1] pour déterminer la structure de l’aiguille du T3SS à une résolution jamais atteinte jusqu’alors.
 | Figure 2.
Une nouvelle approche méthodologique pour élucider la structure 3D de l’aiguille du T3SS. La structure de l’aiguille de S. typhimurium est présentée avec une vue prise de face (A) ou du dessus (B), cette dernière vue permettant d’observer le pore interne de l’aiguille. C. Image en microscopique électronique des aiguilles produites in vitro. D. Rotor de RMN du solide. E. Spectre de RMN du solide. Ppm : partie par millions. F. Interface supramoléculaire entre deux sous-unités PrgI. |
|
La structure de l’aiguille du T3SS de Salmonella typhimurium Des aiguilles de S. typhimurium ont été produites in vitro par polymérisation de la sous-unité protéique, puis ont été étudiées par RMN du solide. La structure RMN obtenue a révélé un tube de 80 Å de diamètre extérieur, comprenant un pore de 25 Å de diamètre. L’aiguille est formée par l’assemblage des sous-unités dans une architecture hélicoïdale, où 11 sous-unités sont nécessaires pour faire un tour complet d’hélice. L’extrémité amino-terminale de PrgI forme un segment rigide de conformation étendue participant à l’interface supramoléculaire. Cette découverte est en contradiction avec le modèle de l’aiguille discuté précédemment [4], où l’extrémité amino-terminale avait été modélisée en hélice α. Dans notre étude, l’architecture de l’aiguille diffère également au niveau de l’extrémité amino-terminale qui se situe sur la face extérieure de l’aiguille et de l’extrémité carboxy-terminale sur la face intérieure, parant ainsi le pore interne, alors que le modèle basé sur la cryomicroscopie présentait une configuration inverse (extrémité carboxy-terminale sur la face externe). Afin de confirmer notre structure RMN, nous avons attaché un tag moléculaire à l’extrémité amino-terminale que nous avons localisé grâce à la microscopie immunoélectronique. Nous avons ainsi confirmé la présence de l’extrémité amino-terminale sur la face externe. Les acides aminés les plus conservés entre les sous-unités des bactéries Salmonella typhimurium, Shigella flexneri, Yersinia pestis et Escherichia coli sont principalement situés dans l’extrémité carboxy-terminale, formant le pore de sécrétion, ce qui explique le rôle primordial de cette extrémité dans le transport des toxines. Les moins conservés se situent à la surface extérieure de l’aiguille et sont donc exposés au milieu extracellulaire. Leur diversité peut constituer une stratégie bactérienne afin d’échapper à la reconnaissance spécifique par les défenses de la cellule hôte. Disposant de cette structure, notre équipe a maintenant pour objectif de découvrir des molécules potentiellement capables d’intervenir dans le processus d’assemblage de l’aiguille, afin de développer une stratégie pour contrecarrer son action durant l’infection. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Loquet
A
,
Sgourakis
NG
,
Gupta
R
, et al.
Atomic model of the type III secretion system needle . Nature.
2012; ; 486 : :276.–279. 2.
Cornelis
GR.
The type III secretion injectisome . Nat Rev Microbiol.
2006; ; 4 : :811.–825. 3.
Fujii
T
,
Iwane
AH
,
Yanagida
T
,
Namba
K.
Direct visualization of secondary structures of F-actin by electron cryomicroscopy . Nature.
2010; ; 467 : :724.–728. 4.
Fujii
T
,
Cheung
M
,
Blanco
A
, et al.
Structure of a type III secretion needle at 7-A resolution provides insights into its assembly and signaling mechanisms . Proc Natl Acad Sci USA.
2012; ; 109 : :4461.–4466. 5.
Loquet
A
,
Gardiennet
C
,
Bockmann
A.
Protein 3D structure determination by high-resolution solid-state NMR . CR Chimie.
2010; ; 13 : :423.–430. 6.
Loquet
A
,
Bardiaux
B
,
Gardiennet
C
, et al.
3D structure determination of the Crh protein from highly ambiguous solid-state NMR restraints . J Am Chem Soc.
2008; ; 130 : :3579.–3589. 7.
Loquet
A
,
Giller
K
,
Becker
S
,
Lange
A.
Supramolecular interactions probed by 13C–13C solid-state NMR spectroscopy . J Am Chem Soc.
2010; ; 132 : :15164.–15166. 8.
Lange
A
,
Giller
K
,
Hornig
S
, et al.
Toxin-induced conformational changes in a potassium channel revealed by solid-state NMR . Nature.
2006; ; 440 : :959.–962. 9.
Habenstein
B
,
Wasmer
C
,
Bousset
L
, et al.
Extensive de novo solid-state NMR assignments of the 33 kDa C-terminal domain of the Ure2 prion . J Biomol NMR.
2011; ; 51 : :235.–243. 10.
Loquet
A
,
Lv
G
,
Giller
K
, et al.
13C Spin dilution for simplified and complete solid-state NMR resonance assignment of insoluble biological assemblies . J Am Chem Soc.
2011; ; 133 : :4727.–4735. |