| |
| Med Sci (Paris). 2012 November; 28(11): 977–982. Published online 2012 November 12. doi: 10.1051/medsci/20122811017.Technologie des lames virtuelles
De la numérisation à la mise en ligne David Ameisen,1* Gilles Le Naour,2** and Christel Daniel3*** 1Université Paris Diderot, Sorbonne Paris-Cité, Institut universitaire d’hématologie, laboratoire de pathologie, groupe hospitalier Saint-Louis Lariboisière Fernand-Widal, Inserm UMR-S 728, 1, avenue Claude Vellefaux, 75010Paris, France 2Faculté de médecine Pierre et Marie Curie, groupe hospitalier Pitié-Salpêtrière, service d’anatomie et de cytologie pathologiques, unité d’imagerie morphologique (UIMAP), 83, boulevard de l’Hôpital, 75013Paris, France 3DIH-HUPO-APHP, Université Paris Descartes, Inserm UMRS 872 eq 20, 15, rue de l’école de médecine, 75006Paris, France |
Les activités de soins sont aujourd’hui engagées dans une nouvelle étape de rationalisation qui implique non seulement l’informatisation généralisée des données, mais aussi la mise en réseau de ces informations. Dans ce contexte, la technologie des lames virtuelles (LV) est passée en 15 ans du stade de technique innovante à celui d’outil pédagogique, clinique et de recherche incontournable. L’anatomie pathologique est l’étude au microscope, après coloration par différentes techniques, des prélèvements de cellules ou de tissus fixés sur lame de verre [
32] (→). Depuis l’invention du microscope à la fin du xvi
e siècle par le hollandais Zacharias Janssen, au moins deux obstacles techniques majeurs ont limité les possibilités de collaboration entre observateurs. Tout d’abord, l’observation d’une lame au microscope ne pouvait être effectuée que par une seule personne à la fois. Par ailleurs, la gestion des lames a toujours été confrontée au rique de bris et aux difficultés d’archivage.
(→) Voir m/s n° 6-7, juin-juillet 2011, page 651
Plusieurs techniques ont été inventées pour apporter des réponses à ces problèmes : le microscope multi-têtes puis, plus récemment, l’envoi de prises de vues des lames par internet ou la retransmission par voie hertzienne des vidéos de déplacement de champs sur la lame. Ces méthodes ont néanmoins une limite commune : une seule personne contrôle le microscope, elle n’examine qu’une partie du champ, et transmet ensuite les images aux autres observateurs qui ne peuvent donc analyser que ce que la première personne a choisi de regarder. Les LV permettent précisément de dépasser ces limites. C’est en 1997 à Baltimore qu’a été conçu et utilisé le premier microscope virtuel, grâce à une collaboration entre le département d’informatique de l’université du Maryland et le service de pathologie de l’hôpital John Hopkins [
1]. Le concept de microscopie virtuelle comporte plusieurs aspects, de l’acquisition des images jusqu’à leur visualisation en réseau depuis un ordinateur ou un smartphone [
2]. |
La numérisation Les différents systèmes de numérisation Tous les systèmes de microscopie virtuelle sont composés d’un système d’acquisition (numérisation) de haute résolution, d’un système de stockage et d’un système de diffusion [ 2,
3] (Figure 1). Le système d’acquisition est composé d’une platine motorisée en X, Y et Z, d’un objectif de microscope et d’une caméra numérique haute résolution contrôlée par un logiciel d’acquisition.
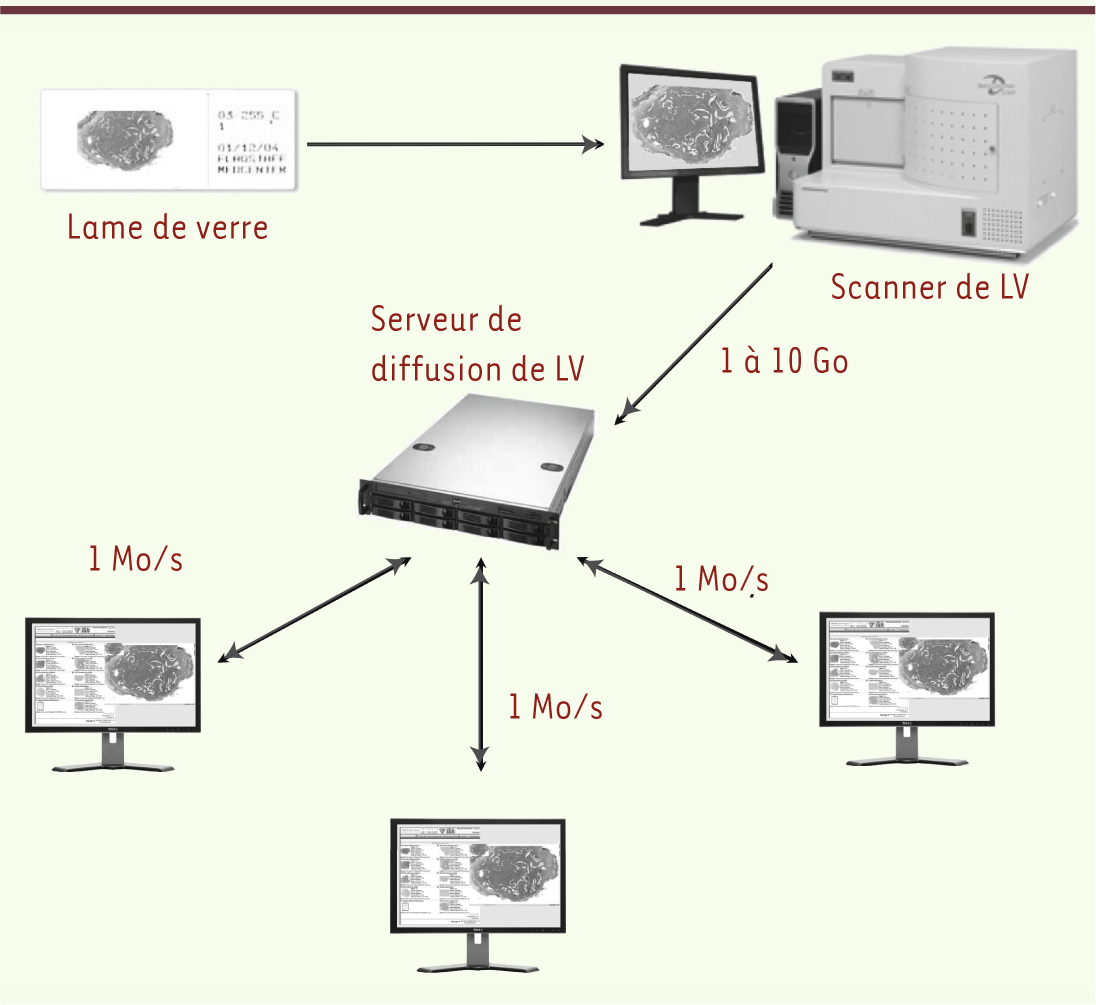
 | Figure 1.
Acquisition et visualisation en ligne des lames virtuelles. La lame de verre est numérisée par un système d’acquisition (ici un scanner). La LV produite est ensuite transférée sur un serveur de diffusion auquel se connectent à distance les utilisateurs. Les problématiques de stockage et de bande passante sont au cœur de la microscopie virtuelle compte tenu du volume de ces images. Si la totalité du fichier (jusqu’à 10 Go) est transférée sur le serveur de diffusion, la bande passante ainsi que l’ensemble des matériels nécessaires à sa visualisation sont minimaux (1 Mo/s). En effet, seules sont téléchargées les régions de l’image accédées par l’utilisateur et affichées sur son écran. |
Historiquement, le premier matériel d’acquisition de LV a été le microscope motorisé. Il conserve les mêmes fonctionnalités qu’un microscope standard, mais il est connecté à une caméra et est contrôlé par ordinateur. La platine sur laquelle est placée la lame se déplace sous l’objectif et une prise de vue est faite par la caméra à chacun des déplacements. Le microscope motorisé offre une grande souplesse d’utilisation, permettant à l’utilisateur de modifier les objectifs, de faire varier la luminosité ou de repositionner la préparation comme avec un microscope classique. Il permet, en outre, d’utiliser facilement un objectif à immersion (grossissement jusqu’à 100x). En revanche, parce qu’il nécessite une recalibration régulière, il est plus adapté à des volumes modérés de lames à numériser. L’autre grand type de matériel d’acquisition, le scanner, est constitué de composants similaires à ceux des microscopes motorisés, mais ont été supprimés les oculaires ainsi que les outils de contrôle manuel de positionnement et de mise au point (Figure 1). Il est pourvu d’une seule ouverture pouvant recevoir jusqu’à plusieurs centaines de lames. Il utilise généralement un objectif 20x avec une grande ouverture numérique permettant une très bonne résolution des images finales. Un doubleur optique peut augmenter encore cette résolution, proche de celle que permet un objectif 40x. Il est également possible d’installer un véritable objectif 40x, au prix toutefois d’un allongement de la durée de numérisation. Destiné à la seule numérisation des LV, fiable et robuste, il est particulièrement adapté aux utilisateurs ayant des volumes de lames importants et des problématiques de numérisation dédiées et standardisées. Principes de la numérisation et formats des images générées Quel que soit le système utilisé, la numérisation commence par une prise de vue de l’ensemble de la lame à très faible grossissement, permettant de sélectionner tout ou une partie de la lame à numériser. Les paramètres numériques de l’image, tels que le contraste ou la luminosité, peuvent alors être modulés. L’échantillon étant nécessairement d’épaisseur hétérogène, le système définira alors la cartographie des variations de mise au point de l’objectif. La numérisation proprement dite est ensuite réalisée au grossissement maximal souhaité. Le microscope motorisé prend une série de clichés de champs carrés ou rectangulaires (tuiles) en se déplaçant de zone en zone contiguës, d’un coin de la lame au coin opposé (Figure 2A). Le scanner quant à lui numérise de longues bandes rectangulaires contiguës d’un coin de la lame ou de la région d’intérêt sélectionnée au coin opposé (Figure 2B).
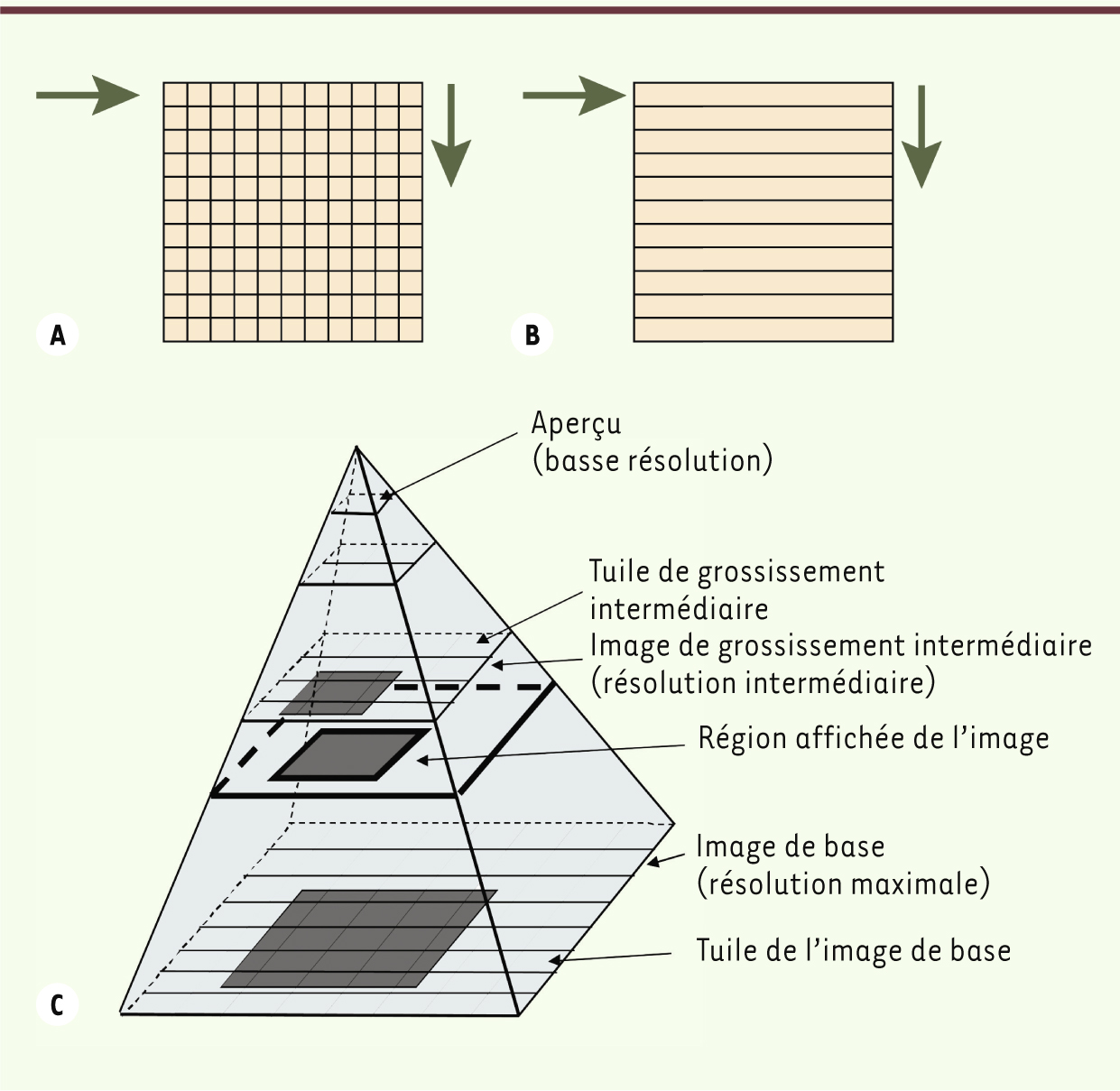
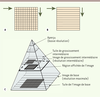 | Figure 2.
Numérisation et visualisation d’une image pyramidale. A. La numérisation d’une région d’intérêt par un microscope motorisé s’effectue par l’acquisition de tuiles carrées ou rectangulaires en parcourant la lame, avec des déplacements de zones en zones contiguës, d’un coin de la lame au coin opposé. B. La numérisation d’une région d’intérêt par un scanner s’effectue par une acquisition de longues bandes rectangulaires contiguës d’un coin de la lame ou de la région sélectionnée au coin opposé. C. Visualisation : lorsque l’utilisateur parcourt la lame à partir de l’aperçu de son ensemble, il peut accéder aux tuiles des différents grossissements, des plus faibles aux plus importants, jusqu’à la résolution maximale (image de base). Si l’image au niveau de grossissement inférieur souhaité n’a pas été numérisée lors de l’acquisition, le logiciel de visualisation génère, à partir de l’image de base, une image de la région à afficher à ce grossissement. Il est alors possible de faire varier le niveau de grossissement de manière continue entre la vue d’ensemble au faible grossissement et le grossissement maximum. |
Ces séries de fichiers sont assemblées en une image de dimension maximale. Le logiciel génère ensuite les grossissements intermédiaires à partir de l’image la plus détaillée grâce à des algorithmes de redimensionnement. Cette image produite est dite pyramidale (Figure 2C), c’est-à-dire composée de plusieurs fichiers ou régions d’intérêt, chacun étant désigné par des coordonnées (X, Y, Z, G) désignant à la fois l’emplacement dans l’image (en largeur, longueur et hauteur ; X, Y et Z) et le grossissement (G). Toutefois, le système peut également générer une seule image « plate » de format usuel (bmp, tiff, jpeg, etc.) en deux dimensions en assemblant les tuiles acquises pour un grossissement donné [2]. La durée de la numérisation et la taille du fichier généré sont proportionnelles à la surface à numériser, au nombre de plans focaux et au grossissement choisi : à titre d’exemple, une lame qui comporte un échantillon de 20 mm × 15 mm sera numérisée avec un objectif 40x en 5 à 10 min. En fonction de la compression choisie, elle occupera entre 1 et 10 gigaoctets (Go) pour une définition de l’ordre du gigapixels (109 pixels). La visualisation Principes généraux Chaque système d’acquisition de LV s’accompagne de logiciels de visualisation ( viewer) locaux et distants qui permettent de naviguer dans la lame à différents grossissements [ 2]. Le viewer peut être installé localement sur l’ordinateur de l’utilisateur et permettre de regarder une LV elle aussi stockée localement. Un viewer installé localement peut également se connecter à un serveur distant qui lui transmet des flux d’images à travers le réseau. Enfin, la visualisation peut être faite en se connectant à une application web avec un navigateur internet usuel. Il y a trois priorités pour les logiciels de visualisation : la qualité des images, la rapidité de leur affichage [
4] et la compatibilité avec les ordinateurs ou périphériques utilisés. Une visualisation sans perte d’information par rapport à l’image acquise est donc essentielle. En outre, pour une navigation fluide dans le cas d’une visualisation à distance, seule est diffusée à l’écran la partie de la lame que l’observateur souhaite afficher. La bande passante requise pour diffuser les lames dépend ainsi, non pas du poids (en octets) de la lame, mais de la résolution de la fenêtre d’affichage à l’écran et du nombre d’utilisateurs connectés au système. La bande passante requise pour visualiser les LV s’accommode d’une connexion ADSL (asymmetric digital subscriber line) usuelle (10 mégabits par seconde [Mbps]). Enfin, la plupart des viewers sont compatibles avec la majorité des systèmes d’exploitation existants. Fonctionnalités avancées Dès lors qu’il s’agit de gérer un grand nombre d’images, les viewers sont intégrés à des plateformes de gestion où les utilisateurs peuvent grouper les images par dossiers et par mots clés. Ils peuvent alors les partager de manière publique ou avec d’autres utilisateurs préalablement enregistrés. Ceux-ci pourront, selon les droits dont ils disposeront, les visualiser (lecture seule) ou collaborer en ajoutant annotations, commentaires ou mots clés (lecture/écriture) [
5]. Par ailleurs, de nombreux outils sont intégrés aux viewers pour augmenter l’intérêt pédagogique ou diagnostique de la lame. Parmi ceux-ci, on peut évoquer la possibilité d’adjoindre des métadonnées, telles que des annotations ou des commentaires. Il est également possible de dépasser les limites du grossissement optique du microscope ou du scanner avec un zoom numérique. Un système multi-fenêtres permet de voir plusieurs lames simultanément : leur déplacement synchronisé est très utile pour observer plusieurs colorations différentes sur des champs identiques ou également pour l’analyse de TMA (tissue microarray) [
6]. Outre les déplacements horizontaux et verticaux, il est possible d’effectuer des rotations ou d’afficher différents plans focaux sur une zone donnée. On peut également citer les projets de visualisation ultra-rapide des LV sur écran géant [
7], ou en trois dimensions grâce à la numérisation de centaines de coupes tissulaires successives pour explorer l’image produite dans son épaisseur. Ce type d’analyse permet de rendre compte du volume des structures analysées par le pathologiste, et non simplement de leur surface [
8]. Enfin, ces fonctionnalités peuvent être complétées par des logiciels d’aide au diagnostic grâce à des outils d’analyse d’image [
9,
10], tels que le comptage automatique des noyaux de cellules présents sur la lame ou la mesure des surfaces de tissus. |
Enjeux autour des lames virtuelles Les défis posés par le stockage et la diffusion des lames virtuelles Les projets de télépathologie concernent avant tout les activités d’expertise ou les examens urgents. La numérisation de toutes les lames de l’activité diagnostique d’un laboratoire pose encore d’importants problèmes de faisabilité surtout liés à la volumétrie importante. Le stockage interne est actuellement limité à une dizaine de teraoctets (To) par ordinateur. Si le coût du stockage local interne ou externe reste modéré (environ 80 €/To), le stockage en réseau quant à lui coûte entre 75 € et 375 €/To/mois. Par ailleurs, la plupart des serveurs de diffusion connectés sur un réseau universitaire ont une bande passante souvent inférieure à 100 Mbps. Dans un contexte hospitalier, la bande passante est souvent encore plus faible. Ainsi, un seul serveur peut accepter au plus mille utilisateurs connectés au site de gestion des LV, cent utilisateurs visualisant des lames distinctes, et quelques dizaines d’utilisateurs visualisant la même lame. En outre, les directions des systèmes d’information (DSI) hospitaliers et universitaires ont souvent des politiques de sécurité strictes, limitant l’accès à certains programmes, sites, protocoles réseaux (http) et ports réseaux (80). Elles doivent donc toujours être associées à la mise en place de systèmes de LV. L’assurance qualité La qualité des LV n’a cessé de s’améliorer grâce à des innovations régulières dans les domaines des systèmes d’acquisition et de visualisation. Ces évolutions sont mesurées et stimulées lors de compétitions biennales de mesure de performance et de qualité entre industriels [
11]. Le contrôle de la qualité reste toutefois un préalable essentiel à l’utilisation à grande échelle des LV [
12–
16]. Si la qualité d’une image peut s’évaluer grâce à différents paramètres quantifiables tels que son poids, sa taille en pixels, sa luminosité, sa saturation, son contraste, etc., la netteté d’une image est en revanche un paramètre beaucoup plus délicat à quantifier [
17]. Une difficulté supplémentaire pour les LV est la nécessité d’assembler et de pondérer les résultats obtenus pour chacune des régions de l’image, à chacun des grossissements. Différents outils d’évaluation automatique de la qualité sont disponibles ou en développement [
18,
19]. Une évaluation en temps réel permettra d’effectuer une renumérisation automatique de tout ou partie d’une lame avec des réglages plus fins, évitant ainsi une rupture du workflow. Ces contrôles permettront également de détecter des dérèglements de la calibration des appareils. La standardisation L’élaboration de standards d’informatique de santé a pour objectif de faciliter et de généraliser les échanges. Le rôle de l’initiative internationale Integrating the healthcare enterprise (IHE) [
20] est de promouvoir l’utilisation de tels standards [
21,
22]. Grâce à une coopération étroite entre utilisateurs et industriels, son objectif est de développer les échanges et l’intégration entre logiciels de différents fournisseurs en s’appuyant sur des normes et standards internationaux reconnus et opérationnels, tels que Health level 7 (HL7) pour l’échange informatisé de données administratives et cliniques, ou Digital imaging and communications in medicine (DICOM) pour l’archivage et la communication des données dans le domaine de l’imagerie médicale [
23]. L’initiative IHE en anatomie pathologique a débuté en 2005. Elle a permis des adaptations spécifiques du format DICOM pour ajouter les LV à l’ensemble des images déjà gérées par ce format. L’efficacité et la flexibilité des activités de stockage, de gestion, de visualisation et de partage des LV ont été également améliorées grâce à l’élaboration d’un profil d’intégration spécifique, Anatomic pathology workflow (APW), destiné à être utilisé par les constructeurs de scanner pour intégrer des LV aux PACS (Picture archiving and communication system). Enfin, IHE anatomic pathology et HL7 anatomic pathology ont défini ensemble un guide d’implémentation de comptes rendus structurés, au format HL7 Clinical document architecture (CDA) [
24]. Ce format permet notamment d’établir des liens entre les comptes rendus et les images, qu’il s’agisse d’images illustratives de petite taille ou de LV [22,
25,
26]. Les possibilités offertes par la décentralisation des données Grâce au principe du cloud computing [
27,
28], le partage en ligne des LV parvient à mettre à disposition d’un grand nombre d’utilisateurs des ressources provenant de structures diverses et/ou éloignées géographiquement (Figure 3). Les utilisateurs se connectent à un portail général qui leur donne accès de manière indifférenciée aux LV provenant de l’un ou l’autre des serveurs de diffusion connectés. Ils pourront alors les partager, les annoter, etc. directement en ligne. Cette structure sécurisée est évolutive et permet de dépasser les contraintes logicielles, matérielles et de bande passante en augmentant à discrétion le nombre de serveurs connectés.
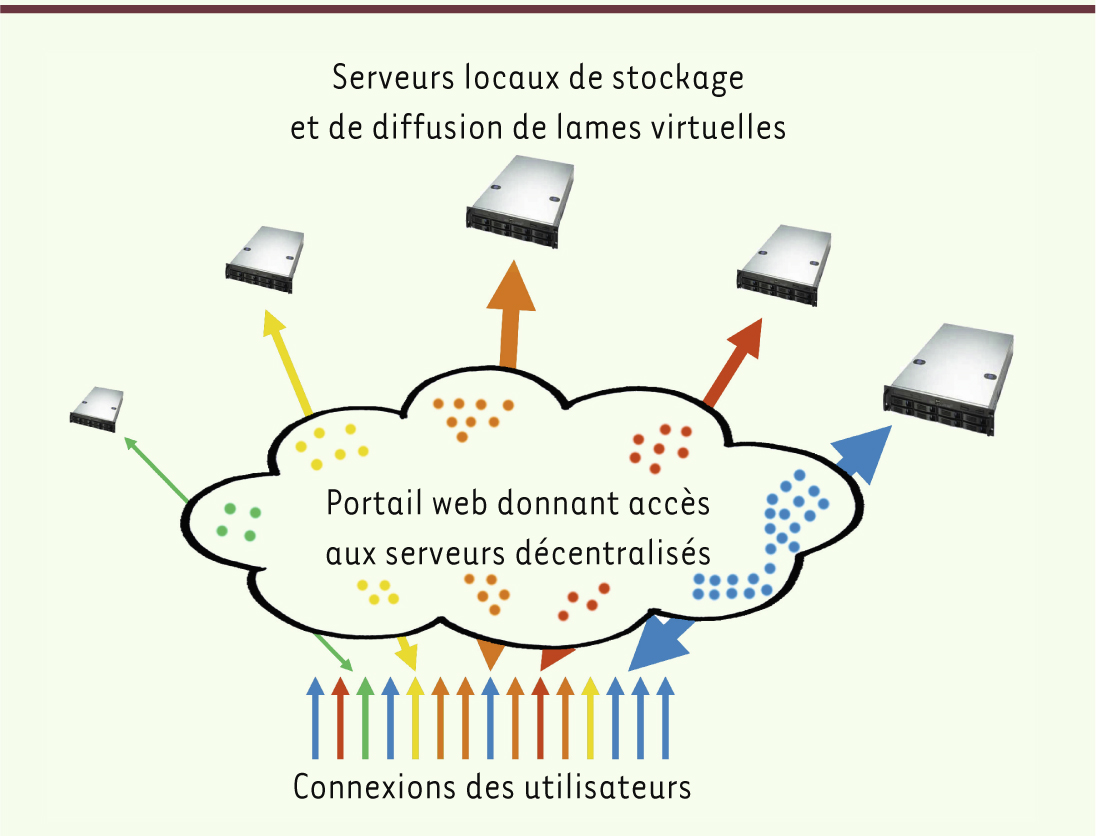
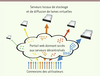 | Figure 3.
Décentralisation et mutualisation des lames virtuelles (LV). Les utilisateurs accèdent à un portail connecté selon le principe du cloud computing, ou nuage informatique, qui donne accès (flèches colorées) de manière indifférenciée aux LV (points colorés) provenant de l’un ou l’autre des serveurs locaux de stockage et de diffusion de LV connectés à ce portail. Cette architecture décentralisée est essentielle pour une utilisation à grande échelle des LV. |
Cette décentralisation des données permet de mutualiser les ressources des différentes structures participantes et, ainsi, de répartir les coûts occasionnés par les prérequis actuels des LV en termes de stockage et de bande passante. Elle permet aussi l’utilisation concomitante de différents systèmes d’acquisition et de visualisation, ainsi que la mutualisation des ressources pour des travaux d’analyse automatisée d’image à grande échelle, tels que l’analyse de la qualité. Il s’agit donc d’une condition essentielle pour penser l’utilisation à grande échelle des LV au sein de structures importantes et, éventuellement, hétérogènes. |
La technologie des LV devient aujourd’hui indispensable dans toutes les dimensions de l’exercice de l’anatomie pathologique. Les défis technologiques du poids des LV, de la complexité et de l’hétérogénéité des formats de ces images, ne constituent plus aujourd’hui un frein à leur utilisation à grande échelle, bien que les problématiques de stockage et de réseau soient encore un défi pour une numérisation systématique des lames. Par ailleurs, la qualité des images obtenues s’améliorera encore grâce à la mise en place de procédures systématiques d’évaluation et de standardisation. Enfin, les progrès récents réalisés par les organismes de standardisation d’informatique de santé dans le domaine de l’anatomie pathologique, sous l’impulsion de l’initiative IHE anatomic pathology, offrent des perspectives de développement du partage et des échanges des LV par les professionnels de santé lors des activités de coordination des soins, mais également de recherche et de formation. Il s’agit désormais de préparer les structures médicales à la prochaine évolution qui s’amorce déjà : la mise en commun des ressources cliniques [
29], pédagogiques [
30] ou de recherches [
31] (→) entre institutions partenaires, dans le cadre de politiques nationales et internationales de coopération et de rapprochement entre laboratoires, universités et hôpitaux. Cette évolution est porteuse de nombreux défis techniques, administratifs et financiers liés aux problématiques de mise en réseau et de mise à l’échelle.
(→) Voir pages 983, 986 et 990 de ce numéro
Dans ce contexte, il convient de souligner les avantages de la décentralisation des données, à savoir un stockage local pour chacun des sites concernés et une diffusion à travers un portail unique ou plusieurs portails interconnectés. Cette décentralisation des données permet en effet de préserver l’autonomie de la création et de la gestion des lames tout en les partageant de manière sécurisée. Enfin, elle permet d’assurer une très grande disponibilité des ressources, malgré la taille importante des fichiers et le nombre croissant d’utilisateurs connectés. De telles infrastructures ne peuvent cependant se concevoir que dans le cadre d’un pilotage à la fois concentré et concerté. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Ferreira
R
,
Moon
B
,
Humphries
J
, et al.
The virtual microscope . Proc AMIA Annu Fall Symp.
1997 ; :449.–453. 2.
Rojo
MG
,
Garcia
GB
,
Mateos
CP
, et al.
Critical comparison of 31 commercially available digital slide systems in pathology . Int J Surg Pathol.
2006; ; 14 : :285.–305. 3.
Saltz
JH.
Digital pathology. The big picture . Hum Pathol.
2000; ; 31 : :779.–780. 4.
Yagi
Y
,
Yoshioka
S
,
Kyusojin
H
, et al.
Ultra high speed whole slide image viewing system . Anal Cell Pathol (Amst).
2012; ; 35 : :65.–73. 5.
Ameisen
D
,
Vergier
B
,
Hauchecorne
O
, et al.
Lames virtuelles en ligne en 2007 : une technologie au service de nombreuses applications en pathologie . Ann Pathol.
2008; ; 28 : :17.–26. 6.
Lahrmann
B
,
Halama
N
,
Westphal
K
, et al.
Robust gridding of TMAs after whole-slide imaging using template matching . Cytometry A.
2010; ; 77 : :1169.–1176. 7.
Treanor
D
,
Jordan-Owers
N
,
Hodrien
J
, et al.
Virtual reality Powerwall versus conventional microscope for viewing pathology slides : an experimental comparison . Histopathology.
2009; ; 55 : :294.–300. 8.
Roberts
N
,
Magee
D
,
Song
Y
, et al.
Toward routine use of 3D histopathology as a research tool . Am J Pathol.
2012; ; 180 : :1835.–1842. 9.
Laurinavicius
A
,
Laurinaviciene
A
,
Dasevicius
D
, et al.
Digital image analysis in pathology : benefits and obligation . Anal Cell Pathol (Amst).
2012; ; 35 : :75.–78. 10.
Huang
CH
,
Veillard
A
,
Roux
L
, et al.
Time-efficient sparse analysis of histopathological whole slide images . Comput Med Imaging Graph.
2011; ; 35 : :579.–591. 12.
Harris
T
,
Leaven
T
,
Heidger
P
, et al.
Comparison of a virtual microscope laboratory to a regular microscope laboratory for teaching histology . Anat Rec.
2001; ; 265 : :10.–14. 13.
Evered
A
,
Dudding
N.
Accuracy and perceptions of virtual microscopy compared with glass slide microscopy in cervical cytology . Cytopathology.
2011; ; 22 : :82.–87. 14.
Harnden
P
,
Coleman
D
,
Moss
S
, et al.
Evaluation of the use of digital images for a national prostate core external quality assurance scheme . Histopathology.
2011; ; 59 : :703.–709. 15.
Walkowski
S
,
Szymas
J.
Quality evaluation of virtual slides using methods based on comparing common image areas . Diagn Pathol.
2011; ; 6 : :S14.. 16.
Ho
J
,
Parwani
AV
,
Jukic
DM
, et al.
Use of whole slide imaging in surgical pathology quality assurance : design and pilot validation studies . Hum Pathol.
2006; ; 37 : :322.–331. 17.
Ameisen
D
,
Bertheau
P.
Méthode de détection et de quantification du flou dans une image numérique . PCT/FR2011/052951 ; brevet n°1060445 déposé à l’Institut national de la propriété industrielle. , le
décembre 13;
2010 . 18.
Ameisen
D
,
Bertheau
P.
Procédé d’analyse de la qualité d’une lame virtuelle d’un objet . FR 1154038 ; brevet déposé à l’Institut national de la propriété industrielle. , le
mai 10;
2011 . 19.
Zerbe
N
,
Hufnagl
P
,
Schlüns
K.
Distributed computing in image analysis using open source frameworks and application to image sharpness assessment of histological whole slide images . Diagn Pathol.
2011; ; 6 : :S16.. 21.
Daniel
C
,
García Rojo
M
,
Bourquard
K
, et al.
Standards to support information systems integration in anatomic pathology . Arch Pathol Lab Med.
2009; ; 133 : :1841.–1849. 22.
Daniel
C
,
Rojo
MG
,
Klossa
J
, et al.
Standardizing the use of whole slide images in digital pathology . Comput Med Imaging Graph.
2011; ; 35 : :496.–505. 23.
Bozec C
Le
,
Henin
D
,
Fabiani
B
, et al.
Refining DICOM for pathology-progress from the IHE and DICOM pathology working groups . Stud Health Technol Inform.
2007; ; 129 : :434.–438. 25.
Daniel
C
,
García Rojo
M
,
Bourquard
K
, et al.
Standards to support information systems integration in anatomic pathology . Arch Pathol Lab Med.
2009; ; 133 : :1841.–1849. 26.
Kussaibi
H
,
Macary
F
,
Kennedy
M
, et al.
HL7 CDA implementation guide for structured anatomic pathology reports methodology and tools . Stud Health Technol Inform.
2010; ; 160 : :289.–293. 27.
Armbrust
M
,
Fox
A
,
Griffith
R
, et al.
Above the clouds : a Berkeley view of cloud computing . EECS Department, University of California; , Berkeley, Tech: . Rep. UCB/EECS-2009–28, 2009. 28.
Boudry
C.
Biologie/médecine 2.0 : état des lieux . Med Sci (Paris).
2012; ; 28 : :653.–658. 29.
Bertheau
P
,
Chabouis
A
,
Fabiani
B
, et al.
Télépathologie par lames virtuelles ou le diagnostic anatomo-pathologique en réseau numérique . Med Sci (Paris).
2012; ; 28 : :983.–985. 30.
Vergier
B
,
Guettier
C.
L’utilisation des lames virtuelles en pédagogie . Med Sci (Paris).
2012; ; 28 : :986.–989. 31.
Janin
A
,
Legrès
L
,
Lebœuf
C
, et al.
Les lames virtuelles en recherche expérimentale et en recherche clinique . Med Sci (Paris).
2012; ; 28 : :990.–992. 32.
Brousset
P
,
Delsol
G.
Émergence d’une spécialité médicale nouvelle : la pathologie . Med Sci (Paris).
2011; ; 27 : :651.–655. |