| |
| Med Sci (Paris). 2013 January; 29(1): 33–36. Published online 2013 January 25. doi: 10.1051/medsci/2013291011.Structure et mode d’injection de l’oncoprotéine CagA d’Helicobacter pylori
Burcu Kaplan-Türköz1 and Laurent Terradot1* 1Institut de biologie et chimie des protéines, université Lyon 1, France ; CNRS, UMR 5086 - bases moléculaires et structurales des systèmes infectieux ; 7, passage du Vercors, 69367Lyon, France MeSH keywords: Animaux, Antigènes bactériens, composition chimique, métabolisme, Protéines bactériennes, Infections à Helicobacter, complications, Helicobacter pylori, Humains, Intégrine alpha5bêta1, Lymphome B de la zone marginale, étiologie, Souris, Souris transgéniques, Structure moléculaire, Protéines oncogènes, Tumeurs de l'estomac |
CagA, la conspiratrice de la carcinogenèse bactérienne
Helicobacter pylori colonise l’estomac de plus de la moitié de la population mondiale et si la grande majorité des personnes infectées ne montrent aucun symptôme, 20 % d’entre elles développent un ulcère gastrique. Plus grave, chez 3 % des patients, la bactérie peut causer des adénocarcinomes ou des lymphomes de type MALT (mucosa-associated lymphoid tissue) et font d’H. pylori la seule bactérie carcinogène de classe 1 (OMS, 1994). La protéine CagA (pour cytotoxin associated gene A) est une protéine d’environ 130 kDa produite par certaines souches d’H. pylori [
1]. Les souches dites cagA-positives sont plus fréquemment associées aux cancers que les souches dites cagA-négatives. Lorsque CagA est surexprimée dans des souris transgéniques, la protéine provoque l’apparition de tumeurs gastriques et de leucémies [
2], démontrant un lien direct entre la protéine bactérienne et l’apparition de cancers. Les souches cagA-positives portent également une batterie de gènes qui codent pour un appareil de sécrétion de type IV (T4SS) [
11]. Cet appareil s’apparente à une seringue moléculaire qui injecte l’oncoprotéine CagA dans les cellules épithéliales. Lors de l’infection, CagA est retrouvée à la pointe de la seringue moléculaire (pilus du T4SS). Cet appendice s’attache aux adhésions focales des cellules et utilise l’intégrine α5β1 comme récepteur. Une fois injectée, CagA reste à la surface interne de la membrane plasmique où elle interagit avec une pléthore de protéines humaines (revue dans [
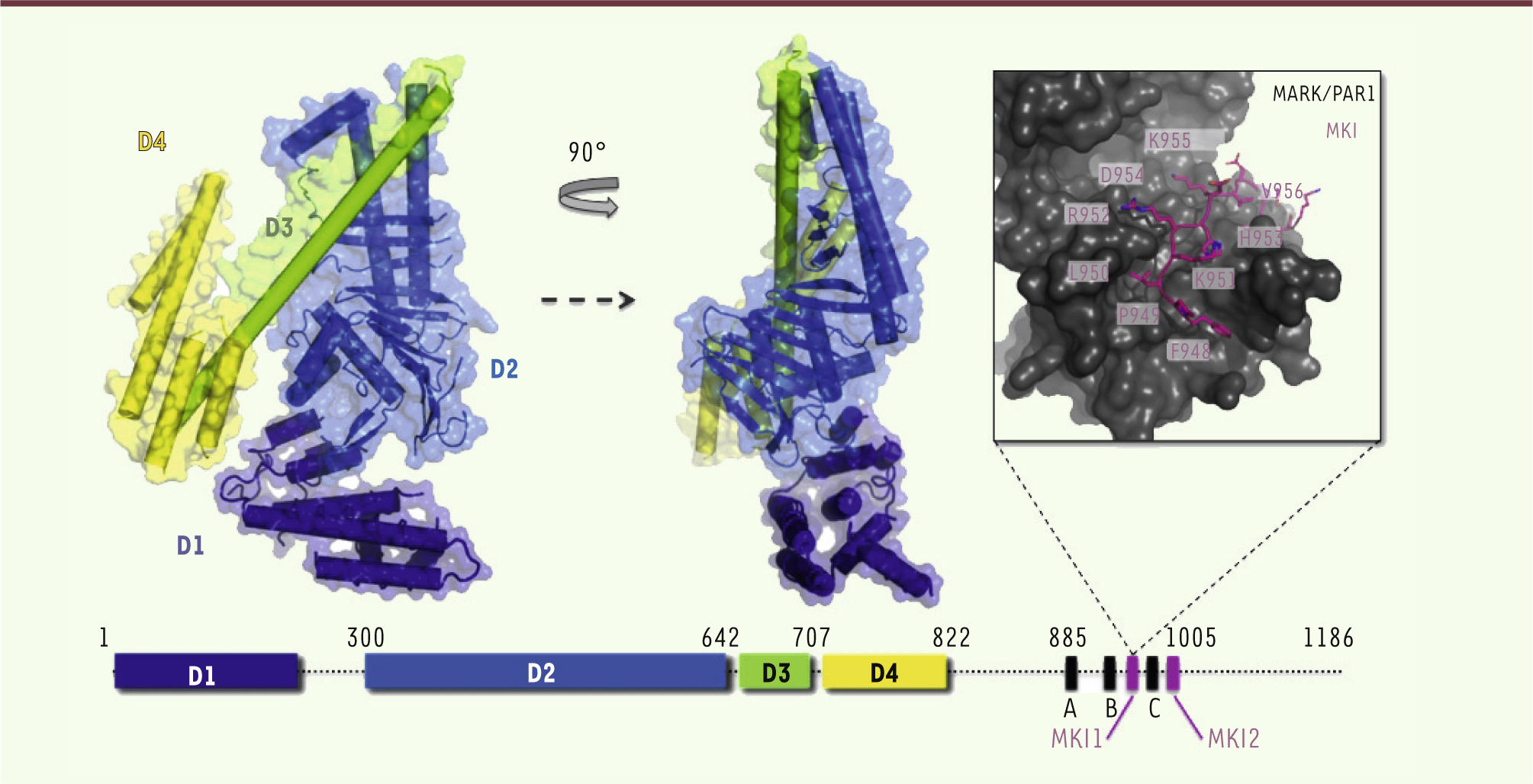
3]). Ainsi, CagA se lie à la kinase MARK1 (MAP/microtubule affinity-regulating kinase 1) par l’intermédiaire de motifs (appelés MKI) situés dans la portion carboxy-terminale (Figure 1). Ces séquences miment le substrat naturel de la kinase et inhibent son activité, ce qui dérègle la polarité des cellules [
4]. La partie carboxy-terminale de CagA peut être phosphorylée (CagA-P) par des kinases de la famille Src sur les tyrosines contenues dans des motifs EPIYA. CagA-P interagit avec les domaines SH2 de nombreuses protéines et en particulier avec ceux de la phosphatase SHP-2. La phosphatase est activée et déphosphoryle les protéines des voies de signalisation, ce qui engendre prolifération et migration cellulaires.
 | Figure 1.
Structure de CagA. Structure cristalline de CagA (résidus 1 à 884) représentée en rubans et tubes (hélices) colorée selon les domaines : D1 (bleu foncé), D2 (bleu clair), D3 (vert) et D4 (jaune). À droite le complexe de la kinase MARK/PAR1 (gris) et du motif MKI de CagA (résidus 948-961) [ 4]. |
|
Vers la détermination de la structure de CagA Malgré l’importance médicale de CagA et l’abondance des études sur ses fonctions, la structure tridimensionnelle de cette protéine restait un mystère. La difficulté principale était d’obtenir un fragment stable de la protéine à partir de son expression recombinante dans Escherichia coli. Nous avons tout d’abord généré une librairie de clones par une approche à haut débit (ESPRIT) en espérant isoler un fragment du domaine carboxy-terminal, dont les fonctions étaient le mieux cernées. Si cette démarche n’a pas permis d’obtenir de structure, elle a apporté des informations déterminantes [
5]. CagA est en fait constituée d’une partie carboxy-terminale non structurée lorsqu’elle est isolée, et d’un large fragment amino-terminal structuré et contenant des éléments de structure secondaire (Figure 1). Ce travail a très récemment abouti à la détermination de la structure du fragment de CagA (résidus 1 à 884 sur 1186 au total) par cristallographie aux rayons X à une résolution de 3,6 Å [
6]. |
CagA, une structure unique La structure de CagA1-884 est modulaire en forme de croissant de lune (Figure 1). Elle contient quatre domaines qui interagissent entre eux assez faiblement et de nombreuses zones restent mal définies. Le premier domaine (D1) est hélical, mais la densité électronique mal définie dans cette région n’a pas permis d’établir sa séquence exacte. Le cœur de la protéine est le domaine D2, formé d’un long feuillet bêta incurvé (single layer beta sheet-SLB) soutenu sur sa face convexe par un petit domaine globulaire (D2’) et par trois hélices formant une épingle. D2 est prolongé par une longue hélice alpha amphipatique (D3) qui sépare D2 du domaine carboxy-terminal D4. Ce dernier est constitué de cinq hélices dont les trois premières forment un bouquet avec l’extrémité de D3. Ainsi, le dernier résidu apparent est Gln822, suggérant que la portion restante est non structurée dans le cristal. L’analyse des résidus exposés et conservés dans les différents variants de CagA disponibles indique plusieurs régions d’intérêt. Ainsi, le bouquet d’hélices (D3 et D4) ressemble au domaine de liaison de l’actine F (F-actin binding domain) retrouvé dans plusieurs protéines du cytosquelette ou des adhésions focales, telles que la kinase des adhésions focales (FAK), la vinculine ou l’a-caténine. Le domaine D2’’ est assez conservé et contient deux résidus responsables de l’attachement de la protéine à la phosphatidylsérine [
7]. Il est probable que cette région soit impliquée dans le maintien de CagA à la surface interne de la membrane plasmique de la cellule hôte. |
Mode d’attachement de CagA sur l’intégrine α5β1 En collaboration avec le groupe du Pr Haas, nous avions précédemment montré que CagA interagissait avec l’intégrine α5β1 [
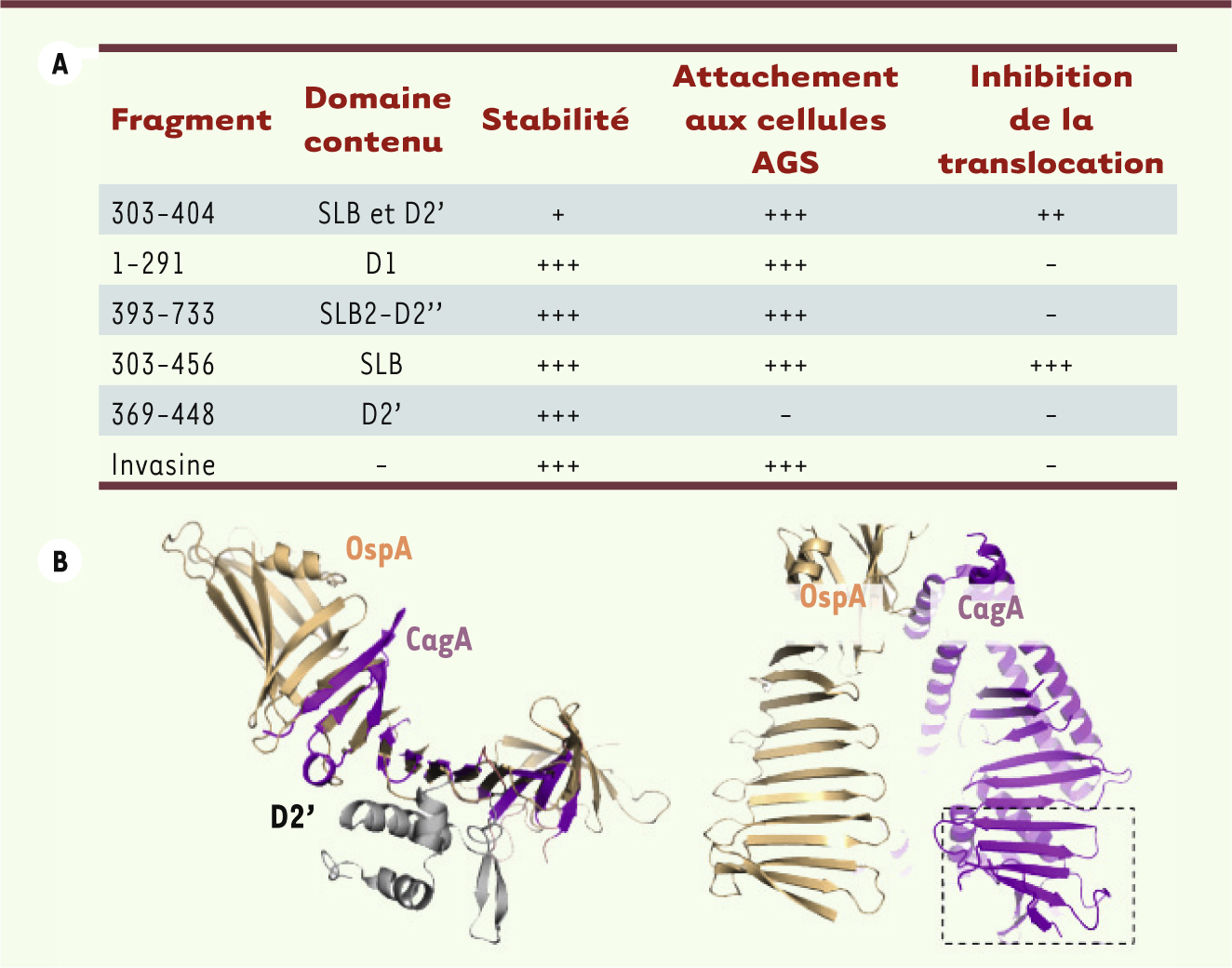
8]. Cependant la zone d’interaction était mal définie et le rôle de l’interaction n’avait pas pu être déterminé. Nous avons donc réalisé une série de mutants de délétion de CagA et testé l’interaction de ceux-ci avec α5β1 par le système double hybride chez la levure. Les résultats indiquent que la région nécessaire à l’interaction contient les acides aminés 303 à 404, ce qui correspond à la moitié proximale de D2 dans la structure de CagA1-884
(Figure 2). Des essais de compétition ont été réalisés à partir de différents fragments de CagA correspondant à différents domaines de CagA (Figure 2). Des cellules AGS (adénocarcinomes gastriques) ont été incubées avec ces fragments purifiés, puis mis en culture en présence d’H. pylori. Nous avons ensuite évalué l’attachement des fragments sur les cellules et détecté l’injection de la protéine CagA par les bactéries. Nous avons observé que les fragments, à l’exception de D2’, s’attachaient sur la cellule. De manière remarquable, les fragments 303-456 et 303-404 étaient capables d’inhiber complètement l’injection de CagA par la bactérie. Ces résultats montrent que l’interaction de CagA avec l’intégrine α5β1 se fait par l’intermédiaire des cinq premiers feuillets β de la SLB. Dans le même essai, la protéine invasine de Listeria n’a pas d’effet sur l’injection de CagA. L’invasine interagit également avec β1, mais par l’intermédiaire de résidus mimant structurellement les motifs RGD des fibronectines, un partenaire naturel des intégrines. Nos résultats indiquent donc que l’interaction de CagA avec α5β1 se fait par un nouveau mécanisme, indépendant du motif RGD. De manière inattendue, la zone d’interaction de CagA ressemble à la protéine OspA (outer surface protein A) produite par la bactérie Borella burgdorferi (Figure 2), l’agent responsable de la maladie de Lyme. Cette bactérie infecte les macrophages et les cellules dendritiques. La protéine OspA joue un rôle prépondérant dans l’infection en interagissant avec l’intégrine αMβ2 [
9]. Les modes d’attachement de CagA sur β1 et de OspA sur β2 pourraient être structuralement similaires.
 | Figure 2.
Analyse fonctionnelle de CagA. Tableau récapitulatif des essais d’inhibition de la translocation de CagA sur des cellules AGS. Comparaison de la structure de CagA (magenta) et de OspA (beige). Le domaine d’interaction de CagA avec l’intégrine α5β1 est indiqué par un rectangle en pointillés. |
|
La structure de CagA représente une étape majeure de la compréhension des mécanismes de l’oncogenèse par H. pylori. L’équipe du Pr Hatakeyama a également réussi à déterminer la structure de CagA et identifier les domaines d’interaction entre les domaines amino-et carboxy-terminaux de la protéine [
10]. La recherche visant à appréhender les détails moléculaires et structuraux du mode d’action de CagA entre donc dans une nouvelle ère et, si nous sommes encore loin de comprendre les détails moléculaires du mode d’action de la toxine, ces structures permettront de nouvelles études plus ciblées. En outre, le fait de pouvoir bloquer la translocation de la protéine est une avancée prometteuse dans la recherche de nouveaux inhibiteurs. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Ce projet a été financé par le programme ATIP-Avenir CNRS-Ligue contre le cancer 2009.
|
1.
Covacci
A
,
Censini
S
,
Bugnoli
M
, et al.
Molecular characterization of the 128-kDa immunodominant antigen of Helicobacter pylori associated with cytotoxicity and duodenal ulcer . Proc Natl Acad Sci USA.
1993; ; 90 : :5791.–5795. 2.
Ohnishi
N
,
Yuasa
H
,
Tanaka
S
, et al.
Transgenic expression of Helicobacter pylori CagA induces gastrointestinal and hematopoietic neoplasms in mouse . Proc Natl Acad Sci USA.
2008; ; 105 : :1003.–1008. 3.
Backert
S
,
Clyne
M
,
Tegtmeyer
N.
Molecular mechanisms of gastric epithelial cell adhesion and injection of CagA by Helicobacter pylori
. Cell Commun Signal.
2011; ; 9 : :28.. 4.
Nesic
D
,
Miller
MC
,
Quinkert
ZT
, et al.
Helicobacter pylori CagA inhibits PAR1-MARK family kinases by mimicking host substrates . Nat Struct Mol Biol.
2010; ; 17 : :130.–132. 5.
Angelini
A
,
Tosi
T
,
Mas
P
, et al.
Expression of Helicobacter pylori CagA domains by library-based construct screening . FEBS J.
2009; ; 276 : :816.–824. 6.
Kaplan-Turkoz
B
,
Jimenez-Soto
LF
,
Dian
C
, et al.
Structural insights into Helicobacter pylori oncoprotein CagA interaction with beta1 integrin . Proc Natl Acad Sci USA.
2012; ; 109 : :14640.–14645. 7.
Murata-Kamiya
N
,
Kikuchi
K
,
Hayashi
T
, et al.
Helicobacter pylori exploits host membrane phosphatidylserine for delivery, localization, and pathophysiological action of the CagA oncoprotein . Cell Host Microbe.
2010; ; 7 : :399.–411. 8.
Jimenez-Soto
LF
,
Kutter
S
,
Sewald
X
, et al.
Helicobacter pylori type IV secretion apparatus exploits beta1 integrin in a novel RGD-independent manner . PLoS Pathog.
2009; ; 5 : :e1000684.. 9.
Pulzova
L
,
Kovac
A
,
Mucha
R
, et al.
OspA-CD40 dyad: ligand-receptor interaction in the translocation of neuroinvasive Borrelia across the blood-brain barrier . Sci Rep.
2011; ; 1 : :86.. 10.
Hayashi
T
,
Senda
M
,
Morohashi
H
, et al.
Tertiary structure-function analysis reveals the pathogenic signaling potentiation mechanism of Helicobacter pylori oncogenic effector CagA . Cell Host Microbe.
2012; ; 12 : :20.–33. 11.
Foulongne
V
,
Michaux-Charachon
S
,
O’Callaghan
D
,
Ramuz
M
. Systèmes de sécrétion des protéines de type IV et virulence bactérienne . Med Sci (Paris).
2002; ; 18 : :439.–447. |