| |
| Med Sci (Paris). 2013 February; 29(2): 219–222. Published online 2013 February 28. doi: 10.1051/medsci/2013292023.Chroniques génomiques Snip et histoire familiale : le match ! Bertrand Jordan1* 1CoReBio PACA, case 901, parc scientifique de Luminy, 13288Marseille Cedex 9, France MeSH keywords: Hybridation génomique comparative, utilisation, Rendement, physiologie, Santé de la famille, Prédisposition génétique à une maladie, Dépistage génétique, méthodes, Étude d'association pangénomique, Humains, Anamnèse, normes, Pedigree, Polymorphisme de nucléotide simple |

Que mes lecteurs en soient prévenus, il va de nouveau s’agir dans cette chronique du caractère prédictif (ou non) des profilages « génome entier » que pratiquent des entreprises comme Navigenics ou 23andMe. Le sujet a déjà été abordé plusieurs fois dans ces pages, mais l’avancée des connaissances modifie le contexte - et la question revêt une grande importance : il s’agit de rien moins que de savoir si les nombreuses études GWAS (genome-wide association studies) pratiquées à grands frais au cours des dernières années permettent, ou non, des prévisions significatives. Rappelons-nous qu’un article dont j’ai fait l’analyse dans une chronique récente [
1 aboutissait, à partir de travaux sur des couples de jumeaux, à un constat assez pessimiste sur le caractère prédictif de notre génome. |
Le sujet est relancé par une récente publication [
2] visant à comparer la pertinence clinique de l’histoire familiale à celle d’un profil détaillé de snip. J’avais évoqué dans une précédente chronique [
3] le cri du cœur d’un expert, disant que les entreprises de génétique DTC (direct to consumer) feraient mieux de jeter les échantillons d’ADN reçus et d’évaluer le risque génétique de leurs clients d’après leur histoire médicale familiale - largement acceptée comme aide au diagnostic, mais dont il faut reconnaître qu’elle n’a pas fait l’objet d’évaluations très précises. Il serait donc utile de comparer objectivement la valeur prédictive de cette information avec celle d’un profil général de snip, ce qui a été abordé dans au moins deux articles récents. Le premier, paru début 2012 [
4] et discuté dans une récente chronique [
5], comparait les prévisions fournies par le test Navigenics avec l’histoire médicale personnelle et familiale de plusieurs milliers de personnes, et concluait à l’absence d’utilité clinique du test. Il était néanmoins plus centré sur l’histoire individuelle de chaque personne que sur la comparaison entre snip et données familiales. Le deuxième, focalisé sur trois cancers fréquents (sein, prostate, côlon) [
6], arrivait lui aussi à des conclusions assez sévères et relevait une forte discordance entre les deux méthodes - mais il était limité à trois affections et portait sur un effectif réduit (44 personnes y compris les témoins). L’article de Bo et al. [2] est donc a priori le bienvenu, puisque son titre (Comparison of family history and SNPs for predicting risk of complex disease) promet une étude générale de l’efficacité de ces deux approches. Premier bémol, la lecture de la rubrique competing interests dûment affichée en première page nous apprend que les quatre auteurs sont tous employés par la firme 23andMe, et que le cofondateur de la revue (Plos Genetics) dans laquelle paraît ce travail est lui-même membre du conseil scientifique de l’entreprise et y détient des stock-options. Cela ne disqualifie pas pour autant les auteurs – d’autant plus que l’un d’eux, Uta Francke, est une généticienne renommée, professeur émérite à Stanford University et ayant à son actif plus de 500 articles - mais on peut néanmoins prévoir que les conclusions de ce papier ne seront pas trop sévères pour 23andMe et consorts. Deuxième déception, il s’agit en fait d’une étude de modélisation utilisant les méthodes de la génétique quantitative pour estimer, en fonction de l’héritabilité et de la fréquence de chaque maladie, le pouvoir prédictif d’une histoire familiale « complète » ou « restreinte » (voir plus bas), et le comparer à celui d’une analyse de snip incorporant toutes les données d’association aujourd’hui connues. Étude de modélisation, donc obligatoirement appareil mathématique et choix de paramètres… difficilement vérifiables sans accès à l’ensemble des calculs. Je me limiterai donc à commenter les résultats présentés et à évaluer leur cohérence avec les données expérimentales disponibles. |
Les limites de l’histoire familiale On l’a déjà dit, l’histoire familiale est largement acceptée comme aide au diagnostic. Elle constitue a priori une source d’information d’accès facile et de coût très bas. Pourtant, comme le font remarquer les auteurs d’un des articles cités par Bo et al. [6], on estime, aux États-Unis, qu’une histoire familiale complète n’est recueillie que dans moins d’un tiers des consultations de cancérologie. À tel point que, pour tenter de remédier à cette situation et toujours aux États-Unis, le Surgeon General, qui est le cadre supérieur du service de santé publique des États-Unis, a mis en place un site intitulé My Family Health Portrait incitant les citoyens à enregistrer leurs données de santé familiales, et visant à leur faciliter la tâche (Figure 1).
 | Figure 1.
Page d’accueil du site My Family Health Portrait (https://familyhistory. hhs.gov/fhh-web/home.action).
|
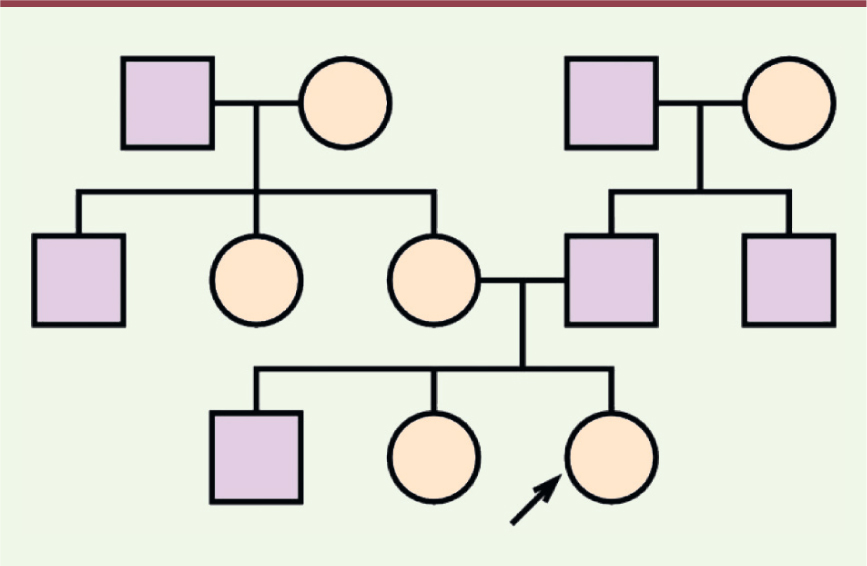
Les auteurs de l’article que je commente ici ont tenu compte de cette difficulté en envisageant deux cas de figure : celle d’une histoire complète pour la famille représentée par la
Figure 2
, et celle d’une histoire limitée à la personne concernée et ses deux parents (trio).
 | Figure 2.
Structure de la famille prise en compte dans la modélisation de C.B. Do et al. [ 2]. Le sujet considéré est la personne indiquée par la fléche (extrait de [ 2], open licence).
|
|
Voyons donc les résultats des simulations effectuées par les auteurs. Je me focaliserai dans cette chronique sur leur expression en termes d’AUC - ces initiales sybillines qui, même développées, ne nous disent pas grand-chose : area under the curve. Quant à la courbe en question, il s’agit de la courbe ROC (receiver operating characteristic), ce qui n’est pas non plus très intuitif ! En fait, j’ai expliqué en détail cette représentation dans une chronique parue début 2012 [5] ; rappelons qu’il s’agit d’une manière de mesurer la valeur d’un test indépendamment des choix faits en matière de sensibilité et de spécificité1,. Un test qui n’est pas meilleur qu’un tirage au sort correspond à une AUC de 0,5, un test parfait (parfaitement spécifique et en même temps parfaitement sensible) à une valeur de 1. On considère en général qu’un test clinique doit avoir une AUC au moins égale à 0,8 pour être utile au diagnostic. Le
Tableau I
montre une partie des résultats présentés dans l’article de Do et al. [2].
Tableau I.
| Maladie |
Risque (vie entière) |
Héritabilité |
Valeurs obtenues pour AUC1
|
% d’héritabilité expliquée |
|
|
|
Histoire complète |
Histoire restreinte |
Profil snip |
Histoire complète |
Histoire restreinte |
Profil snip |
|
| Diabète de type 2 |
0,339 |
0,30 |
0,610 |
0,587 |
0,592 |
18,6 % |
11,7 % |
13,0 % |
| Accident vasculaire cérébral |
0,190 |
0,17 |
0,564 |
0,552 |
0,528 |
9,8 % |
6,3 % |
1,9 % |
| Cancer (prostate) |
0,165 |
0,42 |
0,606 |
0,583 |
0,614 |
10,2 % |
6,3 % |
11,9 % |
| Alzheimer |
0,132 |
0,79 |
0,743 |
0,712 |
0,648 |
27,1 % |
20,6 % |
10,0 % |
| Cancer (sein) |
0,123 |
0,25 |
0,560 |
0,544 |
0,586 |
5,2 % |
2,8 % |
10,5 % |
| Cancer (poumon) |
0,069 |
0,08 |
0,527 |
0,519 |
0,525 |
2,6 % |
1,3 % |
2,3 % |
| Schizo-phrénie |
0,007 |
0,66 |
0,607 |
0,578 |
0,54 |
2,80 % |
1,50 % |
0,40 % |
Extrait du tableau 2 de [ 2] ( open licence). AUC : area under the curve. |
La première chose qui saute aux yeux au vu de ce tableau est que les AUC obtenues sont très modestes, inférieures à 0,6 pour la plupart alors que, rappelons le, une valeur de 0,5 indique un test sans aucune utilité et que 0,8 est généralement considéré comme un minimum. L’histoire familiale, sauf peut-être pour la maladie d’Alzheimer, s’avère bien peu prédictive et guère meilleure lorsqu’elle est détaillée que lorsqu’elle se limite à la personne et à ses deux parents. Quant aux profils de snip, ils sont en général moins performants que l’histoire complète, sauf - mais de manière marginale - pour les cancers de la prostate et du sein. On attendait beaucoup mieux d’après les sites Internet alléchants des entreprises de profilage génétique : la page d’accueil de 23andMe, par exemple, annonce « 23andMe vous dira quel est votre risque génétique et ce que vous pouvez faire »2. Ce tableau indique aussi une valeur pour l’héritabilité de chaque affection, qui semble cohérente avec la littérature, et la fraction de cette héritabilité dont rendent compte respectivement histoire familiale et profil de snip. On voit que cette fraction est au mieux de 27 % (pour la maladie d’Alzheimer et l’histoire familiale complète), et qu’elle tourne généralement autour de 10 %, souvent moins, les snip ne s’avérant à cet égard guère performants (sauf pour le cancer du sein où ils font un peu mieux que les données familiales) : la majeure partie de l’héritabilité reste bien introuvable [
7] ; et d’ailleurs les toutes dernières données de séquençage semblent indiquer que l’espoir de la découvrir dans les variations rares risque fort d’être déçu [
8]. |
En somme, pour ces maladies multigéniques et multifactorielles qui présentent généralement une héritabilité importante (voir la troisième colonne du tableau), les profils de snip tout comme l’histoire familiale s’avèrent assez peu performants. C’est peut-être le résultat le plus intéressant de ce travail (même s’il est fondé sur des modélisations dont on peut toujours discuter les paramètres) que de montrer qu’un prédicteur aussi généralement reconnu que l’histoire familiale est en fait peu informatif. On peut évidemment se demander si les deux indicateurs mesurent les mêmes paramètres, en d’autres termes espérer qu’à eux deux ils donneraient de meilleurs résultats. Les données (expérimentales, cette fois) apportées par l’article de Bloss et al. [4] - d’ailleurs non cité dans celui de Do et al. [2] - n’allaient pas dans ce sens puisque dans leur cas l’ajout des résultats du profilage effectué par Navigenics n’augmentait pas la précision de la prévision [5]. Des résultats aussi peu flatteurs n’encouragent pas - c’est le moins que l’on puisse dire - l’introduction de ces analyses de snip « génome entier » dans la pratique médicale, comme le souligne un récent commentaire particulièrement virulent [
9]. On doit néanmoins rappeler que l’avancée des connaissances devrait normalement améliorer la situation en rendant les profils de snip plus informatifs. Mais il reste vraiment frappant de constater la distance entre le sentiment, très répandu, que notre santé est inscrite dans nos gènes3, et la réalité que montre cet article qui émane pourtant de chauds partisans de ces profilages bien peu prédictifs. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Jordan
B.
Notre génome est-il prédictif ?
Med Sci (Paris).
2012; ; 28 : :785.–788. 2.
Do
CB
,
Hinds
DA
,
Francke
U
,
Eriksson
N.
Comparison of family history and SNPs for predicting risk of complex disease . PLoS Genet.
2012; ; 8 : :e1002973.. 3.
Jordan
B.
Les tests génétiques grand public en « caméra cachée » . Med Sci (Paris).
2011; ; 27 : :103.–106. 4.
Bloss
CS
,
Topol
EJ
,
Schork
NJ.
Association of direct-to-consumer genome-wide disease risk estimates and self-reported disease . Genet Epidemiol.
2012; ; 36 : :66.–70. 5.
Jordan
B.
Les tests génétiques grand public ont-ils une utilité clinique ?
Med Sci (Paris).
2012; ; 28 : :325.–328. 6.
Heald
B
,
Edelman
E
,
Eng
C.
Prospective comparison of family medical history with personal genome screening for risk assessment of common cancers . Eur J Hum Genet.
2012; ; 20 : :547.–551. 7.
Jordan
B.
À la recherche de l’héritabilité perdue….
Med Sci (Paris).
2010; ; 26 : :541.–543. 8.
Kaiser
J.
Human genetics. Genetic influences on disease remain hidden . Science.
2012; ; 338 : :1016.–1017. 9.
Frebourg
T.
Direct-to-consumer genetic testing services: what are the medical benefits?
Eur J Hum Genet.
2012; ; 20 : :483.. |