| |
| Med Sci (Paris). 2013 February; 29(2): 223–225. Published online 2013 February 28. doi: 10.1051/medsci/2013292024.Inactivation du chromosome X chez l’humain XACT et XIST, à chacun son chromosome Céline Vallot1 and Claire Rougeulle1* 1UMR 7216 Épigénétique et destin cellulaire, Université Paris Diderot, Sorbonne Paris Cité, CNRS, bâtiment Lamarck, 35, rue Hélène Brion, 75205Paris Cedex 13, France MeSH keywords: Animaux, Chromosomes X humains, génétique, Cellules souches embryonnaires, métabolisme, physiologie, Femelle, Humains, Mâle, Souris, Modèles génétiques, ARN long non codant, ARN non traduit, Inactivation du chromosome X |
XX versus XY : un X de trop Chez les mammifères, la présence de chromosomes sexuels hétéromorphes X et Y induit un déséquilibre génique entre le mâle et la femelle : le chromosome X est présent en double copie chez la femelle mais en unique exemplaire chez le mâle. Pour compenser cette différence, un chromosome X est inactivé chez la femelle, la transcription y est réprimée. Cette régulation différentielle de deux chromosomes homologues est un modèle de mécanisme épigénétique, c’est-à-dire qui influe sur l’expression des gènes sans modifier la séquence génomique. L’inactivation du chromosome X est un processus qui se met en place très tôt au cours du développement embryonnaire. L’essentiel de nos connaissances concernant l’inactivation et sa mise en place lors des étapes précoces du développement vient de l’étude du modèle murin et, en particulier, des cellules souches embryonnaires de souris [
1,
2] (Figure 1). L’ARN non codant Xist est l’acteur principal de ce processus. Son expression induit le début de l’inactivation et sa propagation sur l’un des chromosomes X chez la femelle. Xist est exprimé à partir du futur X inactif, ce qui entraîne la répression transcriptionnelle du chromosome, puis l’acquisition de marques épigénétiques répressives sur la chromatine [
3]. Si cette inactivation est dans l’ensemble conservée chez tous les mammifères euthériens, des différences importantes existent, notamment chez l’humain. Chez l’homme, les stades précoces de l’inactivation du X n’ont été que très peu caractérisés, mais une étude sur des embryons humains surnuméraires issus de fécondation in vitro (FIV) a montré que chacun des deux chromosomes X était recouvert par l’ARN de XIST et pourtant toujours actif [
4] (Figure 1). Ces résultats indiquent que le processus d’inactivation n’est pas strictement dépendant de XIST chez l’humain, contrairement à ce qui a été montré chez la souris, et laissent ainsi supposer l’existence d’acteurs et de mécanismes de régulation de l’inactivation propres à l’homme.
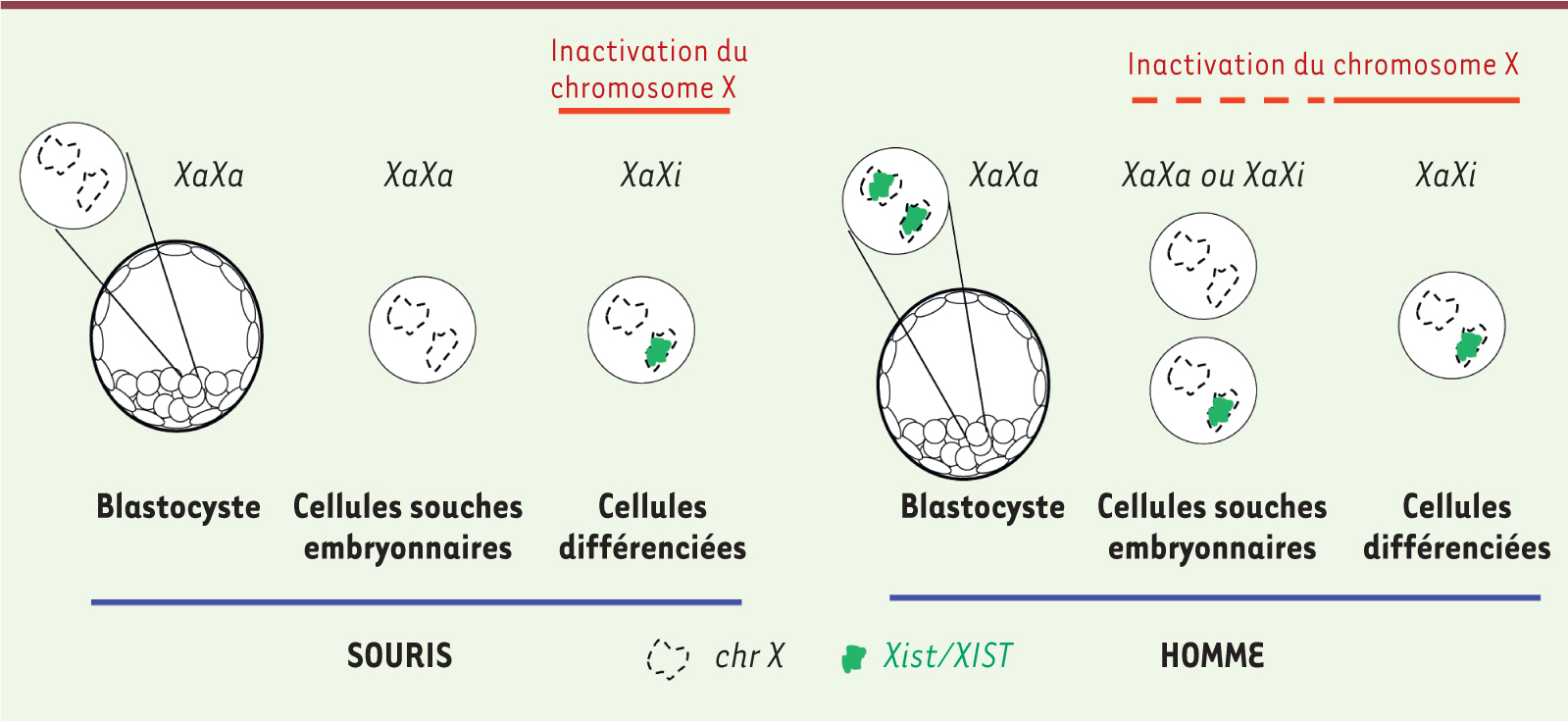
 | Figure 1.
Mise en place de l’inactivation du chromosome X chez la souris et chez l’humain. Les deux chromosomes X sont représentés par des formes en pointillés : le X actif est dénommé Xa et le X inactif Xi. Le processus d’inactivation présente plusieurs différences entre la souris et l’homme : il est déjà partiellement mis en place dans les cellules souches embryonnaires humaines, alors que dans les cellules souches murines les deux X sont actifs. Par ailleurs, XIST est exprimé dans les blastocystes humains, alors qu’à ce stade de développement Xist n’est pas exprimé chez la souris. |
|
Les cellules souches embryonnaires : outil pour comprendre l’inactivation du X chez l’humain Les cellules souches embryonnaires humaines (CSEH) constituent un outil plus accessible que les embryons humains per se pour étudier les stades précoces du développement humain, et offrent notamment une fenêtre sur les étapes initiales de l’inactivation du chromosome X. Les premières études sur les CSEH ont révélé que, si le processus d’inactivation peut débuter dans ces cellules, il n’en est qu’à ses balbutiements et reste très instable. Dans la majeure partie des cellules, un des deux X est inactivé, mais il se réactive fréquemment en culture [
5,
6]. L’inactivation semble donc incomplète et pas totalement verrouillée (Figure 1). Ce phénomène d’instabilité n’est pas retrouvé chez la souris et les CSEH constituent ainsi un modèle unique pour appréhender la complexité de la mise en place de l’inactivation de l’X chez l’humain. |
XACT : un long ARN non codant qui recouvre le X actif
XIST est l’acteur le plus étudié de l’inactivation, mais d’autres ARN non codants interviennent dans l’inactivation du X chez la souris, notamment Tsix et Jpx [
7,
8]. Afin d’identifier de nouveaux ARN potentiellement impliqués dans l’inactivation chez l’humain, nous avons utilisé le séquençage à haut débit de la totalité des ARN issus des CSEH. Nous avons ainsi identifié un nouveau transcrit d’environ 250 kilobases (kb) issu du chromosome X. Ce transcrit se situe en Xq23 dans une vaste région de 1,7Mb dépourvue de gènes, riche en séquences répétées et bordée par les gènes AMOT (angiomotin) et HTR2C (5-hydroxytryptamine receptor 2C). Nous avons étudié le profil d’expression de ce long transcrit grâce à la technique d’ARN-FISH (fluorescent in situ hybridation
1) dans des CSEH. De manière surprenante, nous avons observé que les molécules d’ARN de XACT s’accumulent dans le noyau pour former un amas (Figure 2A), ce nuage recouvrant le chromosome X actif. Nous avons nommé ce transcrit XACT pour X active coating transcript. L’ARN de XACT occupe un volume nucléaire similaire à celui occupé par l’ARN de XIST, environ 4 µm3 des 500 µm3 du noyau. XACT est le second exemple d’ARN non codant (après XIST) capable de recouvrir le chromosome à partir duquel il est transcrit. XACT est cependant le premier ARN qui décore un chromosome actif ; les ARN non codants sont en effet généralement associés à des processus de répression génique [
9]. Contrairement à la majorité des longs ARN non codants répertoriés, XACT n’est pas épissé bien qu’il s’étende sur plus de 250 kb. XACT est par ailleurs polyadénylé et principalement nucléaire. Le promoteur de XACT s’articule autour d’une multitude de sites d’initiation de la transcription regroupés sur une centaine de paires de bases. XACT ne s’exprime que dans des cellules peu ou pas différenciées. Nous n’avons en effet pas détecté de trace de XACT dans des cellules ou tissus adultes. En jouant sur la pluripotence des CSEH par différenciation et reprogrammation successives, nous avons établi que XACT ne s’exprime que dans un contexte cellulaire lié à la pluripotence. L’expression de XACT est ainsi restreinte aux stades auxquels l’inactivation du chromosome X reste malléable.
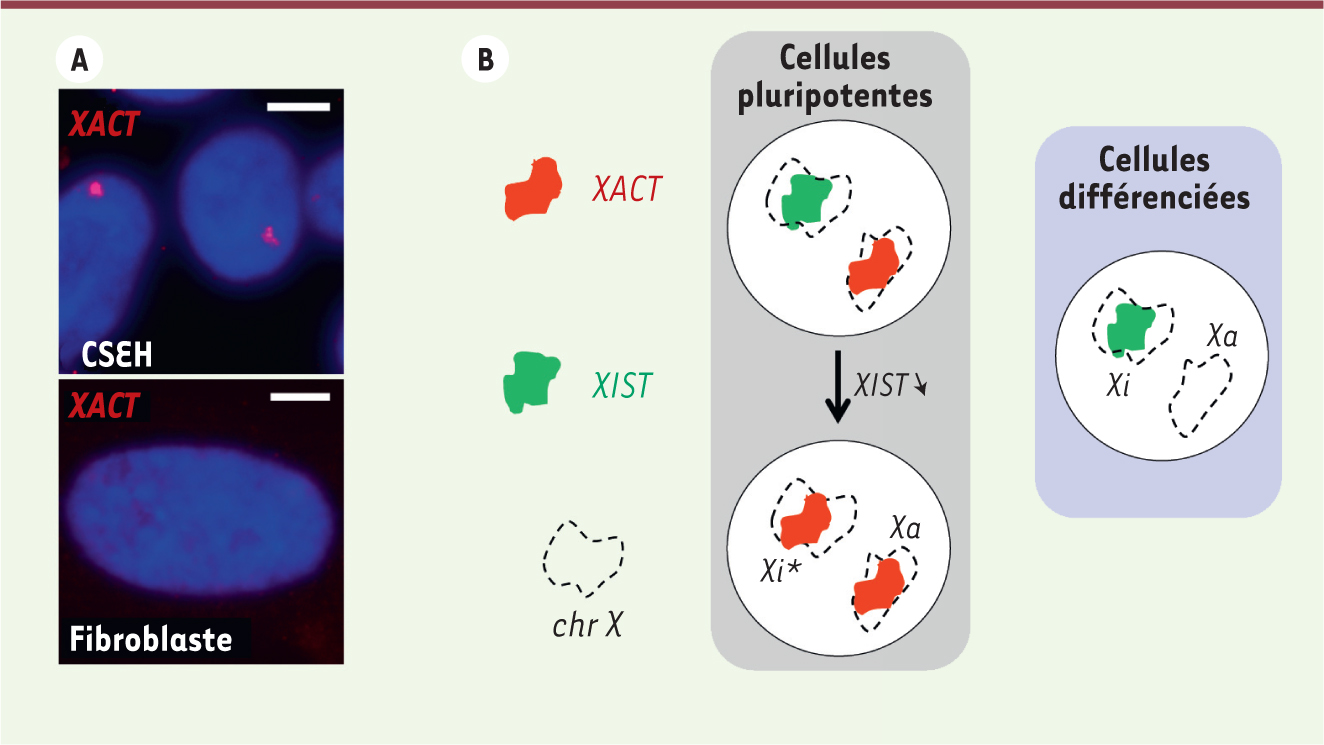
 | Figure 2.
XACT et XIST, la balance de l’inactivation dans les cellules pluripotentes humaines. A. Les images montrent une expérience d’ARN-FISH révélant le profil d’expression de XACT dans des cellules souches embryonnaires humaines (CSEH) et des fibroblastes. B. Le schéma illustre les profils d’expression de XACT et XIST à l’état pluripotent, où l’inactivation est métastable et l’ARN de XIST peut être perdu (*), et à l’état différencié. |
|
XACT et XIST : le Yin et le Yang de l’inactivation chez l’humain ? En détectant simultanément l’ARN de XIST et celui de XACT dans les CSEH, nous avons confirmé que XACT est exprimé en miroir de XIST dans les CSEH avec un X inactif : le X actif est recouvert par XACT alors que le X inactif est décoré par XIST (Figure 2B). Nous avons étudié le profil d’expression de XACT quand le processus d’inactivation est perturbé dans les CSEH. Soumises à des périodes de culture prolongées, les CSEH dont un X inactif est initialement recouvert par XIST ont en effet tendance à perdre l’expression de XIST. Dans ce cas, XACT est alors transcrit à partir des deux chromosomes X et les recouvre (Figure 2B). Reste à déterminer les liens de causalité entre l’apparition de XACT et la perte d’expression de XIST. Chez la souris, si l’organisation de la région génomique autour de XACT est conservée (les gènes Amot et Htr2c sont également voisins et entourent une large région dépourvue de gènes), son promoteur n’est pas conservé. Le promoteur de XACT n’est en effet retrouvé que chez le chimpanzé, suggérant qu’il correspond à une évolution tardive du génome. Par ailleurs, aucun transcrit analogue à XACT n’est produit à partir de la région synténique dans les cellules souches embryonnaires de souris. Ainsi, le profil d’expression de XACT, en miroir de XIST, son lien à la pluripotence, ainsi que son absence de conservation chez la souris, suggèrent que ce très long ARN non codant pourrait jouer un rôle dans les étapes précoces de la mise en place de l’inactivation propres à l’humain. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Chow
J
,
Heard
E.
X inactivation and the complexities of silencing a sex chromosome . Curr Opin Cell Biol.
2009; ; 21 : :359.–366. 2.
Delaroche
L
,
Demailly
P
,
Ancelin
K
,
Patrat
C.
Le modèle de l’inactivation du chromosome X chez la souris . Med Sci (Paris).
2012; ; 28 : :526.–530. 3.
Augui
S
,
Nora
EP
,
Heard
E.
Regulation of X-chromosome inactivation by the X-inactivation centre . Nat Rev Genet.
2011; ; 12 : :429.–442. 4.
Okamoto
I
,
Patrat
C
,
Thepot
D
, et al.
Eutherian mammals use diverse strategies to initiate X-chromosome inactivation during development . Nature.
2011; ; 472 : :370.–374. 5.
Shen
Y
,
Matsuno
Y
,
Fouse
SD
, et al.
X-inactivation in female human embryonic stem cells is in a nonrandom pattern and prone to epigenetic alterations . Proc Natl Acad Sci USA.
2008; ; 105 : :4709.–4714. 6.
Silva
SS
,
Rowntree
RK
,
Mekhoubad
S
, et al.
X-chromosome inactivation and epigenetic fluidity in human embryonic stem cells . Proc Natl Acad Sci USA.
2008; ; 105 : :4820.–4825. 7.
Lee
JT
,
Davidow
LS
,
Warshawsky
D.
Tsix, a gene antisense to Xist at the X-inactivation centre . Nat Genet.
1999; ; 21 : :400.–404. 8.
Tian
D
,
Sun
S
,
Lee
JT.
The long noncoding RNA, Jpx, is a molecular switch for X chromosome inactivation . Cell.
2010; ; 143 : :390.–403. 9.
Khalil
AM
,
Guttman
M
,
Huarte
M
, et al.
Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression . Proc Natl Acad Sci USA.
2009; ; 106 : :11667.–11672. |