| |
| Med Sci (Paris). 2013 June; 29(6-7): 647–655. Published online 2013 July 12. doi: 10.1051/medsci/2013296020.Étude de la résistance de Plasmodium falciparum aux antipaludiques au sein du réseau international des Instituts Pasteur (RIIP-Palu) Didier Ménard,1* Frédéric Ariey,2 and Odile Mercereau-Puijalon2 1Institut Pasteur du Cambodge, unité d’épidémiologie moléculaire du paludisme, 5, boulevard Monivong – BP 983, 101 Phnom Penh Phnom Penh, Cambodge 2Institut Pasteur, unité d’immunologie moléculaire des parasites, Centre national de la recherche scientifique, unité de recherche associée 2581, 25-28, rue du Docteur Roux, 75015Paris, France |

Le paludisme est une maladie provoquée par des hématozoaires appartenant au genre Plasmodium . Deux espèces parmi les cinq qui infectent l’homme, Plasmodium falciparum et Plasmodium vivax, ont un impact majeur en santé publique. P. falciparum provoque une infection aiguë, potentiellement mortelle chez les sujets peu ou non immuns. P. vivax, longtemps considéré comme provoquant des infections bénignes, est maintenant reconnu comme un pathogène susceptible de provoquer des complications graves, responsables de décès [
1]. En cas d’infection palustre, un antipaludique efficace doit être administré le plus rapidement possible pour éviter les complications graves. Le traitement précoce des patients a comme conséquence supplémentaire de réduire la probabilité de transmission du parasite au moustique. En l’absence de vaccin, les antipaludiques jouent un rôle central dans la lutte contre le paludisme. Malheureusement, comme c’est le cas pour de nombreuses infections microbiennes, la pression médicamenteuse exercée au cours des dernières décennies a sélectionné des parasites résistants. L’apparition et la diffusion de parasites résistants aux antipaludiques ont eu des conséquences désastreuses en santé publique dans la seconde moitié du xx
e siècle. Elles ont alourdi le coût économique du paludisme et ont mis à mal les progrès obtenus lors de la campagne mondiale d’éradication lancée dans les années 1950 [
2]. Ainsi, à partir des années 1990, l’incidence du paludisme a augmenté, de même que la mortalité palustre [
3]. Depuis une dizaine d’années, la mobilisation de la communauté internationale, la prise de conscience par les responsables politiques, l’engagement de fondations privées et d’initiatives internationales ont permis de mobiliser des moyens et de faire à nouveau reculer le paludisme [
4]. Cependant, malgré les progrès considérables accomplis, plus de trois milliards d’individus restent exposés au risque d’infection palustre. Ces parasites provoquent encore plus de 210 millions de cas par an et sont responsables d’une mortalité importante, évaluée entre 655 000 et 1,2 millions de décès en 2010 [
5]. La plupart de ces décès surviennent en Afrique chez les enfants de moins de cinq ans, bien que des estimations récentes indiquent que près d’un demi-million de décès interviendraient chez des sujets plus âgés [
6]. |
Nécessité de surveiller la résistance aux antipaludiques Pendant des siècles, le paludisme et les fièvres intermittentes ont été traités avec des préparations issues de la pharmacopée traditionnelle (infusions de qinghaosu, Artemisia annua ou de quinquina, Cinchona calisaya et C. pubescens ). La production massive de chloroquine, vers 1945, a permis de disposer d’un traitement efficace, peu toxique et très bon marché. Son utilisation pour le traitement des accès ou en chimioprophylaxie a constitué un des deux piliers de la campagne d’éradication du paludisme des années 1950-1970 avec la lutte antivectorielle au moyen de DDT dichloro-diphényltrichloroéthane). Les premiers cas de résistance à la chloroquine ont été rapportés en plusieurs points du monde entre 1957 et 1960 (Colombie et Venezuela, Cambodge et Thaïlande, Papouasie Nouvelle Guinée, Philippines). La résistance s’est peu à peu propagée à toutes les zones d’endémie [
7]. La marche infernale de la sélection médicamenteuse s’est poursuivie après le déploiement des médicaments qui ont remplacé la chloroquine : résistance à l’amodiaquine, aux antifolates, à la méfloquine. La montée des multirésistances a amené l’OMS (Organisation mondiale de la santé) à préconiser, il y a une dizaine d’années, l’utilisation de combinaisons comprenant un dérivé de l’artémisinine (ACT, artemisinin combination therapy ) [
8]. Ces combinaisons sont maintenant recommandées dans la plupart des pays d’endémie, mais leur accès est encore loin d’être général. Aujourd’hui, une des préoccupations majeures est que l’on assiste en Asie du Sud-Est aux premiers signes d’émergence de parasites résistants aux dérivés des artémisinines [
9–
12]. Aucune solution thérapeutique de remplacement n’est actuellement disponible. La situation devient donc critique, l’urgence étant d’enrayer la dissémination des parasites résistants [
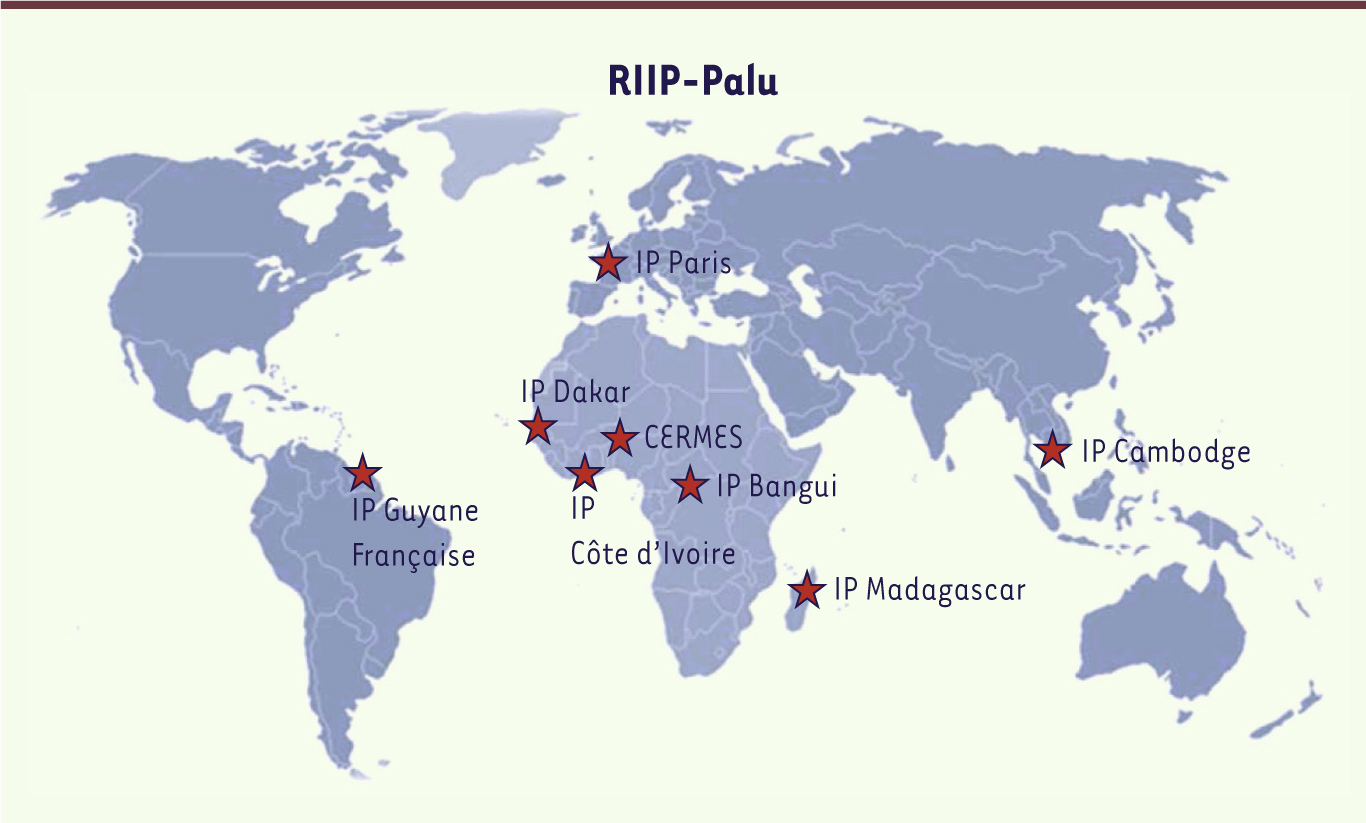
13]. Les équipes de terrain travaillant sur le paludisme, comme celles qui sont implantées au sein du réseau international des Instituts Pasteur (RIIP-Palu), sont en première ligne pour mener des recherches sur les résistances, en quantifier l’importance et proposer des alternatives pour les combattre (Figure 1) . Leurs efforts combinent surveillance et recherche fondamentale. Les travaux de surveillance menés en collaboration avec les autorités sanitaires visent à étudier la distribution des résistances dans différentes zones d’endémie, détecter l’émergence de nouvelles résistances, développer des outils de surveillance pour chiffrer l’étendue des résistances multiples dans une région donnée, et enfin identifier les facteurs épidémiologiques favorisant l’émergence et la propagation des résistances. Les recherches fondamentales portent sur la compréhension des mécanismes moléculaires et cellulaires impliqués dans la résistance.
 | Figure 1.
Les différentes unités du RIIP-Palu travaillant à travers le monde sur le paludisme.
|
|
Un réseau unique pour étudier les résistances Les unités du RIIP-Palu sont considérées comme des laboratoires de référence. Localisées en Afrique, en Asie et en Amérique du Sud, elles travaillent en étroite collaboration avec les programmes nationaux de lutte contre le paludisme, des partenaires internationaux (OMS, Centre national de référence du paludisme pour la France métropolitaine, Institut de recherche pour le développement, Wellcome trust, National institutes of health, US Naval medical research, WorldWide antimalarial resistance network, Fondation Mérieux, Foundation for innovative new diagnostics, etc.), ainsi que des universités françaises et étrangères. S’appuyant sur des plateaux techniques incluant technologies de pointe et expertise scientifique, elles répondent aux exigences de l’assurance qualité. Les unités du RIIP-Palu travaillent de façon concertée pour former des jeunes scientifiques et médecins et développer des recherches avec des méthodologies communes permettant des comparaisons géographiques. De plus, la pérennité des Instituts autorise le RIIP-Palu à jouer un véritable rôle de mémoire à la fois par le recueil longitudinal de données permettant d’étudier l’évolution temporelle de l’efficacité des antipaludiques, et par la constitution de biothèques exploitables pour étudier les mécanismes et les marqueurs de résistance, ainsi que leur évolution temporelle. Jusqu’en 2000, les activités de recherche des différentes unités étaient essentiellement à visée nationale. Au cours de la dernière décennie, une dimension régionale et internationale a été ajoutée à travers des programmes coordonnés d’étude sur les résistances, en étroite collaboration avec l’Institut Pasteur de Paris (plate-forme de séquençage de la génopole et unité d’immunologie moléculaire des parasites) et plusieurs Instituts du réseau (IP Madagascar, IP Cambodge, IP Guyane, IP Dakar, CERMES [Centre de recherche médicale et sanitaire] et IP Côte d’Ivoire). Cette approche a permis de réaliser un transfert rapide de technologies, de mutualiser les efforts visant à développer de nouveaux outils et à accélérer la formation des personnels scientifiques locaux. Sur le plan scientifique, ce réseau représente une opportunité unique de tester l’efficacité in vitro de molécules en cours de développement et de dresser une carte simple mais pertinente de l’efficacité des antipaludiques dans le monde. Ces travaux ont en particulier mis en évidence des disparités géographiques importantes qui permettent de mieux comprendre les paramètres clés liés à l’émergence et à la diffusion des résistances aux antipaludiques au niveau mondial (Annexe 1 à consulter sur le site de médecine/sciences, www.medecinesciences.org). |
Approches méthodologiques Les unités du RIIP-Palu ont une part active dans les quatre axes majeurs de recherches sur les résistances : (1) efficacité clinique, (2) sensibilité in vitro, (3) caractéristiques moléculaires, et (4) épidémiologie de la résistance. Les outils dont nous disposons à l’heure actuelle pour étudier les résistances sur le terrain sont résumés dans l’ Encadré et un grand nombre sont mis en œuvre dans les unités du RIIP-Palu (Tableau I) . Dans cette revue, nous nous concentrerons sur les analyses de la susceptibilité in vitro et les études moléculaires.
Tableau I.
| Sites d’études |
MadagascarIPM |
CambodgeIPC |
GuyaneIPG |
Côte d’IvoireIPCI |
SénégalIPD |
RCAIPB |
| Méthodologie d’étude |
Collaboration avec le PNLP – réseau d’étude de la résistance (RER) dans plusieurs sites sentinelles |
Collaboration avec le PNLP – réseau de sites sentinelles |
Centre national de référence |
Collaboration avec le PNLP |
Suivis de cohorte et études épidémiologiques |
Collaboration avec le PNLP – centres de santé, Bangui |
|
|
P. falciparum
|
Suivi d’efficacité thérapeutique |
Collaboration avec le PNLP – protocole OMS |
Collaboration avec le PNLP, l’OMS et les équipes de recherche – protocole OMS et autres protocoles de recherche |
Collaboration avec les hôpitaux et InVS |
Collaboration avec le PNLP – protocole OMS |
Suivis de cohortes,études transversales |
Collaboration avec la PNLP – protocole OMS |
| Étude de la susceptibilité ex vivo / in vitro
|
Techniques isotopique et fluorimétrique |
Techniques isotopique et microscopique (RSA) |
Technique isotopique |
– |
Technique isotopique |
Technique isotopique |
| Adaptation d’isolats en culture |
Non |
Oui |
Oui, clonage |
Non |
Non |
Non |
| Marqueurs moléculaires |
PCR/RFLPPCR/séquencagePCR/HRM Pfcrt, Pfmdr-1, Pfdhfr/dhps, Pfnhe qPCR : nombre de copies du gène Pfmdr-1
|
PCR/séquencage Pfcrt, Pfmdr-1, Pfdhfr/Pfdhps, Pfcytb, Pfnhe, PfATPase6 qPCR : nombre de copies du gène Pfmdr-1 PCR-LDRFlexiChip Séquençage Illumina ( Whole genome sequencing ) |
PCR/séquençage Pfcrt, Pfmdr-1,
Pfdhfr/dhps, Pfcytb, PfATPase6, Pfcg2 Microsatellites qPCR : nombre de copie du gène Pfmdr-1 RT-PCR Pfmdr1
|
PCR/séquencage Pfcrt, Pfmdr-1,
Pfdhfr/dhps,
PfATPase6
|
PCR/séquencage Pfcrt, Pfmdr-1,
Pfdhfr/dhps, Pfcytb, PfATPase6
|
PCR/RFLP
Pfcrt, Pfmdr-1,
Pfdhfr/dhps
|
|
|
P. vivax
|
Suivi d’efficacité thérapeutique |
Collaboration avec la PNLP – réseau d’étude de la résistance (RER) |
Collaboration avec le PNLP, l’OMS et les équipes de recherche – protocole OMS et autres protocoles de recherche |
Collaboration avec les hôpitaux et InVS |
Non |
Non |
Non |
| Étude de la susceptibilité ex vivo
|
Non |
Technique microscopique et ELISA |
Technique microscopique |
Non |
Non |
Non |
| Adaptation d’isolats en culture |
Non |
Non |
Non |
Non |
Non |
Non |
| Marqueurs moléculaires |
PCR/séquencage Pvcrt, Pvmdr-1, Pvdhfr/dhps qPCR (nombre de copies du gène Pvmdr-1 ) |
PCR/séquencage Pvcrt, Pvmdr-1, Pvdhfr/dhps qPCR (nombre de copies du gène Pvmdr-1 ) |
En cours |
Non |
Non |
Non |
Approches méthodologiques et outils déployés dans les unités du RIIP-Palu. RCA : République centrafricaine. |
Études in vitro
Une des compétences mondialement reconnues du RIIP-Palu est le phénotypage des profils de sensibilité ex vivo/in vitro aux antipaludiques. Grâce à un financement du Prix Louis D. de l’Académie des sciences relayé par le programme PAL+ (ministère français de la Recherche), puis le Fonds de solidarité prioritaire (FSP)-résistances (ministère français des Affaires étrangères), des études coordonnées et multicentriques évaluant la sensibilité in vitro de P. falciparum à un panel d’antipaludiques ont été conduites. Standardisation des protocoles et des réactifs, formation aux bonnes pratiques de laboratoire, ateliers de mise à niveau et stages dans les différents centres de référence comme le centre national de référence du paludisme pour la France métropolitaine ( www.cnrpalu-france.org) ont permis de développer un savoir-faire unique. De très nombreuses études ont été menées dans les différents instituts, permettant d’analyser la sensibilité à un panel de molécules pour des milliers d’isolats récoltés chez des patients lors de l’admission ou lors d’une rechute (Annexe 1 à consulter sur le site de médecine/sciences, www.medecinesciences.org). Du fait de la présence d’équipes sur place et de l’utilisation de protocoles standardisés, des études plus originales ont pu être menées sur la dynamique temporelle d’évolution des résistances in vitro et, en particulier, sur les conséquences des changements de traitement de première intention, en Guyane française [
14], au Cambodge [
15] ou à Madagascar [
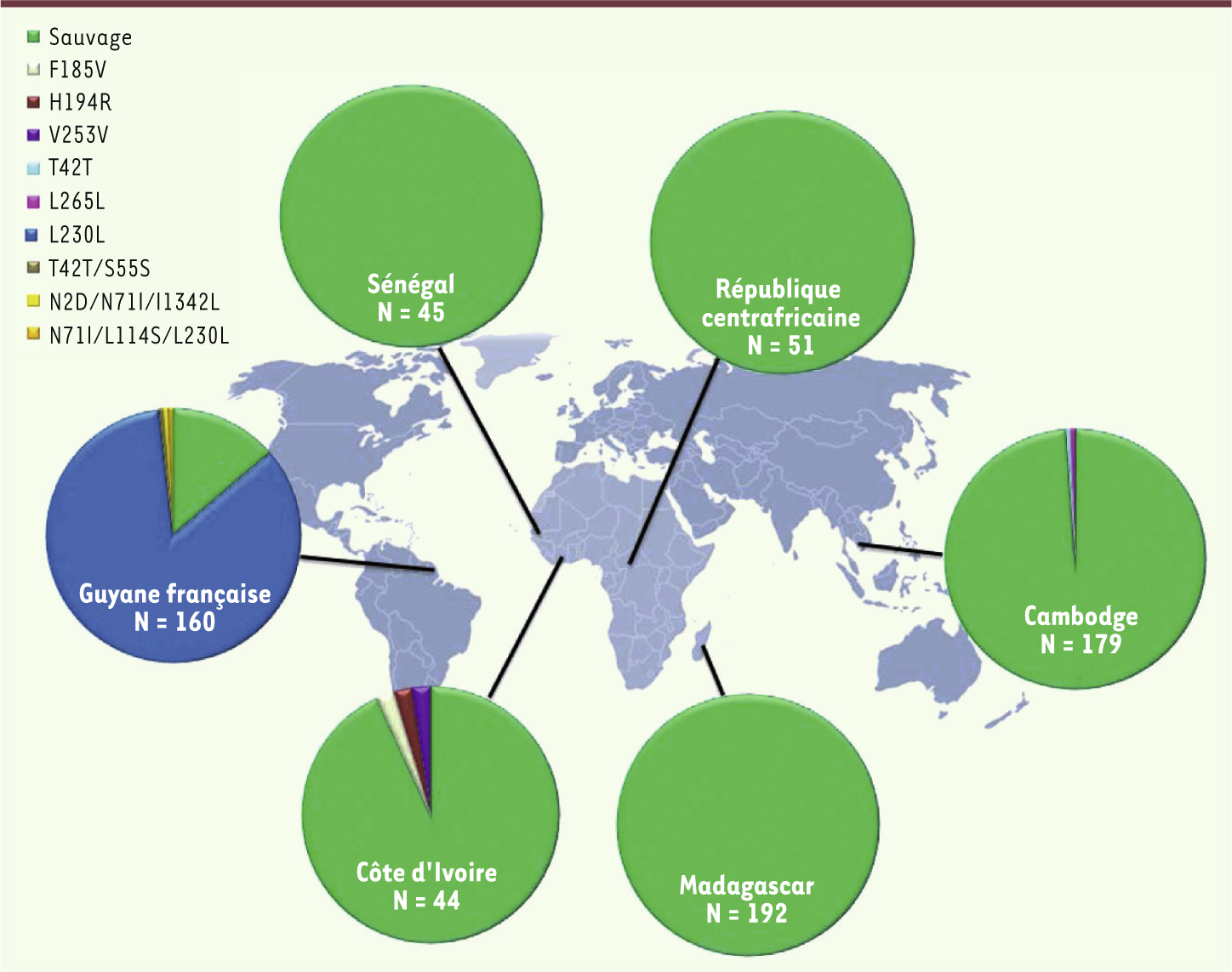
16]. Depuis quelques années, un programme innovant visant à étudier la résistance de P. vivax est conduit au sein du RIIP-Palu. Il permet de partager les efforts de recherche pour la mise au point de tests visant à étudier sa sensibilité in vitro . Marqueurs moléculaires et spécificités géographiques En parallèle des études in vitro, des travaux ont été conduits qui portent sur l’étude des marqueurs moléculaires impliqués dans la résistance des parasites à divers antipaludiques (Tableau I) . Outre la simple recherche des mutations/SNP ( single nucléotide polymorphism ) à l’aide de PCR ( polymerase chain reaction ) spécifiques, le projet ResMalChip (commission européenne) a permis de développer le séquençage systématique des allèles circulant dans les diverses zones d’endémie. Le but de ce projet était de définir, à partir d’isolats de terrain, le polymorphisme des gènes impliqués dans la résistance, et de rechercher la présence éventuelle de nouvelles mutations dans les gènes cibles. Un équipement identique (en particulier pour la PCR) a été mis en place dans les différents sites et une méthodologie d’étude commune a été développée. La stratégie utilisée, « apprendre en réalisant », a permis de développer plus d’une dizaine de protocoles et d’effectuer une étude coordonnée avec des procédures standardisées. En pratique, cette stratégie a été appliquée pour documenter l’étendue du polymorphisme des gènes impliqués dans la résistance aux antifolates, à la chloroquine, à la méfloquine, à l’atovaquone et de gènes candidats pour la résistance aux dérivés de l’artémisinine (Annexe 1 à consulter sur le site de médecine/sciences, www.medecinesciences.org). Plusieurs études multicentriques ont été menées : l’étude du polymorphisme du gène codant pour le cytochrome b ( Pfcytb ) (Figure 2) [
17], l’étude de la sensibilité in vitro d’isolats de P. falciparum aux dérivés de l’artémisinine [
18–
21,
37,
38].
 | Figure 2.
Étude du polymorphisme du gène cytochrome b (Pfcytb) de P. falciparum. L’étude a été effectuée à partir de 671 isolats provenant d’Afrique, d’Asie et d’Amérique du Sud [ 17]. |
L’accélération et l’accroissement des moyens consacrés à la lutte créent une dynamique de changement des conditions épidémiologiques. Les pressions médicamenteuses s’exercent sur les survivants des pressions précédentes et s’accompagnent de flux de gènes qui peuvent être différents dans les divers contextes épidémiologiques où des mutations différentes ont atteint la fixation. Jusqu’à très récemment, on concentrait les analyses sur quelques locus ou SNP, négligeant les interactions possibles entre locus. Il se trouve que nos données indiquent que certains marqueurs pertinents pour prédire la résistance aux antipaludiques dans certaines régions (comme l’Afrique par exemple) doivent être réévalués pour des contextes particuliers comme Madagascar [
22] ou la Guyane française [
23]. Ces données indiquent que l’évolution des populations parasitaires diffère dans différents contextes épidémiologiques et géographiques. |
Les outils disponibles pour l’étude de la sensibilité aux antipaludiques Les tests d’efficacité thérapeutiques Méthodes de référence standardisées par l’OMS [
36], ces tests permettent d’évaluer l’efficacité clinique des schémas thérapeutiques recommandés pour traiter les accès palustres non compliqués. L’efficacité du principe actif est déduite de la disparition de la parasitémie et de l’amélioration du tableau clinique du patient. Le suivi biologique et parasitologique, pendant 28 ou 42 jours, des patients traités permet de distinguer quatre types de réponse au traitement : (1) l’échec thérapeutique précoce (ETP), (2) l’échec parasitologique tardif (EPT), (3) l’échec clinique tardif (ECT), et (4) une réponse clinique et parasitologique adéquate (RCPA) qui caractérise un parasite sensible. Ces études cliniques sont généralement coordonnées avec des tests in vitro et moléculaires. Les tests ex vivo et in vitro
Ils consistent à mettre en culture, soit directement (test ex vivo ), soit après adaptation en culture continue (test in vitro ), un isolat sauvage de P. falciparum en présence d’une concentration croissante d’un antipaludique [37]. La mesure de la croissance des parasites (microscopique, isotopique, ELISA ou fluorimétrique) en fonction de la concentration d’un antipaludique permet de définir son niveau de sensibilité. Le résultat est objectivé par la concentration inhibitrice 50 % (IC50), valeur correspondante à la concentration d’antipaludique permettant d’inhiber la croissance de 50 % des parasites (par rapport au témoin sans drogue). Même si cette technique nécessite une logistique et un plateau technique de qualité, elle permet de tester plusieurs antipaludiques en parallèle et de s’affranchir des facteurs liés à l’hôte (immunité, variation individuelle de la concentration sérique en antipaludique). Les marqueurs moléculaires de résistance Il s’agit d’un ensemble de marqueurs impliqués dans les mécanismes moléculaires de résistance. Pertinents et spécifiques pour prédire le niveau de résistance d’une population parasitaire aux antipaludiques, ils ont une place de choix dans la surveillance de l’activité de tel ou tel antipaludique. Certains marqueurs sont associés à un défaut d’accumulation des pharmacophores au niveau de la cible parasitaire, d’autres à une modification de la cible parasitaire. Ils présentent l’énorme avantage de pouvoir être étudiés sur une large échelle (à partir d’échantillons sanguins prélevés au bout du doigt et déposés sur papier filtre) et sont potentiellement automatisables. Il n’en existe que pour un nombre restreint d’antipaludiques. Aucun marqueur n’est actuellement disponible pour évaluer la sensibilité aux dérivés de l’artémisinine. Les principaux marqueurs moléculaires utilisés dans la surveillance de l’efficacité aux antipaludiques dans le RIIP-Palu sont les suivants :
-
P. falciparum chloroquine transporter ( Pfcrt ). Ce gène situé sur le chromosome 7 code pour un transporteur membranaire de la vacuole digestive. La mutation sur le codon 76 (K→T), associée à sept autres points de mutation [38,
39], permet au parasite de limiter l’accumulation de chloroquine dans sa vacuole digestive, où elle exerce son action inhibitrice [
40]. Pfcrt est également impliqué dans la baisse de sensibilité du parasite à l’amodiaquine et à la quinine [
41,
42]. Dans les zones où les allèles de résistance ne sont pas fixés, on observe une augmentation de la fréquence de l’allèle sauvage après abandon de la chloroquine [
43,
44]. L’analyse de ce locus renseigne sur la pression médicamenteuse exercée au sein des populations.
-
P. falciparum multi-drug resistance 1 ( Pfmdr-1 ). Situé sur le chromosome 5, ce gène code pour un transporteur de type ABC ( ATP binding cassette ). La protéine PfMDR-1 est impliquée dans la modulation de la sensibilité à de multiples antipaludiques et, plus particulièrement, dans l’efflux des antipaludiques hydrophobes [
45]. Les mécanismes de résistance sont liés : (1) soit à des phénomènes de duplication, entraînant une augmentation de l’expression de la protéine [
46] et la résistance aux aryl-amino-alcool (comme la méfloquine ou la luméfantrine) et une baisse de sensibilité aux dérivés de l’artémisinine (mais sans lien statistiquement établi avec l’efficacité clinique des ACT [
47–
49]) ; (2) soit à l’apparition de mutations au niveau des codons 86 (N→Y), 184 (Y→F), 1034 (S→C), 1042 (N→D) et 1246 (D→Y), entraînant une altération de sensibilité des parasites à certains antipaludiques comme les amino-4-quinoléines [45]. Il existe un effet antagoniste entre la sensibilité à la chloroquine et à la méfloquine : la mutation 86Y diminue la sensibilité des parasites à la chloroquine, mais augmente celle de la méfloquine. De même, l’augmentation du nombre de copies du gène (86N) augmente la résistance à la méfloquine et à l’inverse accroît la sensibilité à la chloroquine.
-
P. falciparum dihydrofolate reductase ( Pfdhfr ). Ce gène, situé sur chromosome 4, code pour une enzyme intervenant dans la voie de synthèse des folates [
50]. Elle est la cible des médicaments anti-folates (pyriméthamine, par exemple) qui, en inhibant son activité enzymatique, entraînent le blocage de la synthèse des pyrimidines et la réplication de l’ADN parasitaire [
51]. L’accumulation de plusieurs mutations spécifiques au sein de cette protéine (codons 50N→R, 51C→I, 108S→N et 164I→L) entraînent la résistance clinique des parasites à l’action des anti-folates.
-
P. falciparum dihydropteroate synthase ( Pfdhps ). La dihydroptéroate synthase (DHPS) est une autre enzyme intervenant dans la synthèse des folates (le gène correspondant est situé sur le chromosome 8). Elle est inhibée par les sulfamides. Les mutations se situant au niveau des codons 436 (S→A/F), 437 (K→G), 540 (K→E), 581 (A→G), 613 (A→S/T) confèrent une résistance à la sulfadoxine [
52]. L’analyse groupée des mutations au niveau des gènes Pfdhfr et Pfdhps permet de prévoir l’efficacité clinique de l’association sulfadoxine-pyriméthamine, largement utilisée en Afrique chez la femme enceinte en traitement préventif (TPI, traitement préventif intermittent) ou en association avec l’artésunate en traitement curatif.
-
P. falciparum cytochrome b ( Pfcytb ). Porté par le génome mitochondrial, le gène cytb code pour le complexe cytochrome bc1 intervenant dans le transport des électrons et la synthèse de l’ATP, et cible de l’atovaquone. Les mutations au niveau du codon 268 (Y→N/S/C) diminuent l’efficacité théorique de l’association atovaquone/proguanil actuellement largement utilisée en chimioprophylaxie par les voyageurs [
53]. Aucune mutation n’a pu être mise en évidence sur des isolats sauvages. Les mutants retrouvés ont tous été identifiés chez des patients au décours d’un traitement par l’atovaquone (sélection intra-hôte), et entraînent une diminution importante de la sensibilité du parasite à l’atovaquone.
-
P. falciparum sodium/hydrogen exchanger gene ( Pfnhe-1 ). Le gène Pfnhe-1 (chromosome 13) code pour une protéine impliquée dans les mécanismes d’homéostasie parasitaire comme la régulation du pH, le volume et la composition ionique du cytoplasme [
54]. Il a été en particulier montré que le niveau d’expression de la protéine PfNHE influençait la sensibilité des parasites à la quinine en liaison avec d’autres facteurs, et que certains allèles du microsatellite intragénique ms4760 étaient associés à des diminutions de sensibilité in vitro d’isolats ou de clones de P. falciparum adaptés en culture [
55].
|
|
La sélection et la dissémination Les données de nombreux groupes, dont ceux du RIIP-Palu, ont montré que l’émergence des résistances diffère selon les régions et la molécule utilisée. La résistance à la chloroquine est apparue dans quelques foyers à partir desquels elle a disséminé. Notre étude multicentrique a confirmé qu’un seul allèle de résistance provenant d’Asie du Sud-Est a disséminé à travers l’Afrique, et que cet allèle est distinct de celui qui a disséminé en Amérique du Sud [
24]. L’analyse des variations temporelles des fréquences alléliques dans une zone d’endémie permet de reconstituer les flux géniques et, dans le cas d’une île comme Madagascar, d’identifier la porte d’entrée des résistances [
25,
26] et donc de proposer des mesures de surveillance appropriées. Les facteurs qui contribuent à la sélection puis à la dissémination des résistances au sein des zones d’endémie sont encore incomplètement compris. Des travaux de modélisation réalisés par le RIIP-Palu sur la structure des populations et l’influence de l’endémicité, de la taille du réservoir et de la pression médicamenteuse, ont apporté un éclairage original sur les conditions dans lesquelles la résistance émerge, puis dissémine [
27,
28]. Une étude réalisée dans un village sénégalais, dans lequel nous avons contrôlé de façon très stricte l’utilisation des médicaments, a fourni également des résultats surprenants éclairant d’un jour nouveau cette question [
29]. |
Le développement de nouveaux outils Un des objectifs du RIIP-Palu au cours de ces dernières années a été de développer des méthodes pratiques, robustes et utilisables sur le terrain, qui permettent d’évaluer rapidement la situation épidémiologique de la résistance de P. falciparum aux médicaments antipaludiques. Dans ce cadre, une puce à ADN (FlexiChip), développée par les équipes du Swiss tropical institute (projet européen ResMalChip) [
30], a été utilisée pour analyser la dynamique des résistances à l’échelle du Niger [
31]. Une méthode associant la PCR en temps réel et l’analyse des courbes de fusion (HRM) a également été validée [
32]. Enfin, la détection de la croissance des souches de P. falciparum en présence de différentes concentrations d’antipaludiques par la mesure avec un fluorophore (SYBR Green I) de la quantité d’ADN produite a été évaluée sur des isolats frais en comparaison avec la méthode standard utilisant de l’hypoxanthine radioactive [
33]. |
La mobilisation du RIIP-Palu en 2013 : étude de la résistance aux artémisinines Actuellement, l’OMS recommande l’utilisation d’associations thérapeutiques à base d’artémisinine pour traiter un paludisme simple à P. falciparum . Les dérivés des artémisinines ont une place à part dans l’arsenal thérapeutique, car ils sont les seuls à agir sur toutes les formes de développement du parasite et ont une action remarquablement rapide. Malheureusement, ces médicaments sont maintenant menacés par l’apparition de parasites présentant une sensibilité diminuée aux artémisinines en Asie du Sud-Est [9–12]. Pour faire face à cette menace, un plan a été élaboré par l’OMS début 2011 [
34] qui vise à endiguer et à prévenir la dissémination des souches résistantes aux artémisinines. Il est freiné par notre manque de compréhension des mécanismes impliqués dans la résistance aux artémisinines et par le manque de moyens de détection rapide des parasites résistants. C’est ce manque que visent à combler les recherches actuelles menées par l’lP Cambodge en collaboration avec l’IP Paris. Les données préliminaires obtenues par l’unité d’épidémiologie moléculaire du paludisme de l’IP Cambodge sont encourageantes. Nous avons mis au point un nouveau test in vitro rendant compte de la baisse d’efficacité thérapeutique des artémisinines dans cette région. Ce test simple ( ring-stage survival assay, RSA) explore la survie des parasites exposés à une forte dose de dihydroartémisinine pendant 6 h, et cultivés ensuite en absence de médicament (www.wwarn.org/about-us/news/new-vitro-assay-assess-artemisinin-resistance) [
35]. La capacité des parasites à survivre à cette dose létale montre la même distribution géographique au sein du Cambodge que la résistance clinique et, selon nos données préliminaires, lui est fortement corrélée. Ces résultats indiquent que la résistance est liée à la survie des stades jeunes. Tout se passe comme si un arrêt de croissance est provoqué par l’exposition des parasites à la drogue. Ce type de résistance par « quiescence » est encore très mal compris et difficile à quantifier et à appréhender. Les objectifs majeurs sont maintenant de mieux comprendre les phénomènes cellulaires en jeu dans les souches de terrain et d’identifier des marqueurs moléculaires responsables de ce phénotype in vitro . Ce travail est indispensable pour cartographier l’étendue des résistances et éviter leur dissémination en proposant des schémas thérapeutiques adaptés. |
La dissémination des résistances de P. falciparum et P. vivax à des médicaments bon marché et faciles d’accès est un défi important pour les programmes de lutte contre le paludisme. Les travaux de recherche fondamentale effectués dans les pays développés qui disposent de technologies de pointe sont souvent réalisés sur les souches de référence, récoltées puis entretenues en laboratoire depuis plusieurs décennies. Sur le terrain, les parasites ont depuis été soumis à de nombreuses pressions médicamenteuses et ont évolué pendant plus d’une centaine de générations. Il est donc indispensable que l’efficacité des médicaments en développement soit étudiée sur des souches contemporaines et provenant de différentes régions du monde. Les laboratoires situés en zone d’endémie ont une position stratégique pour participer au développement de nouveaux médicaments ou de nouvelles combinaisons médicamenteuses. De nouveaux problèmes, comme l’émergence de la résistance aux artémisinines, nécessitent une approche multidisciplinaire pour explorer de nouvelles hypothèses sur les mécanismes de résistance et faire avancer les connaissances fondamentales. L’accélération des développements technologiques et la masse de données générées doivent pouvoir bénéficier le plus rapidement possible aux équipes sur le terrain. Ceci nécessite une bonne structuration des réseaux de collaborations locales et internationales. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Références bibliographiques supplémentaires (PDF 266 KB)
|
Nous tenons à rendre hommage et à remercier nos collègues et collaborateurs du RIIP-Palu et de l’Institut Pasteur à Paris qui, depuis le terrain jusqu’au séquençage et phénotypage au laboratoire, ont contribué aux travaux relatés dans cette revue. Nous ne pouvons les citer tous faute de place et les prions de nous en excuser. Nous exprimons nos remerciements à nos collègues des Programmes nationaux de lutte contre le paludisme, ainsi qu’à ceux du Centre national de référence du paludisme pour la France métropolitaine et de l’hôpital Avicenne qui nous ont fait bénéficier de leur expertise, de leurs méthodologies et nous ont procuré des réactifs de standardisation. Nous voulons aussi remercier nos autres partenaires avec qui nous avons exploré divers aspects de la résistance et qui nous ont donné accès aux plates-formes technologiques.
Les recherches ont bénéficié de financements de la fondation Louis D. de l’Académie des sciences, du programme PAL+ (ministère français de la Recherche), du Fonds de solidarité prioritaire (FSP)-résistances du ministère des Affaires étrangères, de la Commission européenne (programme ResMalChip contrat QLK2-CT20021-1503), de la division internationale de l’Institut Pasteur (actions concertées interpasteuriennes), de l’OMS, de la banque Natixis et du Rotary club. Nous remercions la direction internationale du réseau des Instituts Pasteur, les directeurs et les directrices des Instituts du RIIP-Palu pour leur soutien.
|
1.
Anstey
NM
,
Russell
B
,
Yeo
TW
,
Price
RN
. The pathophysiology of vivax malaria . Trends Parasitol.
2009; ; 25 : :220.–227. 2.
Stratton
L
,
O’Neill
MS
,
Kruk
ME
,
Bell
ML
. The persistent problem of malaria: addressing the fundamental causes of a global killer . Soc Sci Med.
2008; ; 67 : :854.–862. 3.
Trape
JF
. The public health impact of chloroquine resistance in Africa . Am J Trop Med Hyg.
2001; ; 64 : :12.–17. 4.
O’Meara
WP
,
Mangeni
JN
,
Steketee
R
,
Greenwood
B
. Changes in the burden of malaria in sub-Saharan Africa . Lancet Infect Dis.
2012; ; 10 : :545.–555. 5.
World Health Organization . World malaria report 2011 . Geneva: : WHO; , 2011. 6.
Murray
CJ
,
Rosenfeld
LC
,
Lim
SS
, et al.
Global malaria mortality between 1980 and 2010: a systematic analysis . Lancet.
2012; ; 379 : :413.–431. 7.
Wongsrichanalai
C
,
Pickard
AL
,
Wernsdorfer
WH
,
Meshnick
SR
. Epidemiology of drug-resistant malaria . Lancet Infect Dis.
2002; ; 2 : :209.–218. 8.
World Health Organization . Guidelines for the treatment of malaria. (2nd ed.) , 2nd ed.
WHO; : 2011. 9.
Amaratunga
C
,
Sreng
S
,
Suon
S
, et al.
Artemisinin-resistant Plasmodium falciparum in Pursat province, western Cambodia: a parasite clearance rate study . Lancet Infect Dis.
2012; ; 12 : :851.–858. 10.
Dondorp
AM
,
Nosten
F
,
Yi
P
, et al.
Artemisinin resistance in Plasmodium falciparum malaria . N Engl J Med.
2009; ; 361 : :455.–467. 11.
Noedl
H
,
Se
Y
,
Schaecher
K
, et al.
Evidence of artemisinin-resistant malaria in western Cambodia . N Engl J Med.
2008; ; 359 : :2619.–2620. 12.
Phyo
AP
,
Nkhoma
S
,
Stepniewska
K
, et al.
Emergence of artemisinin-resistant malaria on the western border of Thailand: a longitudinal study . Lancet.
2012; ; 379 : :1960.–1966. 13.
Enserink
M. If artemisinin drugs fail, what’s plan B?
Science.
2010; ; 328 : :846.. 14.
Legrand
E
,
Volney
B
,
Meynard
JB
, et al.
In vitro monitoring of Plasmodium falciparum drug resistance in French Guiana: a synopsis of continuous assessment from 1994 to 2005 . Antimicrob Agents Chemother.
2008; ; 52 : :288.–298. 15.
Lim
P
,
Wongsrichanalai
C
,
Chim
P
, et al.
Decreased in vitro susceptibility of Plasmodium falciparum isolates to artesunate, mefloquine, chloroquine, and quinine in Cambodia from 2001 to 2007 . Antimicrob Agents Chemother.
2010; ; 54 : :2135.–2142. 16.
Andriantsoanirina
V
,
Ratsimbasoa
A
,
Bouchier
C
, et al.
Plasmodium falciparum drug resistance in Madagascar: facing the spread of unusual pfdhfr and pfmdr-1 haplotypes and the decrease of dihydroartemisinin susceptibility . Antimicrob Agents Chemother.
2009; ; 53 : :4588.–4597. 17.
Ekala
MT
,
Khim
N
,
Legrand
E
, Sequence analysis of Plasmodium falciparum cytochrome b in multiple geographic sites . Malar J.
2007; ; 6 : :164.. 18.
Jambou
R
,
Legrand
E
,
Niang
M
, et al.
Resistance of Plasmodium falciparum field isolates to in-vitro artemether and point mutations of the SERCA-type PfATPase6 . Lancet.
2005; ; 366 : :1960.–1963. 19.
Legrand
E
,
Volney
B
,
Meynard
JB
, et al.
Resistance to dihydroartemisinin . Emerg Infect Dis.
2007; ; 13 : :808.–809. 20.
Ibrahim
ML
,
Khim
N
,
Adam
HH
, et al.
Polymorphism of PfATPase in Niger: detection of three new point mutations . Malar J.
2009; ; 8 : :28.. 21.
Jambou
R
,
Martinelli
A
,
Pinto
J
, et al.
Geographic structuring of the Plasmodium falciparum sarco(endo)plasmic reticulum Ca2+ ATPase (PfSERCA) gene diversity . PLoS One.
2010; ; 5 : :e9424.. 22.
Andriantsoanirina
V
,
Ratsimbasoa
A
,
Bouchier
C
, et al.
Chloroquine clinical failures in P. falciparum malaria are associated with mutant Pfmdr-1, not Pfcrt in Madagascar . PLoS One.
2010; ; 5 : :e13281.. 23.
Legrand
E
,
Yrinesi
J
,
Ekala
MT
, et al.
Discordant temporal evolution of Pfcrt and Pfmdr1 genotypes and Plasmodium falciparum in vitro drug susceptibility to 4-aminoquinolines after drug policy change in French Guiana . Antimicrob Agents Chemother.
2012; ; 56 : :1382.–1389. 24.
Ariey
F
,
Fandeur
T
,
Durand
R
, et al.
Invasion of Africa by a single pfcrt allele of South-East Asian type . Malar J.
2006; ; 5 : :34.. 25.
Andriantsoanirina
V
,
Bouchier
C
,
Tichit
M
, et al.
Origins of the recent emergence of Plasmodium falciparum pyrimethamine resistance alleles in Madagascar . Antimicrob Agents Chemother.
2010; ; 54 : :2323.–2329. 26.
Menard
D
,
Randrianarivo-Solofoniaina
AE
,
Ahmed
BS
, et al.
Drug-resistant malaria parasites introduced into Madagascar from Comoros Islands . Emerg Infect Dis.
2007; ; 13 : :1759.–1762. 27.
Ariey
F
,
Duchemin
JB
,
Robert
V
. Metapopulation concepts applied to falciparum malaria and their impacts on the emergence and spread of chloroquine resistance . Infect Genet Evol.
2003; ; 2 : :185.–192. 28.
Ariey
F
,
Robert
V
. The puzzling links between malaria transmission and drug resistance . Trends Parasitol.
2003; ; 19 : :158.–161. 29.
Noranate
N
,
Durand
R
,
Tall
A
, et al.
Rapid dissemination of Plasmodium falciparum drug resistance despite strictly controlled antimalarial use . PLoS One.
2007; ; 2 : :e139.. 30.
Steenkeste
N
,
Dillies
MA
,
Khim
N
, et al.
FlexiChip package: an universal microarray with a dedicated analysis software for high-thoughput SNPs detection linked to anti-malarial drug resistance . Malar J.
2009; ; 8 : :229.. 31.
Ibrahim
ML
,
Steenkeste
N
,
Khim
N
, et al.
Field-based evidence of fast, global increase of Plasmodium falciparum drug-resistance by DNA-microarrays, PCR/RFLP in Niger . Malar J.
2009; ; 8 : :32.. 32.
Andriantsoanirina
V
,
Lascombes
V
,
Ratsimbasoa
A
, et al.
Rapid detection of point mutations in Plasmodium falciparum genes associated with antimalarial drugs resistance by using high-resolution melting analysis . J Microbiol Methods.
2009; ; 78 : :165.–170. 33.
Rason
MA
,
Randriantsoa
T
,
Andrianantenaina
H
, et al.
Performance and reliability of the SYBR Green I based assay for the routine monitoring of susceptibility of Plasmodium falciparum clinical isolates . Trans R Soc Trop Med Hyg.
2008; ; 102 : :346.–351. 34.
World Health Organization . Global plan for artemisinin resistance containment (GPARC) . Geneva: : WHO; , 2011. 35.
Witkowski
B
,
Khim
N
,
Chim
P
, et al.
Reduced artemisinin susceptibility of Plasmodium falciparum ring stages in western Cambodia . Antimicrob Agents Chemother.
2013; ; 57 : :914.–923. 36.
World Health Organization . Methods for surveillance of antimalarial drug efficacy . Geneva: : WHO Press; , 2009. 37.
Basco
LK.
, Field application of in vitro assays sensitivity of human malaria parasites antimalarial drugs . Geneva: : WHO Press; , 2007
38.
Sidhu
AB
,
Verdier-Pinard
V
,
Fidock
DA
. Chloroquine resistance in Plasmodium falciparum malaria parasites conferred by pfcrt mutations . Science.
2002; ; 298 : :210.–213. 39.
Warhurst
DC
. A molecular marker for chloroquine-resistant falciparum malaria . N Engl J Med.
2001; ; 344 : :299.–302. 40.
Sanchez
CP
,
Stein
WD
,
Lanzer
M
. Is PfCRT a channel or a carrier? Two competing models explaining chloroquine resistance in Plasmodium falciparum
. Trends Parasitol.
2007; ; 23 : :332.–339. 41.
Cooper
RA
,
Lane
KD
,
Deng
B
, et al.
Mutations in transmembrane domains 1, 4 and 9 of the Plasmodium falciparum chloroquine resistance transporter alter susceptibility to chloroquine, quinine and quinidine . Mol Microbiol.
2007; ; 63 : :270.–282. 42.
Tinto
H
,
Guekoun
L
,
Zongo
I
, et al.
Chloroquine-resistance molecular markers (Pfcrt T76 and Pfmdr-1 Y86) and amodiaquine resistance in Burkina Faso . Trop Med Int Health.
2008; ; 13 : :238.–240. 43.
Laufer
MK
,
Thesing
PC
,
Eddington
ND
, et al.
Return of chloroquine antimalarial efficacy in Malawi . N Engl J Med.
2006; ; 355 : :1959.–1966. 44.
Noranate
N
,
Durand
R
,
Tall
A
, et al.
Rapid dissemination of Plasmodium falciparum drug resistance despite strictly controlled antimalarial use . PLoS One.
2007; ; 2 : :e139.. 45.
Duraisingh
MT
,
Cowman
AF
. Contribution of the pfmdr1 gene to antimalarial drug-resistance . Acta Trop.
2005; ; 94 : :181.–190. 46.
Nishiyama
Y
,
Okuda
Y
,
Kim
HS
, et al.
Genetic analysis of mefloquine-resistant mechanism of Plasmodium falciparum . Nucleic Acids Symp Ser (Oxf).
2004; ; 48 : :163.–164. 47.
Price
RN
,
Uhlemann
AC
,
Brockman
A
, et al.
Mefloquine resistance in Plasmodium falciparum and increased pfmdr1 gene copy number . Lancet.
2004; ; 364 : :438.–447. 48.
Pickard
AL
,
Wongsrichanalai
C
,
Purfield
A
, et al.
Resistance to antimalarials in Southeast Asia and genetic polymorphisms in pfmdr1 . Antimicrob Agents Chemother.
2003; ; 47 : :2418.–2423. 49.
Cowman
AF
,
Galatis
D
,
Thompson
JK
. Selection for mefloquine resistance in Plasmodium falciparum is linked to amplification of the pfmdr1 gene and cross-resistance to halofantrine and quinine . Proc Natl Acad Sci USA.
1994; ; 91 : :1143.–1147. 50.
Foote
SJ
,
Cowman
AF
. The mode of action and the mechanism of resistance to antimalarial drugs . Acta Trop.
1994; ; 56 : :157.–171. 51.
Sibley
CH
,
Hyde
JE
,
Sims
PF
, et al.
Pyrimethamine-sulfadoxine resistance in Plasmodium falciparum : what next? . Trends Parasitol.
2001; ; 17 : :582.–588. 52.
Gregson
A
,
Plowe
CV
. Mechanisms of resistance of malaria parasites to antifolates . Pharmacol Rev.
2005; ; 57 : :117.–145. 53.
Srivastava
IK
,
Morrisey
JM
,
Darrouzet
E
, et al.
Resistance mutations reveal the atovaquone-binding domain of cytochrome b in malaria parasites . Mol Microbiol.
1999; ; 33 : :704.–711. 54.
Nkrumah
LJ
,
Riegelhaupt
PM
,
Moura
P
, et al.
Probing the multifactorial basis of Plasmodium falciparum quinine resistance: evidence for a strain-specific contribution of the sodium-proton exchanger PfNHE . Mol Biochem Parasitol.
2009; ; 165 : :122.–131. 55.
Ferdig, MT
,
Cooper
RA
,
Mu
J
, et al.
Dissecting the loci of low-level quinine resistance in malaria parasites . Mol Microbiol 2004. ; 52 : :985.–997. |