Selon Simon Munnery, le génie génétique n’est pas réellement une ingénierie. Les travaux des pionniers ont consisté à jeter des pierres dans l’eau et à déclarer, s’ils parvenaient à traverser, qu’ils avaient créé un pont. La biologie de synthèse s’efforce désormais de créer des ponts comme on en faisait au siècle précédent, pour que la construction soit solide et dure des siècles. En l’occurrence, ce pont est construit avec des briques interchangeables : une multitude de ponts peuvent être bâtis.
Pour prendre une deuxième métaphore, la standardisation du pas de vis nous a permis d’entrer dans l’ère industrielle. La biologie de synthèse s’efforce de faire de même : la standardisation permet l’échange des données. Le Registry of standard biological parts 1 est ainsi une base de données physique et virtuelle stockant des séquences d’ADN standardisées et facilement échangeables, et permet ainsi de faire passer la biologie vers l’ingénierie. La compétition iGEM repose sur cette idée de standardisation : les biobriques créées par les anciennes équipes doivent pouvoir être réutilisées par les nouvelles équipes (Figure 1).
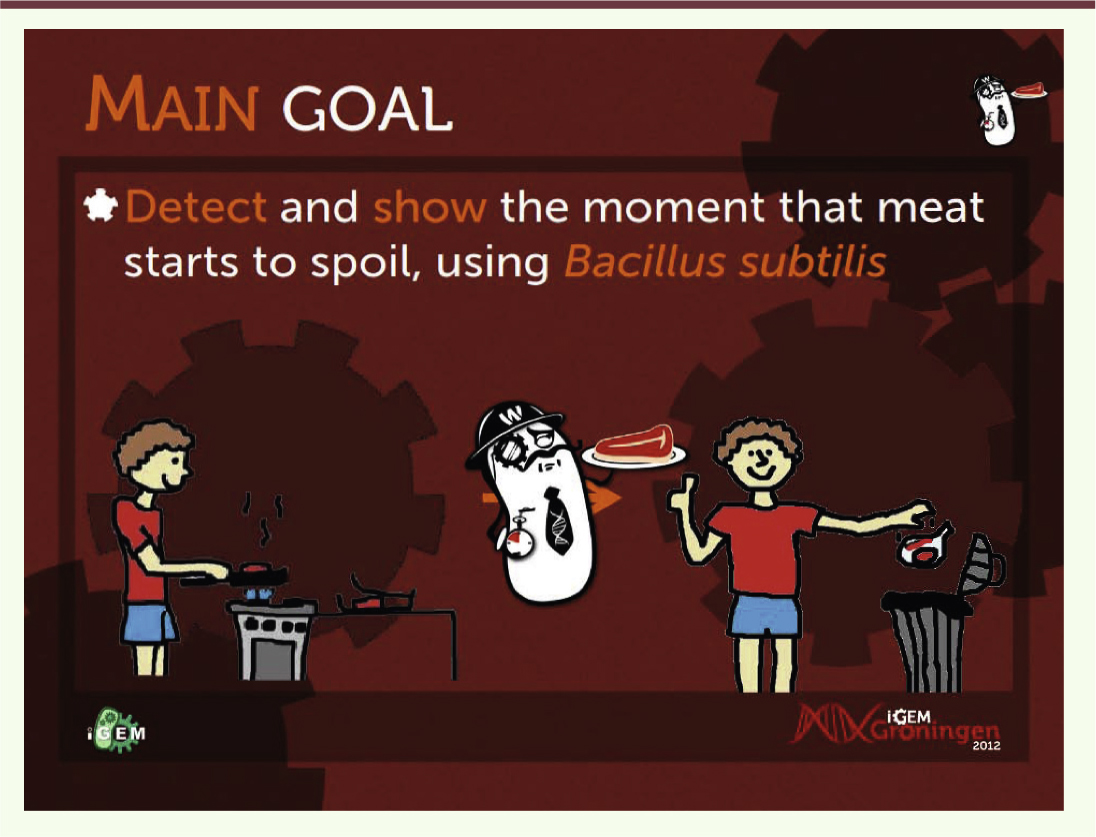
L’équipe qui a remporté l’iGEM 2012 de Groningen (Pays-Bas) est partie du constat que 20 % à 30 % de la viande produite dans le monde étaient jetés parce que la date limite de consommation était dépassée (Figure 2). Son ambition était de créer un biosenseur pour détecter si une viande est avariée et donc réduire les quantités de viande mise au rebut alors qu’elle est encore saine2. L’équipe a d’abord réalisé une puce ADN pour identifier les gènes exprimés quand la viande pourrit, puis a utilisé ces gènes pour réaliser des biosenseurs. Les bactéries sont placées dans un sticker poreux à l’air, mais dont elles ne peuvent pas sortir. Une seconde chambre permet de détruire les bactéries quand elles ne sont plus utiles. Le projet présente la particularité d’allier innovation biologique et considérations pratiques.
Le vainqueur de l’année 2011 était une équipe de Washington qui présentait deux projets. Le premier consistait en la fabrication d’un ensemble d’alcanes, composants du diesel, à partir de sucre et grâce à des bactéries modifiées. Le second projet a permis d’améliorer de cent fois l’activité d’enzymes déjà existantes pour la dégradation du gluten.
L’équipe de Slovénie concourant dans le cadre d’iGEM 2012 a modifié des cellules pour qu’elles produisent des médicaments. Ces cellules sont placées dans des billes d’alginates qui peuvent être introduites dans l’organisme des patients afin de diffuser le médicament quand l’organisme en a besoin. Ce projet comporte une part importante de modélisation pour améliorer la robustesse du dispositif. Il soulève également des interrogations éthiques.
L’équipe d’Évry à laquelle j’appartiens est interdisciplinaire (Figure 3) : elle rassemble des biologistes, des informaticiens, des mathématiciens et un philosophe. Nous venons de différentes écoles et universités de la région parisienne. Nous avons commencé à chercher un projet en décembre 2011.
La biologie de synthèse recourt fréquemment au terme « châssis » qu’elle a emprunté à l’ingénierie : un même châssis de Renault Mégane® permet de réaliser la version décapotable, le grand Scénic® et d’autres véhicules. Il en va de même en biologie de synthèse : tous les acteurs peuvent partir d’une même base et s’échanger les outils utilisables pour l’améliorer. Ce châssis est le plus souvent la bactérie Escherichia coli, la bactérie Bacillus subtilis ou la levure Saccharomyces cerevisiae (Figure 4).
Notre projet visait à faire passer la biologie de synthèse au niveau supérieur, c’est-à-dire à passer des organismes unicellulaires le plus souvent utilisés à des organismes pluricellulaires - en l’occurrence un vertébré, le xénope. Ce dernier présente l’avantage d’être composé d’organes et de cellules différenciées, d’avoir un comportement défini et un génome totalement séquencé.
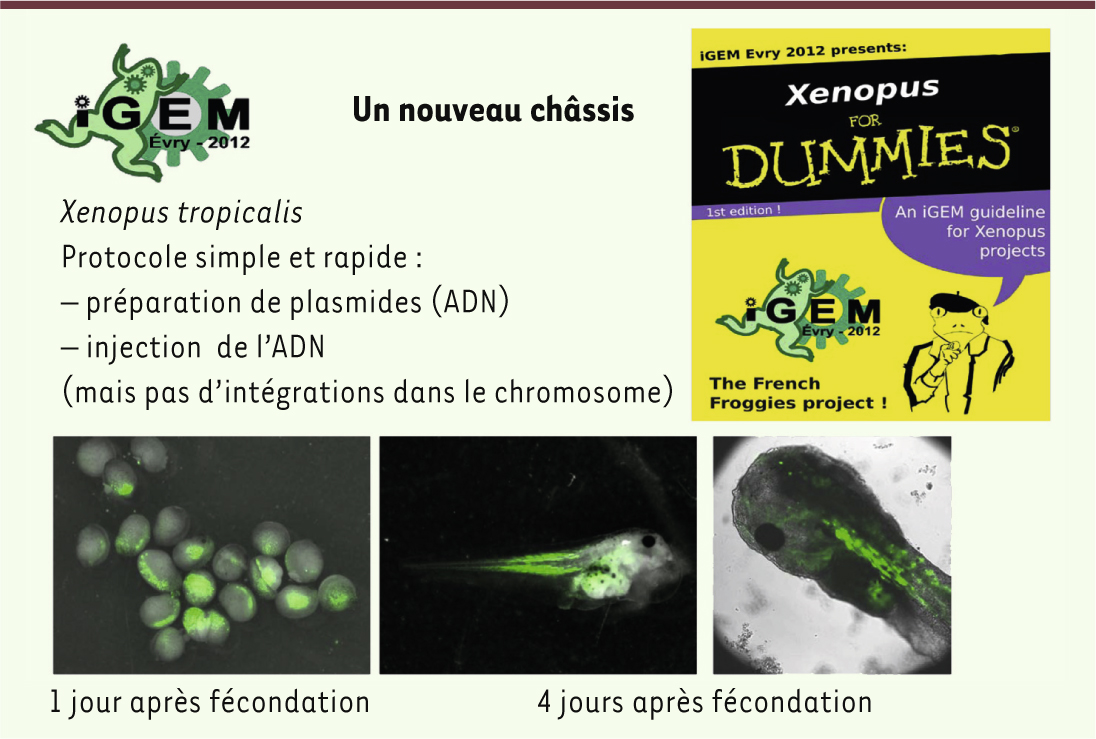
Nous avons d’abord voulu montrer que des étudiants pouvaient facilement injecter de l’ADN dans des œufs de têtard fécondés pour produire des molécules. La preuve de principe a été faite via des morceaux d’ADN codant pour des molécules fluorescentes - préalablement produits dans des bactéries et purifiés - qui ont été injectés directement dans les œufs : quatre jours après l’injection, des molécules fluorescentes vertes étaient produites (Figure 5). Pour des raisons éthiques et dans un premier temps, nous n’avons pas intégré les plasmides au niveau chromosomique. Les plasmides se dégradent au fil du temps, ne se répliquent pas et ne sont pas transmis à la descendance, quand il y en a une. Nous avons rédigé un livret sur l’utilisation du xénope en biologie de synthèse (Figure 5) pour expliquer ces enjeux et pour que d’autres scientifiques puissent profiter des connaissances que nous avons acquises.
Une fois la preuve de principe faite, nous sommes passés à la production d’auxine, une hormone végétale, pour développer un système hormonal de communication intertissulaire orthogonal. Nous avons également monté un écosystème entre différents châssis : nous avons fait ingérer à nos têtards de xénope des bactéries E. coli produisant des molécules fluorescentes rouges ; nous avons montré que les bactéries restaient vivantes après plusieurs jours dans l’intestin du têtard et continuaient de produire ces molécules fluorescentes. Ce résultat pourra servir à l’étude des microsystèmes microbiens de l’intestin.
Nous avons remporté à Amsterdam le prix du meilleur modèle informatique/mathématique (Best model). Les échanges entre scientifiques de différentes disciplines sont en effet essentiels en biologie de synthèse. Nous avons travaillé avec nos mathématiciens et nos informaticiens sur un modèle informatique capable de reproduire nos manipulations en laboratoire et d’en prédire le résultat : nous pouvons ainsi éviter les expériences vouées à l’échec. Notre modèle s’applique à plusieurs échelles : nous commençons par injecter le plasmide pour étudier sa distribution dans l’œuf entier à l’échelle du nanomètre. À l’échelle supérieure, nous produisons l’auxine dans quelques cellules et la détectons dans un autre tissu. Nous passons enfin à la communication à l’échelle de l’organisme, du tissu entier qui produit l’auxine, au transport et enfin à la réception. Ces résultats découlent directement des échanges approfondis entre représentants de différentes disciplines, même si nous nous sommes d’abord heurtés à quelques problèmes de compréhension.
Notre équipe est épaulée par une équipe de recherche dirigée par Nicolas Pollet qui s’efforcera d’aller plus loin, jusqu’à la publication. Nous avons remporté deux prix lors de la finale européenne, celui du Best model et celui du Best human practices advance. Nous avons présenté notre projet lors de la finale mondiale, où il a été plutôt bien reçu.
La participation au concours iGEM nous a donné d’autres compétences en administration et en management ; cela est dû à la recherche de sponsors, à la communication avec les médias et au travail d’équipe. Ces compétences ne sont pas (ou peu) développées à l’université ; on ne s’intéresse pas au bio-art et on aborde peu les questions d’éthique. Nous avons beaucoup gagné à échanger avec des représentants d’autres disciplines ainsi qu’avec des professionnels.
iGEM est une compétition étudiante : c’est une occasion rare pour un jeune de moins de 30 ans de créer un projet complet de biologie et de mener le travail jusqu’à son terme, avec tout le temps nécessaire, les moyens et les ressources appropriés. Nous avons rencontré les autres équipes d’iGEM, mais aussi de grands chercheurs venus du monde entier pour découvrir et évaluer les projets. Enfin, la devise d’iGEM est : « Have fun! ».
L’équipe iGEM Évry 2013 (dont Tristan Cerisy est l’advisor) est constituée et présentera un projet sur l’hémochromatose. Renseignements complémentaires à : tristan.cerisy@gmail.com
|
Catherine Paradeise Il est significatif qu’iGEM soit un projet du MIT : tous les étudiants de cette université sont autonomes, peuvent construire eux-mêmes leurs propres conditions de coopération et sont encouragés à prendre des responsabilités interdisciplinaires et d’organisation. Le concours a le mérite de diffuser largement ces pratiques, dont les Européens en particulier peuvent s’inspirer. |